Method Article
Fluoreszierend markierte Bakterien als Tracer, um neuartige Wege des organischen Kohlenstoffflusses in aquatischen Ökosystemen zu enthüllen
In diesem Artikel
Zusammenfassung
Hier wird ein Protokoll für eine einzellige, epifluoreszenzmikroskopische Technik zur Quantifizierung der Weideraten in aquatischen Raub-Eukaryoten mit hoher Präzision und taxonomischer Auflösung vorgestellt.
Zusammenfassung
Die Aufklärung trophischer Wechselwirkungen, wie Raubund und ihre Auswirkungen, ist eine häufige Aufgabe für viele Forscher in der Ökologie. Das Studium von mikrobiellen Gemeinschaften hat viele Einschränkungen, und die Bestimmung eines Raubtiers, Beute und Raubtierraten ist oft schwierig. Präsentiert wird hier eine optimierte Methode, die auf der Zugabe von fluoreszierend gekennzeichneter Beute als Tracer basiert, die eine zuverlässige Quantifizierung der Weideraten in aquatischen Raub-Eukaryoten und die Schätzung des Nährstofftransfers zu höheren trophischen Werten ermöglicht.
Einleitung
Heterotrophe Prokaryoten sind eine wichtige biologische Komponente in aquatischen Systemen und machen einen signifikanten Anteil der Planktonbiomasse1,2,3aus. Faktoren, die ihren Überfluss, ihre Vielfalt und ihre Aktivität kontrollieren, sind entscheidend für das Verständnis ihrer Rolle beim biogeochemischen Kreislauf (d. h. das Schicksal von organischem Kohlenstoff und anderen Nährstoffen und Energiefluss von Prokaryoten zu höheren trophischen Niveaus). Protozoen Weide ist einer dieser wichtigen Faktoren. Bacterivory von heterotrophen Nanoflagellaten und Ziliaraten erzwingt eine starke Kontrolle von oben nach unten über prokaryotische Fülle, Gemeinschaftsfunktion, Struktur, Vielfalt und sogar zelluläre Morphologie und Wachstumsrate bestimmter Bakteriengruppen4, 5,6. In einigen Systemen dienen Protisten als Hauptursache für die bakterielle Sterblichkeit6,7.
Der Standardansatz zur Bewertung von Protozoen-Bakterien, der seit einiger Zeit verwendet wird, beinhaltet die Verwendung fluoreszierend markierter Bakterien (FLB) als Beuteanaloga und Epifluoreszenzmikroskopie. Zellspezifische Aufnahmeraten können durch Quantifizierung der Anzahl der markierten Beutepartikel in Protistan-Lebensmittel-Vakuolen über einen ausgewählten Zeitverlauf bestimmt werden8. Dieser Ansatz hat mehrere Vorteile. Tracer wird natürlichen Proben mit natürlichen Raubtier- und Beuteansammlungen hinzugefügt. Es gibt minimale Probenmanipulationen vor der Inkubation, minimale Probenänderung durch den hinzugefügten FLB-Tracer und Inkubationszeiten sind kurz, um sicherzustellen, dass unter nahezu in-situ-Bedingungen fundierte Ergebnisse erzielt werden. Alternativ können in Umgebungen mit geringer Anzahl bakterivorer Protisten oder Zooplankton (z. B. Offshore-Meeressysteme) die Verschwindenraten von FLB, die Proben in geringen Mengen (2%-3% Tracer) zugesetzt werden, über die Durchflusszytometrie langfristig nachgewiesen werden (12-24 h) Inkubationsexperimente. Dann werden die FlB-Zahlen am Anfangs- und Endpunkt (die Die Auswirkungen aller Bakterivoren integrieren) durch Strömungszytometrie quantifiziert (Details siehe vorherige Publikation9). Ein solcher Parameter stellt jedoch nur die aggregierten Bakteriumsraten dar, die nicht direkt auf bestimmte Protistan- und Zooplankton-Weidegruppen oder -arten zurückgeführt werden können.
Insgesamt kann die genaue und ökologische Quantifizierung der protistan-arten- oder morphotypspezifischen bakteriellen Sterblichkeitsraten in der aquatischen Umwelt eine Herausforderung darstellen. Einige Protisten sind selektive Weidetiere, und die Größe und Zellform des hinzugefügten FLB-Tracers kann die natürlichen Raten der Beuteaufnahme10,11verzerren. Darüber hinaus sind Protistan Aktivität und Stoffwechsel hochtemperaturempfindlich12; Daher muss die Menge des hinzugefügten FLB-Tracers für jeden einzelnen Probentyp sorgfältig manipuliert werden (nicht nur basierend auf der natürlichen Häufigkeit, Größe und Morphologie von Bakterien und vorherrschenden Arten von Bakterienfressern, sondern auch auf der Temperatur). Die meisten Studien konzentrieren sich auf Bulk Protistan Weideaktivität; die Bakterivory bestimmter Protistan-Arten haben jedoch oft einen viel höheren Informationswert und können vorzuziehen sein. In diesem Fall ist taxonomisches Wissen über die protistischen Arten, die in einer Probe vorhanden sind, und das Verständnis ihres Verhaltens erforderlich. Daher sind erhebliche Mengen an Zeit und Arbeit erforderlich, um solide Ergebnisse über artspezifische Bacterivory-Raten zu erhalten, die einer bestimmten Protistan-Gruppe oder -Art zuzuschreiben sind.
Trotz dieser Schwierigkeiten bleibt dieser Ansatz das derzeit am besten geeignete Instrument zur Bewertung von Protistan-Bacterivory in natürlichen Umgebungen. Hier wird eine umfassende, leicht verständliche Methode zur Verwendung von FLB als Tracer in aquatischen mikrobiellen Ökologiestudien vorgestellt. Alle genannten problematischen Aspekte des Ansatzes werden berücksichtigt und ein verbesserter Workflow beschrieben, mit zwei Experimenten aus kontrastierenden Umgebungen sowie kontrastierenden Zilienarten als Beispiele.
Die erste Fallstudie wurde in einer epiimnetischen Umgebung aus dem mesotrophischen Wasserreservoir von Mov in der Tschechischen Republik durchgeführt, das Weide- und Bakterienmengen zeigt, die mit den meisten Oberflächensüßwasserkörpern vergleichbar sind (vgl.5,7). Die zweite Fallstudie wurde in der hochspezifischen Umgebung in Fallen der aquatischen fleischfressenden Pflanze Utricularia reflexadurchgeführt, die eine extrem hohe Anzahl von beiden grasenden Mixotrophen(Tetrahymena utriculariae) beherbergt. und Bakterienzellen. Es werden Berechnungen der zellspezifischen Weideraten und der bakteriellen Bestandsbestände in beiden Stichprobentypen dargestellt. Anschließend wird eine Reihe ökologischer Interpretationen der Ergebnisse diskutiert und schließlich Beispiele für mögliche Folgestudien vorgeschlagen.
Protokoll
1. Probensammlung
-
Sammlung von Reservoir-Wasser-Probe: die erste Fallstudie (Exp I; niedrigere natürliche in situ Raubtier und Beute Fülle System)
- Sammeln Sie Wasserproben von der gewünschten Stelle in geeigneter Tiefe. Bewahren Sie die Proben in einem temperaturgeregelten Kühler auf, der bei In-situ-Temperatur gefüllt wird (Vermeidung von Temperaturschock; es ist zu beachten, dass die Aufnahmeraten von Protisten temperaturabhängig sind) während des Transports zum Labor.
ANMERKUNG: Unsere Probenahme wurde im meso-eutrophen Canyon-förmigen Reservoir von Mov durchgeführt (Südböhmen, Volumen 34,5 x 106 m3, maximale Tiefe 43 m, mittlere Retentionszeit 100 Tage, dimictic). Die Probenahmestelle befand sich in einer Tiefe von 30 m in der Nähe des Staudamms. Eine gemischte Probe in einer Tiefe von 0,5 m wurde von einem 2-l Friedinger Sampler gesammelt. - Fahren Sie so schnell wie möglich mit Abschnitt 1.2 fort.
- Sammeln Sie Wasserproben von der gewünschten Stelle in geeigneter Tiefe. Bewahren Sie die Proben in einem temperaturgeregelten Kühler auf, der bei In-situ-Temperatur gefüllt wird (Vermeidung von Temperaturschock; es ist zu beachten, dass die Aufnahmeraten von Protisten temperaturabhängig sind) während des Transports zum Labor.
-
Sammlung von Flüssigkeit aus den Fallen fleischfressender Utricularia-Reflexa-Pflanzen: die zweite Fallstudie (Exp II; ein System mit hoher Raubtier- und Beutemenge, extrem geringes Probenvolumen)
- Schütteln Sie die Pflanzen vorsichtig unter Wasser, entfernen Sie sie aus dem Anbaubehälter und legen Sie sie auf saugfähiges Material, um überschüssiges Wasser aufzunehmen. Je nach Robustheit der Pflanzen und Größe der Fallen wählen Sie 8-10 Pflanzenpersonen.
- Teilen Sie jeden Trieb in etwa gleiche Teile auf, indem Sie Blattknoten zählen, die Fangorgane tragen. Jedes Triebsegment dient dazu, gemischte Proben zu sammeln, die junge, mittlere und alte Fallen darstellen.
- Befestigen Sie eine dünne Glaskapillare und eine Eppendorf-Durchstechflasche zur Probenentnahme an einer Peristaltikpumpe. Setzen Sie die Kapillarspitze in die Fallöffnung ein. Mit der Vakuumpumpe saugen Sie die gesamte Flüssigkeit aus jeder Falle heraus, bis für jede Fallalterskategorie 900 x 100 l Fallenflüssigkeit gesammelt werden.
- Verwenden Sie für Protistan-Weideexperimente dreifache Subsamples von 200 l der gepoolten Fallenflüssigkeit. Verarbeiten Sie diese sofort, wie in Abschnitt 3 beschrieben. Bewahren Sie die verbleibenden 300 l der Probe für alle anderen Analysen der in der Flüssigkeit lebenden mikrobiellen Komponenten auf, wie unten beschrieben (Abschnitt 2).
- Fahren Sie sofort mit Abschnitt 2 fort.
2. Fixierung der gesammelten Proben
- Exp I und II: Fix wasser/trap fluid sub-proben für die bakterielle Aufzählung (Abschnitt 4; ca. 20 ml bzw.) mit Formaldehyd für mindestens 1 h, um eine 2% Endvolumen-Volumen-Volumenkonzentration in jeder Probe zu erhalten.
HINWEIS: Behandeln Sie Formaldehyd ausschließlich in der Dunstabzugshaube und tragen Sie bei der Manipulation von Proben jederzeit Handschuhe.
3. Probenfiltration
- Probenverdünnung (Exp I): Für die Wasserproben des Reservoirs ist keine Verdünnung erforderlich. (Exp II): Verdünnen Sie die Trap-Flüssigkeitsprobe 10x-100x mit partikelfreiem MQ-Wasser, um eine geeignete Verteilung der Zielmikroben auf Filteroberflächen zu erreichen, bevor sie über die Epifluoreszenzmikroskopie gezählt werden.
- Filtern Sie 1-2 ml (Exp I) des Reservoirwassers oder 10-30 l (Exp II) der Unterprobe der Trapflüssigkeit für die bakterielle Zählung auf schwarze Filter mit einer Porengröße von 0,2 m, wobei ein Filtertrichter (25 mm Durchmesser) verwendet wird.
- Die Filter mit DAPI (4',6-diamidino-2'-phenylindole dihydrochlorid, 0,2% Endkonzentration) für 4 min färben.
HINWEIS: Vermeiden Sie Haut- und Arbeitsflächenkontamination und tragen Sie Handschuhe. - Legen Sie den Filter mit konzentrierten Mikroben auf einen Tropfen Tauchöl (für die Fluoreszenzmikroskopie) auf einem Mikroskopschlitten. Legen Sie einen weiteren Öltropfen auf die Mitte des Filters und decken Sie mit einem Deckel, um sicherzustellen, dass das Öl gleichmäßig verteilt ist.
- An dieser Stelle, vorzugsweise Prozessproben sofort, oder alternativ, lagern Sie sie im Gefrierschrank (-20 °C) für mehrere Wochen bis Monate bis zu einer weiteren Analyse.
4. Aufzählung der Bakterienzahlen auf den Filtern
- Platzieren Sie den Schlitten unter das Epifluoreszenzmikroskop (mit dem Filtersatz, der dem Fluorchrom-DAPI entspricht). Legen Sie ein 10 x 10 Zählgitter in eines der Okularen. Verschieben Sie die Folie in eine zufällige Position.
- Quantifizieren Sie Bakterienzellen (blaue Fluoreszenz) im Bereich des Zählgitters (unter der Vergrößerung 1000x). Schließen Sie in die Zählungen die Zellen ein, die den linken und oberen Rand des Zählrasters kreuzen, während diejenigen, die sich über den rechten und unteren Rand befinden, ausgeschlossen sind.
- Wechseln Sie zu einer anderen zufälligen Position und wiederholen Sie die Aufzählung auf mindestens 10-15 Zählrastern, die insgesamt 500 Zellen zählen.
- Erstellen Sie einen Umrechnungsfaktor für ein gegebenes Mikroskop und eine Vergrößerung, basierend auf dem Wissen über das Verhältnis der Fläche eines Rasters zur gesamten effektiven Filtrationsfläche des Filters. Teilen Sie dann die Gesamtzahl der Zellen, die durch die Anzahl der gezählten Gitter gezählt werden, wodurch eine durchschnittliche Anzahl von Bakterien pro Raster ergibt.
- Multiplizieren Sie den letztgenannten Parameter mit dem ermittelten Umrechnungsfaktor und normalisieren Sie die resultierende Anzahl pro ml einer Probe (abhängig vom Volumen der gefilterten Probe), um die gesamte bakterielle Häufigkeit pro ml zu erhalten.
5. Bestimmung der Protistan-Fülle
- Befestigen Sie die Wasserproben (Exp I) oder Trap-Flüssigkeit (Exp II) mit Glutaraldehyd (1% Endkonzentration, besser geeignet für Proben mit dem Vorhandensein von Chlorophyll enthaltenden Partikeln, die Tage bis Wochen nach der Fixierung verarbeitet werden) oder Formol-Thiosulfat-Dekofarbierungstechnik unten angegeben.
HINWEIS: Beide Konservierungstechniken verhindern die Verunglimpfung des aufgenommenen Materials aus Nahrungsvakuumen von Protisten8. - Fügen Sie in Bezug auf die Formol-Thiosulfat-Dekofarbierungstechnik 100 l/1 l der Lugol-Lösung in 20 ml/200 l der Unterprobe Wasser/Trap-Flüssigkeit (Exp I/Exp II) hinzu.
- Folgen Sie sofort mit der Zugabe von 0,5 ml/50 l Borat-gepuffertes Formalin dann 20 l/2 l l von 3% Natriumthiosulfat (Exp I/Exp II).
HINWEIS: Das Natriumthiosulfat entfärbt die gelbe Farbe von Lugol, um die Beobachtung von Zellen unter dem Epifluoreszenzmikroskop8zu ermöglichen. - Filtern Sie ein bekanntes Probenvolumen (abhängig von der Anzahl der Zielprotisten) auf 1 m Poren-große Schwarze-Polycarbonat-Filter.
- Schätzen Sie die Anzahl der protistischen Arten ist durch Zählen von mindestens 200 Zellen unter Vergrößerung 600x mit dem Zählgitter (siehe oben).
- Reduzieren Sie das Volumen der Probe unter einem Vakuum im Filtrationstrichter durch niedriges Vakuum auf ca. 2 ml. Dann lassen Sie den Unterdruck los und fügen Sie das DAPI-Fluorchrom (4',6-Diamidino-2-Phenylindole-Dihydrochlorid13, 0,2% Endkonzentration) für 2 min hinzu.
6. Bestimmung der Gemeinschaftsstruktur von Zilien in Planktonproben
HINWEIS: Ciliate Gemeinschaften in Süßwasser-Lebensräume sind sehr vielfältig14,15,16,18, und ihre mikroskopische Bestimmung ist eine Herausforderung. Die Sortierung der Ziliengruppen in funktionelle Gilden10,14,16,17 ermöglicht eine detailliertere Analyse verschiedener Ziliengruppen als pelagische Bakterivoren.
- Bewerten Sie die Ciliate-Community-Struktur, indem Sie Folgendes kombinieren:
- DAPI-gefleckte Proben in der Epifluoreszenzmikroskopie (zur Lokalisierung der Ziliatzellen mit heller Fluoreszenz von Makro- und Mikrokernen unterschiedlicher Größe und Morphologie) kombiniert mit der Aufnahme fluoreszierend markierter Bakterien (FLB8; für Details, siehe unten), die Fähigkeit von Ziliaten verfolgen, sich von Bakterien zu ernähren.
- Live-Probenbeobachtung in ausgewählten Fällen17,19. Weitere Einzelheiten zu den oben genannten Ansätzen und Kriterien für die Gruppierung von Zilien in verschiedene taxonomische Kategorien finden Sie in früheren Veröffentlichungen16,17.
ANMERKUNG: Zitierte Studien haben gezeigt, dass unter den Zilien, Omnivoren arten aus Stichotrichia (Genera Halteria und Pelagohalteria)und Oligotrichia (nämlich Rimostrombidium spp.) die wichtigsten pelagischen Verbraucher von Bakterioplankton in einer überwiegenden Mehrheit der Süßwasserlebensräume10,17,18.
7. Schätzung der Zilien-Weideraten
- Berechnen Sie die Aufnahmeraten von Zilien auf Bakterien auf der Grundlage von Veränderungen in der durchschnittlichen Anzahl von Tracer [d.h. FLB8 pro Zilit im Zusammenhang mit der Zeit der Inkubation (5-15 min)] und Tracer Menge an FLB hinzugefügt, wobei höchstens 5%-15% der gesamten Bakterien.
- Um die Aufnahmeraten zwischen verschiedenen Protistan-Arten zu vergleichen, normalisieren Sie die Aufnahmeraten als Anzahl der Bakterien pro Zilit pro Stunde, wobei Berechnungen auf der Grundlage der tatsächlichen Inkubationszeit und des Anteils des Tracers FLB hinzugefügt werden.
ANMERKUNG: Das allgemeine Schema der ANWENDUNG der FLB-Methode zur Schätzung sowohl zell- als auch artspezifischer und massenhäliger Bakterbakterienraten in natürlichen Proben ist in Abbildung 1dargestellt. - Herstellung von FLB aus Bakterienstämmen, die in einer Süßwasserumgebung heimisch sind8
- Wählen Sie eine geeignete Größe (mittleres Zellvolumen = MCV) und Morphologie von Bakterien, so dass sie effektiv die typischen Größen von Bakterioplankton/Bakterienzellen im untersuchten aquatischen System imitieren.
HINWEIS: Für Exp I wurde eine Mischung aus isolierten Stämmen aus dem Studienort der Gattung Limnohabitans und Polynucleobacter verwendet (d. h. typisch, reichlich Bakterioplankton in Seen und Teichen)20. Einzelheiten zur Morphologie und Größe der Stämme finden Sie in früheren Publikationen3,17,18,21. - Ernten Sie Bakterienzellen aus der Kultur durch Zentrifugation (5.000 x g) für 15 min in der frühen stationären Phase und mischen Sie sie in einem numerischen Verhältnis, das MCV - SD der Zellen in der Mischung ergibt, die der typischen MCV von Bakterien an der gewählten Stelle entspricht.
- Die Pellets in 10 ml phosphatgepufferter Saline (PBS; pH = 9) aussetzen.
- 2 mg des gelbgrünen Fluoreszenzfarbstoffs 5-(4,6-dichlorotriazin-2-yl)-Aminofluorescein (DTAF, bindet an Proteine) in die Zellsuspension im Phosphat-Salzin-Puffer geben und in einem 60 °C-Wasserbad für 2 h brüten.
- Zentrifugieren Sie nach der Inkubation die Zellen nach unten, dekantieren Sie die DTAF-Lösung und waschen und zentrifugieren 3x mit PBS.
- Nach der letzten Wäsche die Zellen in 20 mldes PP i-Saline-Puffers wieder aufhängen.
- Die FLB-Aufhängung und Pipette 1,5 ml in 2 ml Kryo-Fläschchen vortex, dann gefroren(bei -20 °C) im PP i-Salin-Puffer bis zum Gebrauch.
- Vorfilter PPi-saline Puffer durch einen 0,2 m Polycarbonatfilter für den nächsten Schritt.
- Zur Bestimmung der FLB-Konzentration einen kleinen Aliquot (in der Regel 20-40 l) auf 2 ml partikelfreienPP-I-Salz-Pufferübertragen, bei 30 W für mehrere 2 s Bursts beschallen und auf 0,2 m Polycarbonat-Schwarzfilter zur Aufzählung über Epifluoreszenz filtern Mikroskopie (1.000X Vergrößerung) unter optischen Filtereinstellungen für DTAF (448 nm/520-540 nm).
- Wählen Sie eine geeignete Größe (mittleres Zellvolumen = MCV) und Morphologie von Bakterien, so dass sie effektiv die typischen Größen von Bakterioplankton/Bakterienzellen im untersuchten aquatischen System imitieren.
- Tracer-Technik zur Schätzung von Zilien-Bacterivory
- Für Weideexperimente in natürlichen planktonischen Lebensräumen 300 ml Proben in gut geschnittene 1 L-Flaschen geben und bei In-situ-Temperatur 15 min inkubieren (damit sich Protisten vom Handhabungsschock erholen können).
- Fügen Sie FLB-Tracer hinzu, um 5%-15% der gesamten Bakterien zu machen, wobei die Mengen je nach Jahreszeit und Wassertemperatur hinzugefügt werden.
ANMERKUNG: Es gibt ein sehr breites, saisonabhängiges Spektrum in ziliten artenspezifischen Aufnahmeraten, die mehrere Größenordnungen umfassen (d. h. von 101-104 Bakterien zililiat-1 pro Stunde)10,16, 17,18,22,23,24. - In Zeiten des verstärkten Auftretens von Zilien mit hohen Aufnahmeraten (in der Regel im Sommer) führen Sie auch eine parallele Inkubation mit sehr geringen FLB-Zusätzen durch, die nur 2%-4% der Gesamtbakterien ausmachen, um eine übermäßige Belastung von Ziliolen durch Tracer FLB zu vermeiden (siehe Beispiele in Abbildung 2).
- Zilien-/Planktonproben mit FLB für 5-15 min inkubieren.
- Es gibt zwei Möglichkeiten zur Probenfixierung, um die Zeutung des aufgenommenen Materials aus Nahrungsvakuumen von Protisten zu verhindern8. Beenden Sie die Inkubationen durch Zugabe von 1% Glutaraldehyd (Endkonzentration, die besser für Proben mit Chlorophyll-haltigen Partikeln, wie Algen, geeignet ist). Alternativ können Sie die Lösung von Lugol mit 100 l/10 l in eine Unterprobe von 20 ml/200 l Wasser/Trap-Flüssigkeit verwenden, gefolgt von der Zugabe von 0,5 ml/10 l Borat-gepuffertem Formalin und dann 200 l/2 l von 3 % Natriumthiosulfat (Exp I/Exp II).
- Nach Zugabe des Fixativs lassen Sie die Proben mindestens 1 h im Dunkeln bei 4 °C ruhen, um eine gründliche Konservierung der Zilitzellen zu gewährleisten.
- Nehmen Sie natürliche Plankton-Subsamples von 4-30 ml/10-30 l (Exp I/Exp II; das Volumen hängt von der Ciliath-Fülle ab) und Färben mit DAPI (Endkonzentration 0,2% wt/vol; für Details siehe Schritt 3.2 oben).
- Durchqueren Sie 1 'm schwarz Filter und inspizieren Sie über die Epifluoreszenzmikroskopie, um Ziliarate zu zählen (600x Vergrößerung) und die Anzahl der aufgenommenen FLB-Tracer (meist bei 1000-facher Vergrößerung) aufzuzählen, wie in früheren Veröffentlichungen2,17 . Proben innerhalb von 7 Tagen nach konservierungsnehmen.
- Um die gesamte protozoanische/artspezifische Beweidung zu schätzen, multiplizieren Sie die durchschnittlichen Aufnahmeraten aller Zilien oder nur der Zilienarten, wie sie durch in situ-Überfluss nachgewiesen werden.
- Das Beispiel für die Berechnung der Aufnahmeraten pro Zelle aus In-situ-Daten aus dem Wasserreservoir von Mov wird wie folgt beschrieben:
- Angenommen, die bakterielle Konzentration beträgt 3,55 x 106 Bakterien/ml und Tracer FLB hinzugefügt ist 0,25 x 106 FLB/ml, was eine Summe von 3,8 x 106 Bakterien/ml der gesamten bakteriellen Partikel ergibt (natürliche Bakterien + FLB = 100% der Beutepartikel) phagotrophischen Protisten in der natürlichen Probe zur Verfügung stehen.
HINWEIS: Die hinzugefügten FLB-Tracer machen somit 6,58 % (ein Projekt von 0,25/0,038) der gesamten bakteriellen Partikel aus. Die durchschnittliche Anzahl der FLB pro Halteria sp. beträgt 6,2 FLB in 5 min Inkubationen. - Um die Aufnahme pro Stunde zu normalisieren, verwenden Sie die folgende Berechnung: (6,2 x 12)/(6.58/100) = 1131 Bakterien pro Halteria-Zelle/h.
ANMERKUNG: Weitere Beispiele für die Verteilung einzelner Aufnahmeraten von Halteria sp., die unter variablen Wassertemperaturen, Mengen (in Prozent) des Tracer FLB und unterschiedlicheinkubationszeiten mit FLB nachgewiesen wurden, finden Sie in Abbildung 3.
- Angenommen, die bakterielle Konzentration beträgt 3,55 x 106 Bakterien/ml und Tracer FLB hinzugefügt ist 0,25 x 106 FLB/ml, was eine Summe von 3,8 x 106 Bakterien/ml der gesamten bakteriellen Partikel ergibt (natürliche Bakterien + FLB = 100% der Beutepartikel) phagotrophischen Protisten in der natürlichen Probe zur Verfügung stehen.
Ergebnisse
Beispiel Experiment Ich wurde in der Wasserreservoir von 'mov (Südböhmen, CZ) durchgeführt, die eine natürliche Stätte mit geringerer natürlicher in situ Raubtier und Beute Fülle ist. Repräsentative Daten werden für die Allesfresser-Zilienart Halteria grandinellagemeldet, die ein reichlich vorhandener und effizienter Weidevon Picoplanktonpartikeln (<2 m) ist10,16,17,18 ,22. Abbildung 3 zeigt Box-and-Whisker-Plots von FLB-Zahlen pro Zelle von Halteria sp. aus dem Reservoir von 'mov ' mov '( Abbildung 3A), die auf raten der bakteriellen Aufnahme pro Stunde neu berechnet wurde (Abbildung 3B) in vier Einzelexperimenten, die im April, Mai, August und September durchgeführt wurden. Die Zilienaufnahmeraten waren sehr unterschiedlich, was hauptsächlich auf die zeitlichen Unterschiede in der Wassertemperatur verursachte.
Es sei darauf hingewiesen, dass der Parameter Q10 die Tatsache widerspiegelt, dass die mikrobiellen Prozesse etwa 2,5x schneller laufen, mit einem Temperaturanstieg von 10 °C12, was auch für Zilienaufnahmeraten bei Bakterien gilt. Unter Berücksichtigung dieser physiologischen Regel wurden für verschiedene Jahreszeiten sehr unterschiedliche Anteile an FLB und Inkubationszeiten verwendet (Details siehe Abbildung 3A). So wurde der erwartete Temperatureffekt kompensiert, und die experimentelle Einstellung ergab optimierte Durchschnitts- und Medianwerte der Aufnahmeraten etwa zwischen 5-10 FLB pro Zilitenzelle. Im Allgemeinen sind diese Mengen der aufgenommenen FLB leicht zählbar (siehe Beispiele in Abbildung 2, zwei linke Fotografien), wodurch genaue Schätzungen des Tracers (meist zwischen 1-15 FLB pro Zilit) Aufnahmeraten generiert werden. Aufgrund der modifizierten FLB-Tracer hinzugefügt (%) und unterschiedliche Zeiten der Probeninkubation unterschieden sich die absoluten Werte (ausgedrückt als Anzahl der untersuchten Bakterien ziliiert pro Stunde) signifikant (p < 0,01, Kruskal-Wallis-Test; gefolgt von Dunns Mehrfachvergleichstest, p < 0,05; siehe Beispiele in Abbildung 3B) unter den Experimenten. Die Daten veranschaulichen auch die typische natürliche Variabilität der absoluten Bakterienraten in den planktonischen Populationen von Halteria grandinella, wobei sie ihren mittleren und mittleren Werten nahe kommen (Abbildung 3).
In Gegenwart von hocheffizienten bakterivoren Ziliaraten in Proben, wie peritrichous Ciliates, können sie von FLB in typischen Tracermengen von 5%-10% der Gesamtbakterien stark "überetikettiert" werden (siehe rechte Seite Foto in Abbildung 2). Dies kann die genaue Quantifizierung der aufgenommenen FLB stark einschränken. In solchen Fällen wird vorgeschlagen, zusätzliche parallele Inkubationen durchzuführen, wobei nur geringe FLB-Mengen nur 1,5%-3% der gesamten Bakterien ausmachen. Im Allgemeinen können jedoch sowohl die Tracermengen als auch die Inkubationszeiten manipuliert werden, um die Anzahl der FLB pro Zelle zu optimieren (Abbildung 2).
Beispielexperiment II: Gezeigt werden die Daten eines Systems mit großen Raubtier- und Beutemengen, bei dem nur ein extrem geringes Probenvolumen zur Verfügung steht, um die Bakterienraten der Ziliten Tetrahymena utriculariae experimentell zu schätzen. 25. Es ist ein moderater bakterieller Weide, der in hohem Überfluss ausschließlich in Fallen fleischfressender Utricularia reflexa Pflanzenlebt 26,27. Abbildung 4 zeigt Box-and-Whisker-Plots der Anzahl der FLB pro Zelle von T. utriculariae unter verschiedenen experimentellen Einstellungen (Abbildung 4A,B), die in Raten der bakteriellen Aufnahme pro Stunde neu berechnet werden (Abbildung 4 C,D) in jungen, reifen und alten Fallen nachgewiesen werden. Interessanterweise wurden in Fallen Chloroplasten tragende Populationen des Ziliats T. utriculariae nachgewiesen, während apochlorhaltige Populationen von T. utriculariae aus Fallen isoliert und auf gemischten bakteriellen Suspensionen gehalten wurden, die auf Weizenkörner im Dunkeln (Details siehe Abbildung 1 in einer früheren Veröffentlichung26).
Die Chloroplasten-tragenden Populationen leben in lichtbeleuchteten Fallen; So können die Chloroplasten dem Ziliatwirt eine zusätzliche organische Kohlenstoffquelle und Sauerstoff liefern. Eine der getesteten Hypothesen war, dass die apochlorischen Zilienpopulationen Bakterien deutlich schneller grasten, da die Bakterien die einzige Partikelquelle organischen Kohlenstoffs darstellen, der dunkel gewachsenen isolierten Subpopulationen des Ziliats zur Verfügung steht.
Zwar gab es keine signifikanten Unterschiede bei den Bakteriumsraten der Zilien, die in jungen, reifen und alten Fallen von Utricularia reflexa lebten (Abbildung 4A,C), die apochlorischen Populationen von T. utriculariae beweideten Bakterien signifikant (p < 0.01, Kruskal-Wallis-Test; gefolgt von Dunns Mehrfachvergleichstest, p < 0,05), etwa 3x schneller als die Chloroplasten tragenden Ziiliaate, die in jungen, reifen und alten Fallen leben (Abbildung 4C,D). Beachten Sie, dass wiederum sowohl die Tracer-Mengen als auch die Inkubationszeiten(Abbildung 4A,B, oben) geändert wurden, um die Anzahl der FLB pro Zelle zu optimieren (in der Regel zwischen 1-15), mit Durchschnitts- und Medianwerten um 5 FLB/Ziliat. Diese Zahlen sind in Zilien-Lebensmittel-Vakuolen unterscheidbar und erlaubt eine genaue Tracer-Zählung. In absoluten Mengen von Bakterien, die pro Stunde grasen, wurden jedoch die Chloroplasten- und Apochlorpopulationen etwa 350 bzw. 1.000 Bakterien pro Stunde grasen lassen. Diese versuchsweise führte zu neuen Erkenntnissen in die metabolischen und physiologischen Merkmale zweier unterschiedlicher Subpopulationen derselben Zilienarten, die unter auffallend unterschiedlichen Umweltzwängen leben25,26, 27.
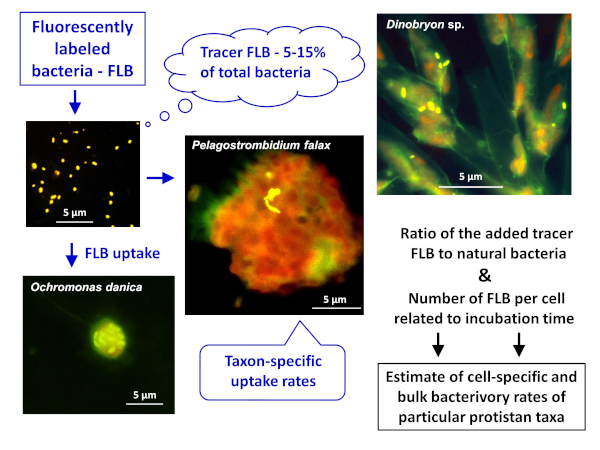
Abbildung 1 : Workflow der Verwendung von fluoreszierend gekennzeichneten Bakterien (FLB) zur Schätzung zell- und artspezifischer Weideraten aus dem Verhältnis des aufgenommenen Tracers FLB zur Gesamtzahl der natürlichen Bakterien in der Probe. Weitere Informationen finden Sie in Abschnitt 7 des Protokolls. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2 : Beispiele für Zilienzellen aus Plankton eines eutrophen Fischteichs. Beispiele werden aus dem Teich mit zählbarem FLB in Zilienzellen (in der Regel 1-10 Tracer FLB pro Zelle, die linken zwei Mikrophotograps) im Vergleich zu einem peritreichen Zilit Pelagovorticella natans (die rechte Seite Mikrofotografie) gezeigt. Selbst während einer kurzen, 5 min Inkubationszeit wurde es vom Tracer FLB "überetikettiert", was die Quantifizierung des aufgenommenen FLB ungenau oder fast unmöglichmachte. In diesem Fall wird vorgeschlagen, die Tracer-Menge auf 1,5%-3% der gesamten Bakterien zu verringern. Im Allgemeinen können jedoch sowohl die Tracermengen als auch die Inkubationszeiten manipuliert werden, um die Anzahl der pro Zelle aufgenommenen FLB zu optimieren. Weitere Informationen finden Sie in Abschnitt 7 des Protokolls. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3 : Box-and-Whisker-Plots mit FLB-Nummern pro Zelle von Halteria sp. aus dem Reservoir von Exp I (A), neu berechnet nach Raten der bakteriellen Aufnahme pro Stunde (B). Die Daten wurden unter verschiedenen saisonalen Einstellungen ermittelt, die durch vier Beispiele von April bis September dargestellt wurden. Die Oberseite des Panels A zeigt Informationen über die Wassertemperatur, verschiedene FLB-Tracer (%) und verschiedene Zeiten der Probeninkubation. Es sollte beachtet werden, dass die beiden letztgenannten Parameter geändert werden können, um die Anzahl der FLB pro Zellen zu optimieren, mit durchschnittlichen (vollzeiligen) und mittleren (gestrichelten Linien) Werten etwa zwischen 5-10 FLB pro Zilitzelle (A). Die Balken zeigen das 25. und 75. Perzentil aller Daten (50-180 zellen inspizierte) und Schnurrhaare stehen für das 1. und 99. Perzentil. (B) Verschiedene Kleinbuchstaben weisen auf signifikante Unterschiede in den zellspezifischen Bakteriumsraten von Halteria sp. während des untersuchten Zeitraums hin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
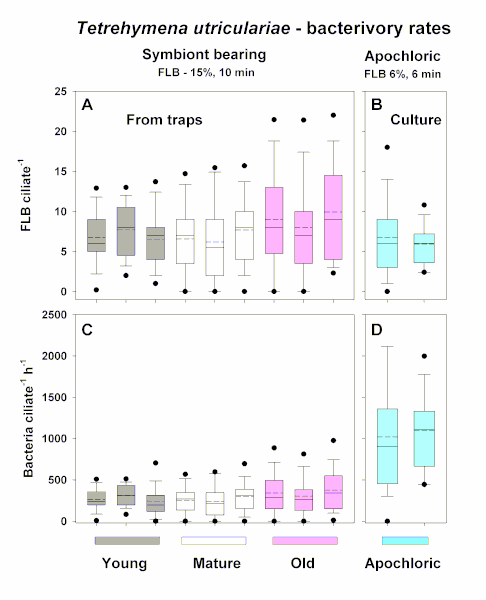
Abbildung 4 : Box-and-Whisker-Plots mit FLB-Nummern pro Zelle. Die Parzellen der Chloroplasten tragenden Tetrahymena utriculariae aus dreifachen Behandlungen von jungen, reifen und alten Fallen von Utricularia reflexa (Exp II) (A), neu berechnet auf raten der bakteriellen Aufnahme pro Stunde (C). Die Daten wurden mit den bakteriellen Aufnahmeraten der doppelten apochlorischen Populationen von T. utriculariae (B,D) verglichen, die aus Fallen isoliert, aber auf gemischten bakteriellen Suspensionen gehalten wurden, die auf Weizenkörnern im Dunkeln wachsen. Auf der Oberseite der Panels A und B wurden verschiedene FLB-Tracer hinzugefügt (%) und verschiedene Zeiten der Probeninkubation angezeigt. Es sollte beachtet werden, dass die beiden letztgenannten Parameter geändert wurden, um die Anzahl der FLB pro Zellen zu optimieren, mit durchschnittlichen (vollzeiligen) und mittleren Werten (gestrichelte Linie) etwa zwischen 5-10 FLB pro Zilitzelle (A,B). Die Balken zeigen das 5. und 95. Perzentil aller Daten (50-100 zellen inspizierte), und Schnurrhaare stehen für das 1. und 99. Perzentil. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Entschlüsselung der trophischen Interaktion in aquatischen Systemen ist immer eine Herausforderung28, vor allem auf der Nano-Plankton-Skala mit Protisten und ihre Beute, Bakterien. Wenn es um Nährstoffaufnahmewege und Quantifizierung geht, ist die Anwendung von Methoden, die erfolgreich auf höheren trophischen Ebenen eingesetzt werden, aufgrund der hohen Komplexität biotischer Wechselwirkungen weniger möglich. Dazu gehören beispielsweise stabile Isotopenkennzeichnungsansätze. Dieses Protokoll zeigt die Vorteile der Verwendung von Epifluoreszenzmikroskopie und fluoreszierend gekennzeichneten Bakterien als Tracer zur Verfolgung und Halbquantifizierung/Schätzung des Kohlenstoffflusses (bakterielle Beute: verschiedene Protistan-Weiden einschließlich mixotropher Grazer29) Wege durch die Basis von mikrobiellen Nahrungsweben. Ein solcher Vorteil ist die hohe Genauigkeit des einzelligen Ansatzes, und der andere ist die beispiellose Auflösung in Bezug auf die Struktur der Weidegemeinschaft und die Unterscheidung zwischen verschiedenen funktionellen Gilden, Arten (Exp I) und sogar Subpopulationen derselben Arten (Exp II).
Kritische Schritte im Protokoll
Es gibt mehrere kritische Schritte im Protokoll, die sicherstellen können, dass die Vorteile der Methodik voll ausgeschöpft werden. Erstens ist ein grundlegendes Verständnis der untersuchten Umgebung vor Beginn des Experiments immer von Vorteil. Dazu gehören mikroskopisches Screening der Vielfalt und Fülle potenzieller Weiden, bakterielle Beutegrößen und Beuteverteilung sowohl 1) in der Wassersäule (z. B. ein vertikales Profil vom Epizipimnion bis zum Hypolimnion) als auch 2) im Falle von Canyon-förmigen Reservoirs, auf dem Damm-Zufluss-Transect. Zweitens sorgt eine sorgfältige Manipulation mit gesammelten Proben für repräsentative Ergebnisse. Die Temperatur ist ein äußerst wichtiger Faktor, der die meisten mikrobiellen Prozesse beeinflusst12, einschließlich der protistischen Weideraten (Abbildung 3).
Drittens wird durch die Manipulation der Menge des Tracers, der auf der Quantifizierung von Bakterienzellen oder der Art des Weidetiers in der Probe hinzugefügt wird, sichergestellt, dass Probleme mit der Überbeschriftung (Abbildung 2) beseitigt werden. Es sei darauf hingewiesen, dass es ein sehr breites Spektrum an ziliten artenspezifischen Aufnahmeraten gibt (Details siehe Schritt 7.2); Um das Protokoll angemessen anzuwenden, ist daher eine vorkenntnisseweise Kenntnis der wichtigsten Zilienarten mit ihren Zeitlichen Aufnahmeraten unerlässlich. Es wird dringend empfohlen, Vorversuche mit unterschiedlichen Tracer-Mengen durchzuführen, um eine mögliche Zilien-Unterbeschriftung zu vermeiden (keine oder zu wenige FLB werden pro Zilientzelle aufgenommen, was statistisch unsolide Daten liefert) oder Überbeschriftung (erscheint als große Anzahl von FLB Die Bildung von "kondensierenden FLB-Wolken" oder Herden in Zilien-Lebensmittel-Vakuolen, die von den Tracern verpackt werden, wodurch ihre genaue Quantifizierung stark eingeschränkt wird; siehe oberes rechtes Beispiel in Abbildung 2). Es sollte auch beachtet werden, dass die Inkubationszeiten mit FLB in der Regel kürzer als 30 min sind, da die durchschnittliche Verdauungszeit von Picoplankton durch Ziliarate etwa 1,5 h beträgt und die Verdauung beginnt (aufgenommene Picoplanktonzellen verlieren ihre typische Form und Farbe) nach 45-60 min30. Ebenso muss eine optimale Verdünnung und Verteilung der Proben auf dem Filter vor der mikroskopischen Betrachtung erreicht werden, um genaue Ergebnisse zu erzielen.
Änderungen und Fehlerbehebung
Die wichtigsten Schritte, mögliche Änderungen und Änderungen bei der Fehlerbehebung an der Technik sind in Abbildung 1 und Abbildung 2dargestellt. Darüber hinaus ist zu beachten, dass bei hohen Konzentrationen von Detritalpartikeln, Phytoplanktonzellen oder deren Kolonien in Plankton solche Proben entsprechend verdünnt werden sollten, um ein Stadium zu erreichen, in dem einzelne Weidezellen auf der Filteroberfläche unterschieden und 2) der Quantifizierung des Lebensmittelvakuumgehalts unterzogen werden.
Einschränkungen
Die Hauptbeschränkung für die erfolgreiche Anwendung dieser Methode liegt in der Anwesenheit verschiedener organischer Detritus oder reichlich anorganischer/organischer Partikel mit angeschlossenen Bakterien oder Aggregaten in Mengen, die eine klare Probenbetrachtung unter der Epifluoreszenz verhindern. Mikroskop und genaue Schätzung einer hinzugefügten Tracermenge. Es sei darauf hingewiesen, dass die vorgestellte Tracer-Technik in erster Linie mit freien (d. h. suspendierten) Bakterien arbeitet, die nicht an Partikeln befestigt sind. Jedoch, basierend auf unseren eigenen Erfahrungen und Literaturreferenzen (siehe frühere Veröffentlichungen2,4,8,10,16,18,21 ,26), die vorgestellte Methodik ist für die meisten aquatischen Umgebungen geeignet. Beispiele für zwei natürliche, kontrastierende Systeme, die sich in trophischem Status, Detritusgehalt und Grasvielfalt und -zahlen unterscheiden (Abbildung 3 und Abbildung 4).
Bedeutung des Ansatzes in Bezug auf bestehende Methoden
Wichtig ist, dass aus der Kenntnis des Überflusses eines Taxons/Taxa von Bakterivoren und ihrer artspezifischen Bakterivory-Sätze der Massenbakteriumssatz des Protistan-Taxons (oder der totalen Ziliat-Assemblage) berechnet werden kann. Wenn dieser Ansatz auf natürliche Planktonumgebungen angewendet wird, die gleichzeitig sowohl für heterotrophe Flagellate als auch für Ziliate (die die Hauptgrazer von Bakterioplankton2,6,7) darstellen, Die grad-induzierte Umkleben von Bakterienpopulationen in einer bestimmten Umgebung kann geschätzt werden16,17,18,22. Solche Daten sind für die Schätzung der Kohlenstoffflussdynamik in mikrobiellen Nahrungsbahnen von grundlegender Bedeutung.
Zukünftige Anwendungen
Es gibt andere spezifische Umgebungen, in denen diese Methode mit einigen Änderungen erfolgreich verwendet werden kann. Dazu gehören Belebtschlammsysteme, Pansenökosysteme, Wassersedimente und hypertrophe Fischteiche17. Die Anwendung in diesen nährstoff- und mikrobenreichen Umgebungen erfordert jedoch vorläufige Tests, um das Protokoll hinsichtlich der richtigen Größe, Morphologie und Anzahl von Tracer FLB zu optimieren, die die typische Größenverteilung und andere Eigenschaften imitieren können. von Beutebakterien, die der Umwelt inhärent sind.
Derzeit wächst das Interesse, diesen Ansatz mit der katalysierten Reporter-Depositionsfluoreszenz-In-situ-Hybridisierung (CARD-FISH) zu kombinieren, bei der die Identität der Weidezelle (z. B. heterotrophes Flagelat) mit einer spezifischen FISH-Sonde und die Aufnahmerate basiert auf FLB-Gehalt in Lebensmittelvakuolen der Flagellate-Zelle auf dem gleichen mikroskopischen Dia31. Ein ausgeklügelter, neuer Ansatz namens Doppelhybridisierung32 ist eine Kombination von FISH-Sonden auf der Ebene der Raubtierzellen und Beutebakterien (die auch speziell durch einen phylogenetischen Stamm, eine bakterielle, linienspezifische FISH-Sonde, gekennzeichnet sind). Der Ansatz ist elegant, aber auch zeitaufwändig und erfordert spezifische Fähigkeiten und Erfahrung31,32, während die Anwendung von verschiedenen FLB-Aufnahmeansatz Modifikationen leichter für den routinemäßigen Einsatz in Laboratorien angenommen werden können.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Studie wurde von der Tschechischen Wissenschaftsstiftung im Rahmen des Forschungsstipendiums 13-00243S bzw. 19-16554S unterstützt, das an K. S. bzw. D. S. verliehen wurde. Dieser Artikel wurde auch durch das Projekt "Biomanipulation als Instrument zur Verbesserung der Wasserqualität von Staudämmen" (Nr. CZ.02.1.01/0.0/0.0/16_025/0007417) unterstützt, das aus dem Europäischen Fonds für regionale Entwicklung im Rahmen des Operationellen Programms Forschung, Entwicklung finanziert wird. und Bildung.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.2-µm pore-size filters | SPI supplies, https://www.2spi.com/ | B0225-MB | Black, polycarbonate track etch membrane filters, diameter approprite for filtering apparatus used |
| 5-(4,6-dichlorotriazin-2-yl) aminofluorescein (DTAF) | Any brand | ||
| Automatic pipettes with adjustable volumes | Any brand, various sizes | ||
| Centrifuge | 22 000 x g | ||
| Cryovials | Any brand, 2 mL size | ||
| DAPI (4´,6-Diamidino-2´-phenylindole dihydrochloride) | Any brand | 1 mg ml-1 | |
| Epiflorescence microscope | Magnification from 400 x up to 1000 x | ||
| Filters appropriate for viewing in the DAPI and DTAF range | |||
| Counting grid in one of the oculars | |||
| Filtering apparatus | Usually with a diameter of 25 mm | ||
| Formaldehyde | A brand for microscopy | ||
| Glutaraldehyde | A brand for microscopy | ||
| Immersion oil for microscopy | Specific oil with low fluorescence | ||
| Lugol´s solution | Any brand or see comment | Make an alkaline Lugol' solution as follows: Solution 1 - dissolve 10 g of potassium iodide in 20 ml in MQ water, then add 5 g of iodine. Solution 2 - add 5 g of sodium acetate to 50 ml of MQ water. Add the solution 2 to the solution 1 and thoroughly mix | |
| Methanol stabilized formalin | Any brand available for microscopy purposes | ||
| Microscope slides and cover slips | Any brand produced for microscopy purposes | ||
| MQ water for diluting samples | Any brand | ||
| Phosphate-buffered saline (PBS; pH = 9) | Any brand | 0.05 M Na2HPO4-NaCl solution, adjusted to pH 9 | |
| PPi-saline buffer | Any brand | 0.02 M Na4P2O7-NaCl solution. Add 0.53 g Na4P2O7 to 100 ml of MQ water plus 0.85 g NaCl | |
| Sampling device | Appropriate for obtaining representative sample | e.g. Friedinger sampler for lake plankton | |
| Sodium thiosulfate solution | Any brand | 3% solution is used in the protocol | |
| Sonicator | Any brand | 30 W | |
| Vortex | Any brand allowing thorough mixing of the solutes and samples | ||
| Water bath | Any brand allowing temperature to be maintained at 60 °C |
Referenzen
- Azam, F., et al. The ecological role of water-column microbes in the sea. Marine Ecology Progress Series. 10, 257-263 (1983).
- Šimek, K., et al. A finely tuned symphony of factors modulates the microbial food web of a freshwater reservoir in spring. Limnology & Oceanography. 59, 1477-1492 (2014).
- Šimek, K., et al. Bacterial prey food characteristics modulate community growth response of freshwater bacterivorous flagellates. Limnology & Oceanography. 63, 484-502 (2018).
- Šimek, K., et al. Changes in bacterial community composition, dynamics and viral mortality rates associated with enhanced flagellate grazing in a meso-eutrophic reservoir. Applied & Environmental Microbiology. 67, 2723-2733 (2001).
- Jürgens, K., Matz, C. Predation as a shaping force for the phenotypic and genotypic composition of planktonic bacteria. Antonie Van Leeuwenhoek. 81, 413-434 (2002).
- Pernthaler, J. Predation on prokaryotes in the water column and its ecological implications. Nature Reviews Microbiology. 3, 537-546 (2005).
- Berninger, U. B., Finlay, J., Kuuppo-Leinikki, P. Protozoan control of bacterial abundances in freshwaters. Limnology and Oceanography. 36, 139-147 (1991).
- Sherr, E. B., Sherr, B. F., Kemp, P. F., Sherr, B. F., Sherr, E. B., Cole, J. J. Protistan grazing rates via uptake of fluorescently labeled prey. Handbook of Methods in Aquatic Microbial Ecology. , 695-701 (1993).
- Vazquez-Dominguez, E., Peters, F., Gasol, J. M., Vaqué, D. Measuring the grazing losses of picoplankton: methodological improvements in the use of fluorescently tracers combined with flow cytometry. Aquatic Microbial Ecology. 20, 119-128 (1999).
- Šimek, K., et al. Ecological role and bacterial grazing of Halteria spp.: Small oligotrichs as dominant pelagic ciliate bacterivores. Aquatic Microbial Ecology. 22, 43-56 (2000).
- Montagnes, D. J. S., et al. Selective feeding behaviour of key free-living protists: avenues for continued study. Aquatic Microbial Ecology. 53, 83-98 (2008).
- Kirchman, D. L. . Processes in Microbial Ecology. 2nd Edition. , (2018).
- Porter, K. G., Feig, Y. S. The use of DAPI for identifying and counting aquatic microflora. Limnology and Oceanography. 25, 943-948 (1980).
- Foissner, W., Berger, H. A user-friendly guide to the ciliates (Protozoa, Ciliophora) commonly used by hydrobiologists as bioindicators in rivers, lakes, and waste waters, with notes on their ecology. Freshwater Biology. 35, 375-482 (1996).
- Foissner, W., Berger, H., Schaumburg, J. Identification and ecology of limnetic plankton ciliates. Informationsberichte des Bayer Landesamtes für Wasserwirtschaft Heft. , 3-99 (1999).
- Šimek, K., et al. Ciliate grazing on picoplankton in a eutrophic reservoir during the summer phytoplankton maximum: a study at the species and community level. Limnology & Oceanography. 40, 1077-1090 (1995).
- Skibbe, O. An improved quantitative protargol stain for ciliates and other planktonic protists. Archiv für. Hydrobiolgie. 130, 339-347 (1994).
- Macek, M., et al. Growth rates of dominant planktonic ciliates in two freshwater bodies of different trophic degree. Journal of Plankton Research. 18, 463-481 (1996).
- Šimek, K., et al. Microbial food webs in hypertrophic fishponds: omnivorous ciliate taxa are major protistan bacterivores. Limnology & Oceanography. , (2019).
- Jezbera, J., et al. Major freshwater bacterioplankton groups: Contrasting trends in distribution of Limnohabitans and Polynucleobacter lineages along a pH gradient of 72 habitats. FEMS Microbiology Ecology. 81, 467-479 (2012).
- Kasalický, V., et al. The diversity of the Limnohabitans genus, an important group of freshwater bacterioplankton, by characterization of 35 isolated strains. PLoS One. 8, 58209 (2013).
- Stabell, T. Ciliate bacterivory in epilimnetic waters. Aquatic Microbial Ecology. 10, 265-272 (1996).
- Zingel, P., et al. Ciliates are the dominant grazers on pico- and nanoplankton in a shallow, naturally highly eutrophic lake. Microbial Ecology. 53, 134-142 (2007).
- Bickel, S. L., Tang, K. W., Grossart, H. P. Ciliate epibionts associated with crustacean zooplankton in german lakes: distribution, motility, and bacterivory. Frontiers in Microbiology. 3 (243), (2012).
- Sirová, D., et al. Hunters or gardeners? Linking community structure and function of trap-associated microbes to the nutrient acquisition strategy of a carnivorous plant. Microbiome. 6, 225 (2018).
- Šimek, K., et al. Ecological traits of a zoochlorellae-bearing Tetrahymena sp. (Ciliophora) living in traps of the carnivorous aquatic plant Utricularia reflexa. Journal of Eukaryotic Microbiology. 64, 336-348 (2017).
- Pitsch, G., et al. The green Tetrahymena utriculariae n. sp. (Ciliophora, Oligohymenophorea) with its endosymbiotic algae (Micractinium sp.), living in the feeding traps of a carnivorous aquatic plant. Journal of Eukaryotic Microbiology. 64, 322-335 (2017).
- Nielsen, J. M., Clare, E. L., Hayden, B., Brett, M. T., Kratina, P. Diet tracing in ecology: Method comparison and selection. Methods in Ecology and Evaluation. 9, 278-291 (2018).
- Beisner, B. E., Grossart, H. P., Gasol, J. M. A guide to methods for estimating phago-mixotrophy in nanophytoplankton. Journal of Plankton Research. , 1-13 (2019).
- Dolan, J. D., Šimek, K. Processing of ingested matter in Strombidium sulcatum, a marine ciliate (Oligotrichida). Limnology and Oceanography. 42, 393-397 (1997).
- Massana, R., et al. Grazing rates and functional diversity of uncultured heterotrophic flagellates. The ISME Journal. 3, 588-596 (2009).
- Grujčić, V., et al. Cryptophyta as major freshwater bacterivores in experiments with manipulated bacterial prey. The ISME Journal. 12, 1668-1681 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten