Method Article
Völlig autonom Charakterisierung und Datenerfassung aus Kristallen von biologischen Makromolekülen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir, wie die automatisierte Screening und Sammlung Datenoptionen verfügbar bei einigen Synchrotron Beamlines verwenden. Wissenschaftler senden Cryocooled Proben an der Synchrotron und die Beugung Eigenschaften gezeigt, die Datensätze gesammelt und verarbeitet werden und, soweit möglich, erfolgt eine Struktur-Lösung – ganz ohne menschliches Zutun.
Zusammenfassung
Hohe Brillanz Röntgenstrahlen gepaart mit Automatisierung führten zu die Verwendung von Synchrotron-basierte makromolekularen Röntgen-Kristallographie (MX) Beamlines sogar die anspruchsvollsten Projekte in Strukturbiologie. Jedoch erfordern die meisten Einrichtungen noch das Vorhandensein eines Wissenschaftlers vor Ort die Experimente durchführen. Eine neue Generation von automatisierten Beamlines widmet sich der vollautomatische Charakterisierung und Datenerfassung aus, Kristalle von biologischen Makromolekülen hat vor kurzem entwickelt. Diese Beamlines vertreten ein neues Tool für Strukturbiologen der Ergebnisse der ersten Kristallisation Studien und/oder die Sammlung einer großen Anzahl von Datensätzen Beugung, ohne sich zu beherrschen, das Strahlrohr auf den Bildschirm. Hier zeigen wir wie ein Experiment für das automatische Screening und Datenerfassung, eingerichtet wie ein Experiment auf das Strahlrohr durchgeführt wird, wie die resultierenden Datensätze verarbeitet werden, und wie, wenn möglich, die Kristallstruktur des biologischen Makromolekül gelöst.
Einleitung
Bestimmung der dreidimensionalen Struktur von bestimmten Proteinen ist entscheidend in der Biologie. Die Informationen, die dabei abgeleitet ist beleuchtet so auf die biologische Funktion und auf die Form und die Spezifität der aktiv und verbindlich Websites enthalten im Molekül unter Studie. In vielen Fällen dadurch Wirkmechanismen ermittelt werden oder gegebenenfalls mögliche therapeutische Moleküle entwickelt werden. MX ist die am häufigsten verwendeten strukturellen Informationen zu Technik, sondern ein Engpass ist die Bestimmung des optimalen Bedingungen gut diffracting Kristalle zu erhalten. Daher Kristallisation Studien sind in zahlreichen unterschiedlichen Bedingungen durchgeführt und dann gezeigt werden, um die besten Kristalle zur Beugung Datenerfassung verwendet werden zu finden. Die Automatisierung des Aufbaus der Kristallisation Studien1 hat in dieser Hinsicht eindeutig geholfen. Jedoch sind die nachfolgenden Schritte (z.B. Kristall Montage, Beugung Screening und Beugung Datenerhebung) in der Regel manuell durchgeführt nimmt einiges an Zeit, Aufwand und Ressourcen. Die Automatisierung der Beugung Screening und Datenerfassung würde daher einen enormen Gewinn an Zeit und Effizienz bedeuten.
Beugung-Screening und Datenerfassung in MX am häufigsten Synchrotron MX Beamlines erfolgt auf auf die Automatisierung dieses Prozesses weitgehend erleichtert hat. In den meisten Fällen ist es jedoch notwendig für den Wissenschaftler in das Strahlrohr während eines Experiments anwesend oder es aus der Ferne bedienen. Vor kurzem wurde eine neue Generation der vollautomatischen MX Beamlines entwickelt2. Hier müssen Benutzer nicht körperlich oder aus der Ferne, während einer experimentellen Sitzung anwesend sein. Dies ermöglicht es Wissenschaftlern, mehr Zeit auf weniger Routineaufgaben, anstatt verbringen ganze Tage und oft Nächte, Screening-Kristalle und Beugung Datenerhebung. Der weltweit erste vollautomatische Strahlrohr ist massiv automatisierten Probe Auswahl integrierter Facility (massiv-1, ID30A-1)2,3 der European Synchrotron Radiation Facility (ESRF). Es verfügt über ein einzigartiges Beispiel Umfeld eine Hochleistungs-Probe-haltigen Dewar im Tandem mit einem Roboter Probenwechsler, das auch als das Strahlrohr Goniometer4,5fungiert. MASSIV-1 ist ein Undulator Strahlrohr ausgerüstet mit einer Single-Photon-zählen Hybrid Pixel Detektor6, das bei einer festen Wellenlänge von 0,969 betreibt Å (12.84 keV) mit einer intensiven Röntgenstrahl (2 x 1012 Photonen/s). Die Strahlgröße bei den Probenort kann zwischen einem Minimum von 10 µm (Runde Balken) zu einem Maximum von 100 µm X 65 µm (Horizontal durch vertikale Balken Größe) eingestellt werden. Im Durchschnitt kann das Strahlrohr zu verarbeiten, in eine vollautomatische Mode (siehe unten), 120 Kristalle in 24 h. Der Betrieb das Strahlrohr basiert auf eine Reihe von Workflows7, von denen jeder intelligente Entscheidungen basierend auf dem Ergebnis der vorherigen Schritte im Workflow braucht, um die Messung der möglichen Daten aus der Probe unter Studie zu gewährleisten. Insbesondere die Bewertung der Beugung Merkmale einer einzelnen Probe Konto Kristall Volumen und Flussmittel berücksichtigt und gewährleistet, wo der Kristall ist größer als der Röntgenstrahl, dass nur die beste Region des Kristalls für nachfolgende Daten verwendet wird Kollektion. Beugung von Datensätzen sind, so optimiert für maximale Auflösung mit minimierter Strahlung Schaden2,3. Anspruchsvolle Sammlung Datenprotokollen, wie Pseudo-spiralförmige (Multi-Positionsdaten) Sammlung Strategien für beide native und Einzel-Wellenlänge anomale Beugung (SAD) Datenerhebung sind auch verfügbar8.
Vollautomatische Experimente bei massiv-1 umfassen Cryocooling und Montage der Kristalle auf einer magnetischen Probe Halterung geeignet für den gewünschten Strahlrohr Ausrüstung Standard pins SPINE9, Eingabe der gewünschten experimentellen Parameter in der "Beugung Plan "Tabelle in das integrierte System für Proteinkristallographie Beamlines (ISPyB)10, ein Web-basiertes Informationsmanagementsystem für MX-Experimente, und senden Sie die Proben an das Strahlrohr. Bei der ESRF, alle Kosten für den Transport der Proben von/nach das Strahlrohr von der ESRF User Office unterstützt werden (siehe die Website der ESRF11 für Details). MASSIV-1, sind keine Beschränkungen auf die Schleifengröße oder Kristallqualität. Bei der Auswahl eines Beugung Plans für einen bestimmten Kristall kann der Benutzer Standardeinstellungen verwenden oder wählen Sie aus speziellen Workflows, die für jede Probe angepasst werden können. Es stehen mehrere vorprogrammierte Abläufe. Im MXPressE3 -Workflow wird die Probe-haltigen Schleife zuerst den Probenort mit optische Zentrierung ausgerichtet. Dann, X-ray-basierten Zentrierung gewährleistet, dass die beste Region des Kristalls zu den Röntgenstrahl zentriert ist. Daten-Sammlung-Strategien werden dann mit eEDNA, einen Rahmen für die Entwicklung von Plugin-basierten Anwendungen speziell für Online-Datenanalyse im Feld x-ray Experimente unter Konto Kristall Volumen und der Echtzeit-Flux auf das Strahlrohr berechnet. Im Anschluss an die Sammlung eines Datensatzes volle Beugung dies werden dann mit eine Reihe von automatischen Datenverarbeitung Rohrleitungen12 verarbeitet und die Ergebnisse werden im ISPyB für Inspektion und Download zur Verfügung gestellt. MXPressE SAD3 Workflow richtet sich an Selenomethionin-haltigen Kristalle des Zielproteins und nutzt die Tatsache, dass die Antriebsenergie von massiv-1 oberhalb der Se K-Kante. Hier der MXPressE eEDNA Sammlung Datenstrategie für traurig Datenerfassung optimiert ist (d. h. hohe Redundanz und mit der Auflösung von wo RZusammenführung zwischen Bijvoet Paaren unter 5 % liegt). Die Beugung Eigenschaften einer Reihe von Kristallen ohne nachträgliche Datenerfassung auf dem Bildschirm, kann die MXScore3 Workflow verwendet werden, um eine Aussage über die Kristalle analysiert voller Qualität zu produzieren. Im MXPressI3 Workflow 180° Drehung Daten gesammelt werden, mit 0,2 ° Schwingungen und mit den Startwinkel Phi und die Auflösung durch eine eEDNA Strategie bestimmt. MXPressO 3 enthält eine preobserved Auflösung in den Workflow (Standard: dmin = 2 Å). Um eine erste Einschätzung der Kristalle aus einer Kristallisation Test zu machen, wird die MXPressM3 Workflow angeboten. Dies führt eine Hochdosis-Netz über die breiteste Ausrichtung der Probenträger keine Datenerfassung scannen oder zentrieren. Vor kurzem wurden zwei neue Experiment-Workflows, MXPressP und MXPressP_SAD, die pseudohelical Datenerhebungen durchführen, implementierte8. Die Ausführung aller Schritte in allen Workflows kann online und in Echtzeit verfolgt werden durch den Benutzer, über ISPyB.
Hier zeigen wir eine vollautomatische MX-Experiment am massiv-1 vorbereiten und Gewusst wie: abrufen und analysieren der Daten, die aus dem Experiment. Als Beispiel verwenden wir menschliche Mitochondrien Glycin Spaltung System Protein H (GCSH). Dieses Lipoic Säure-haltige Protein ist Teil des Systems der Glycin-Spaltung verantwortlich für den Abbau von Glycin. Dieses System beinhaltet das P-Protein, einem Pyridoxal-Phosphat-abhängigen Glycin-Decarboxylase T-Protein, ein Enzym Tetrahydrofolate erfordern und das L Protein, ein Lipoamide-Dehydrogenase. GCSH überträgt die Methylamin-Gruppe von Glycin aus dem P-Protein mit dem T-Protein. Mängel in der H-Protein sind die Ursache für Nonketotic Hyperglycinemia (NKH) Menschen13.
Protokoll
Hinweis: Die Produktion, Reinigung und Kristallisation des GCSH sind in zusätzliche Datei 1beschrieben.
1. Kurzbeschreibung der offline Vorbereitung und Kristall-Montage
- Position einer Nylon-Schleife oder einer anderen Kristall-Halterung bereits fest mit einer Wirbelsäule Pin unter einem oder mehreren Kristallen und heben sie aus der Niederschlag-Lösung (20 µL 0,5 M Natrium Formiat pH 4.0 + 25 µL Proteinlösung).
- Entfernen Sie die Masse Flüssigkeit rund um den Crystal(s) durch Berühren der Halterung mit einem Papier-Docht, überschüssige Flüssigkeit absaugen.
- Genießen Sie die Crystal(s) in der Cryoprotective-Lösung mit dem Niederschlag Lösung plus 30 % Glycerin; dann entfernen Sie die Kristall-Unterstützung und der Crystal(s).
- Entfernen Sie die Masse Flüssigkeit rund um den Crystal(s) durch Berühren der Halterung mit einem Papier-Docht, überschüssige Flüssigkeit absaugen.
- Die Halterung in einer Wirbelsäule Fläschchen gefüllt mit flüssigem Stickstoff eintauchen und speichern es, zusammen mit anderen Kristallen, die in ähnlicher Weise zubereitet, in einem European Molecular Biology Laboratory (EMBL) / ESRF Probe Wechsler Puck9 bei Temperatur von flüssigem Stickstoff.
Hinweis: Die Crystal(s) sind stabil in diesem Zustand, bis die Strahlzeit verfügbar ist.
2. Beantragung Strahlzeit von massiv-1
- Strahlzeit so früh wie möglich auf der ESRF Homepage (http://www.esrf.eu/UsersAndScience/UserGuide/Applying) anfordern.
Hinweis: Es gibt eine Reihe von möglichen Modi des Zugangs zu der ESRF MX Beamlines. Laboratorien können gemeinsam als Teil ein Block Zuweisung Gruppe (BAG), Strahlzeit zugeteilt für 2 Jahre haben anwenden. Wenn Gruppen einzeln bewerben möchten, können sie Rollen Zugang, ermöglicht ihnen schnellen Zugriff auf die Beamlines nach Begutachtung beantragen. Vorschlag der Gruppe wird überprüft und von der ESRF Safety Group, die weitere Details anfordern kann gelöscht werden. Wenn der Vorschlag angenommen wird, werden ein Experimentnummer und das Passwort mitgeteilt. Proprietäre Forschung kann durch den Kauf von Strahlzeit durchgeführt werden. - Füllen Sie die erforderliche Sicherheit Ausbildung online (bei http://www.esrf.eu/UsersAndScience/UserGuide/Preparing/SafetyTraining).
- Buch Strahlzeit im MASSIF-1-Kalender.
Hinweis: Es ist möglich, maximal 50 Probenhalter pro Schicht analysiert werden zu buchen. - Füllen Sie in A-Form , eine Post-Experiment (http://www.esrf.eu/UsersAndScience/UserGuide/Preparing/new-a-form), zu erklären, zusammen mit den erforderlichen Informationen für die Proben, die gemessen werden sollen.
3. Erstellen von Beugung in ISPyB
Hinweis: Die Beugung Plan enthält alle Informationen, die für eine Probe in ISPyB und kann enthalten zusätzliche Informationen, um das Experiment durchgeführt für jede Probe individuell.
- Offene ISPyB (bei https://exi.esrf.fr/).
- Wählen Sie MX-Experimente.
- Das Experimentnummer und Passwort einloggen.
- Klicken Sie auf Sendung | Hinzufügen neuer und die notwendigen Auskünfte erteilen. Klicken Sie auf Speichern.
- Klicken Sie auf Add Paket und geben Sie die angeforderten Informationen. Klicken Sie auf Speichern.
- Klicken Sie auf Add Container, geben Sie den Puck Barcode als den Namen, und wählen Sie Wirbelsäule Puck. Klicken Sie auf Speichern.
- Klicken Sie auf den Container Symbol und Bearbeiten, und füllen Sie die notwendigen Informationen, wie Proteinnamen, bevorzugte Workflow, Kristall-Position in den Puck usw., über die Proben.
- Wählen Sie das Protein (z. B. GCSH oder Lysozym), das von der ESRF Safety Group genehmigt wurde.
- Geben Sie einen eindeutigen Probe jeder einzelnen Probe zu identifizieren. Es ist möglich, optional den Pin Barcode scannen. Der Rest der folgenden Informationen ist optional.
- Geben Sie die optionale Informationen.
- Für jede einzelne Probe das Experiment eingeben (z.B. MXPressE_SAD, SCORE oder MXPressO, etc., Standard MXPressE) unter EXP-Typ. Dadurch wird definiert, welche automatische Workflow verwendet wird, um jeden Kristall zu verarbeiten. Angesichts der Tatsache, dass die GCSH-Kristalle Nadeln sind, wählen Sie MXPressP.
- Geben Sie eine Raumgruppe (z. B. C2, P1 und P212121), falls bekannt. Falls vorhanden, wird dies für Daten Sammlung Strategie Berechnungen und durch die automatische Datenverarbeitung-Pipelines verwendet werden.
- Geben Sie die gewünschte Auflösung (Standard: dmin = 2,0 Å). Dadurch wird den Kristall-Detektor-Abstand für die erste Masche Scans, Charakterisierung und Standard-Datensammlung definiert.
- Legen Sie die gewünschten Schwellenwert Auflösung (z. B. 1,5 Å oder 2.3Å), die Sammlung von vollständigen Datensätzen aus Kristallen zu verhindern, die dieses Limit nicht beugen zu tun. Dies spart Platz und Analyse Speicherdauer der Daten.
- Legen Sie die erforderlichen Vollständigkeit (Standard: 0,99). Festlegen die erforderlichen Vielfalt (Standardwert: 4). wenn mehr als ein Kristall auf die Probe-Unterstützung enthalten ist, legen Sie die maximale Anzahl von Kristallen analysiert werden. Der Standardwert ist 1 oder 5 für MXPressP.
- Wählen Sie die entsprechenden Balken-Größe (Standard: 50 µm). Wenn ein bestimmter Wert nicht ausgewählt ist, werden die X-ray-Zentrierung und Daten Sammlung Strategie Berechnungen mit einem Strahl von 50 µm durchgeführt.
Hinweis: Während jeder nachfolgende Auflistung von vollständigen Datensätzen, wird die Strahlgröße automatisch angepasst werden. - Setzen Sie in der Raumgruppe, wenn bekannt, in der Spalte "erzwungene Raum-Gruppe". Legen Sie die Strahlenempfindlichkeit der Kristalle (0,5-2,0 für niedrige bis hohe Empfindlichkeit mit einem Standardwert von 1).
- Falls gewünscht, stellen insgesamt Drehwinkel für die vollständigen Dataset Sammlung gesammelt werden (Standard: die gesamten Drehwinkel durch eEDNA bestimmt).
- Speichern Sie die Werte. Klicken Sie auf zurück zu Sendungen. Drücken Sie die Taste senden Sendung, ESRF.
- Drucken Sie das Versandetikett aus und senden Sie die Proben. Benutzer sollten mit einem Kurier, unter Nutzung der ESRF Kontoangaben eine Abholung vereinbaren.
Hinweis: Es ist sehr wichtig, wählen Include Rücksende-Etikett ermöglicht die nahtlose Rückkehr der Proben (siehe https://www.esrf.eu/MXDewarReimbursement).
4. Datenerfassung, Anzeige und Abfrage
Hinweis: Am Tag des Experiments werden Proben, massiv-1 hohe Kapazität Dewar (HCD) übertragen. Strahlrohr Wissenschaftler starten dann die Datensammlung, die von Benutzern aus der Ferne verfolgt werden können. Für jede unterschiedliche Probentyp erhalten Benutzer eine E-mail informiert, dass die Datenerhebung begonnen hat. Wie bereits erwähnt, die Ausführung aller Schritte in allen Workflows kann online und in Echtzeit verfolgt werden durch die Benutzer über ISPyB, aus denen die Ergebnisse angezeigt und heruntergeladen werden können.
- Untersuchen Sie für jede Probe analysiert die Ergebnisse des automatischen Experiments in ISPyB (https://exi.esrf.fr/).
- Melden Sie sich an, mit dem Experimentnummer und Kennwort, und klicken Sie auf die gewünschte experimentelle Sitzung am ID30A-1.
- Wählen Sie die bevorzugte (Top-scoring) Autoprocessing Rohrleitung (beispielsweise Granaten oder XDS_APP) und laden Sie die Daten in die richtigen Felder-Gruppe mit der höchsten Vollständigkeit und höchste Auflösung durch Anklicken der Letzten sammeln Ergebnisse ausgeschrieben und dann herunterladen.
Hinweis: Alle Mesh, Linie und Charakterisierung Bilder sind in einem Unterverzeichnis für jede Probe, genannt /MXPressE_01. Der ESRF läuft automatisch fünf getrennte Bearbeitung Pakete, nämlich EDNA_proc12, Granaten12, XDS_APP14, AutoPROC15und XIA216. Datenintegration basiert auf XDS, mit Ausnahme von XIA2, basiert auf Zifferblättern. Alle Pakete laufen auch in anomale und nonanomalous Modi, so dass die automatische Erkennung eines anomalen Signals, wenn in den Daten in SAD phasing-Protokolle verwendet werden. Jedes Paket nutzt verschiedene Parameter und Entscheidungsbäume, was bedeutet, dass einige Pakete mit bestimmten Proben besser laufen. Dies können jedoch für eine große Anzahl von Ergebnissen bei der Anzahl der Pakete und Raum Gruppen berücksichtigt wird. Die Ergebnisse sind, daher Platz etwa anhand der Auflösung und andere Qualitätsmetriken wie Rverschmelzen in der niedrigsten Auflösung Shell, CC(1/2) und Vollständigkeit. Dies richtet sich an die Führung des Benutzers an die besten Daten-Sets, aber alle möglichen Raum Gruppen und Ergebnisse sollten sorgfältig geprüft werden.
- Entpacken Sie die heruntergeladenen Ordner, darunter alle Log-Dateien und noch XDS_ASCII. HKL und fusionierte und skalierten .mtz Dateien.
Hinweis: für den Fall, dass die Struktur des Proteins des Interesses oder eine enge Homolog (im PDB-Format) in ISPyB zu Beginn des Experiments hochgeladen wurde, die Autoprocessing Pipeline am ESRF führt automatisch einen molekulare Ersatz (MR) laufen mit dieser Struktur als die Modell auf das beste erzielte Lösung zu suchen. Die Ergebnisse der Herr Pipeline werden im ISPyB angezeigt und finden Sie im Ordner "Daten" (z. B. /data/visitor/mx2112/id30a1/20180711/PROCESSED_DATA/GCSH/GCSH-x5/autoprocessing_GCSH-x5_run1_1/grenades_fastproc/user_nohet.pdb_ Mrpipe_dir /). Hier wird das endgültige Modell coot1.pdb und Reflexion Daten sidechains.mtz benannt werden. Beachten Sie, dass die Pipeline die Symmetrie der Zelle (primitive Zelle Reduktion) verringern könnte, um die Wahrscheinlichkeit eine Lösung zu finden. Im vorliegenden Fall schrieb die Herr-Pipeline, die Lösung in einem monoklinen Zelle (C2) nicht in eine orthorhombic Zelle (C2221). Details zum molekularen Ersatz ausführen manuell ausführen (beispielhaft für die zweite am besten scoring Autoprocessing Lösung) in die Zusätzlichen Dateienenthalten sind.
Ergebnisse
Der MXPressP-Workflow diente bei der ESRF Strahlrohr massiv-1, vollautomatisch, montieren, zentriert in der Röntgenstrahl zu charakterisieren und volle Beugung von Datensätzen aus einer Reihe von Kristallen des menschlichen GCSH zu sammeln. Die Proben wurden montiert und die Schleife analysiert für einen Bereich (Abbildung 1, links) zu scannen. Nach der Beugung-Analyse wurden vier Punkte im Inneren des Kristalls für die Datenerfassung (Abbildung 1rechts) ausgewählt. Folgeverarbeitung durch automatisierte Analyse Datenpipelines, einschließlich der Herr Pipeline, qualitativ hochwertige Datensätze (Tabelle 1) ergab für die eine Herr Lösung gefunden wurde. Letzteres erlaubt Benutzern, schnell beurteilen, ob die erhaltenen Dataset und der verwendeten Suchmodell für Ausstieg durch molekularen Austausch geeignet sind. Darüber hinaus kann das Vorhandensein von Liganden beurteilt werden, so dass des Benutzers nur auf die vielversprechendsten Datensätze für weitere Analysen konzentrieren. Manuelle Strukturbestimmung von Herrn ergab eine qualitativ hochwertige Elektron-Dichte-Karte nach eine einzigen Verfeinerung Zyklus (Abbildung 2a automatisierten). Für dieses Dataset schneiden Sie die automatisierte Pipeline die Daten an ein 1,32 Å Auflösung; Allerdings können Benutzer immer noch entscheiden, schneiden Sie die Daten mit einer niedrigeren Auflösung, verschiedene Statistiken (CC1/2, < I/σ(I) > RMeas) in der höchsten Auflösung-Shell zu erreichen. Die Kristallstruktur des menschlichen GCSH Struktur ist ähnlich wie die bovine Protein (3KlR)16.
Kontinuierliche Elektronendichte ist für die gesamte Aminosäurekette, abgesehen von der N-terminalen Histidin Tag sichtbar. Von den vier Ersetzungen, die menschlichen und bovine GCSH unterscheiden, sind drei leicht erkennbar in der Elektronendichte (Ile/Val66, Asp/Glu98 und Leu/Phe149; Abbildung 2 b -d). Dies ist weniger klar für den Asp/Lys125 Ersatz für den Elektronendichte der Seitenkette durch Flexibilität (Abbildung 1e) nur teilweise gelöst wird. Die derzeit erzielten Modell hat Rarbeiten undfrei R-Werte von 20,4 % und 23,8 %, bzw., und kann durch weitere Zyklen von automatisierten und manuellen Modellbau und Raffinesse weiter optimiert werden.
| Pipeline-Granaten | XDS_APP Rohrleitung | |
| Datenerhebung und-Verarbeitung | ||
| X-ray Source / Breite Linie | ESRF / MASSIV-1 | |
| Wellenlänge (Å) | 0.966 | |
| Auflösung (Å) | 41,88 – 1,48 (1,53 – 1,48) | 41.86-1,32 (1,39 – 1,32) |
| Gesamt/Unique Reflexionen | 127670 / 28644 | 177332 / 40134 |
| (12178 / 2775) | (23772 / 5714) | |
| Raumgruppe für Indizierung, Skalierung und zusammenführen | C222 | C2221 |
| Zellengröße | ||
| a, b, C (Å) | 42.20, 83.75, 95,85 | 42.19, 83.72, 95,82 |
| Mosaicity | 0.05 | 0.05 |
| RMeas (%) | 10,0 (110,7) | 11.1 (198,2) |
| < I/σ(I) > | 9.6 (1.3) | 7.6 (0,7) |
| CC1/2 (%) | 99,7 (53,9) | 99,7 (19,1) |
| Vollständigkeit (%) | 99,6 (99,6) | 99,5 (98,6) |
| Multiplizität | 4.5 (4.4) | 4.4 (4.2) |
| Molekulare Ersatz und vorläufigen Modell Raffinesse | ||
| Raumgruppe zur schrittweisen | C2 | C2221 |
| Zellengröße | ||
| a, b, C (Å) | 83.74, 42,18, 95,82 | 42.19, 83.72, 95,82 |
| Α, Β, Γ (°) | 90, 90.03, 90 | 90, 90, 90 |
| Suche Modell für Herrn (PDB) | 3KLR | 3KLR |
| Protein-Moleküle / ASU | 2 | 1 |
| Proteinrückstände | 250 | 125 |
| Rarbeiten/rfrei (%) nach 1. Verfeinerung | 24,3 / 26,5 | 20.4 / 23,8 |
| RMSD Bindungslänge (Å) nach 1. Verfeinerung | 0,01 | 0,01 |
| RMSD Anleihe Winkel (°) nach 1. Verfeinerung | 1.2 | 1,83 |
| Rotamer Ausreißer (%) nach 1. Verfeinerung | 1.07 | 4.29 |
| Ramachandran begünstigt/zulässig/nicht zulässig (%) nach 1. Verfeinerung | 95.93 / 4,07 / 0 | 95.12 / 4,88 / 0 |
Tabelle 1: x-ray Diffraction Sammlung, Verfeinerung und Validierung Statistik. Werte für die höchste Auflösung Shell sind in Klammern angegeben.
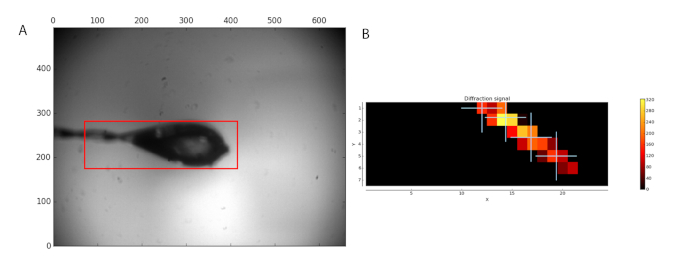
Abbildung 1: Analyse vor der Datenerhebung zu probieren. (A) der Region ausgewählt für Scannen von einem roten Feld angezeigt wird. (B) ist die Analyse der Beugung Bilder als eine Heatmap gezeigt. Vier Positionen innerhalb der befindet sich Kristall wurden für die Datenerhebung ausgewählt. Bitte klicken Sie hier für eine größere Version dieser Figur.
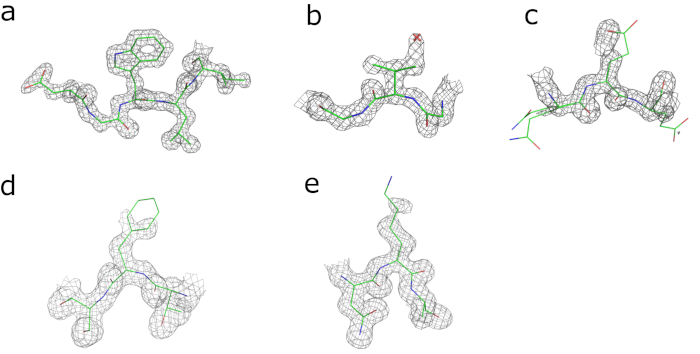
Abbildung 2: visuelle Validierung von Elektron-Dichte-Maps nach Raffinesse erhalten. Elektron Dichte Karten 2 x r.m.s Ebene um (ein) Trp143, (b) Val66 (Ile in menschlichen GCSH) und (c) Glu98 (Asp in menschlichen GCSH) und Karten konturiert konturiert, bei 1 x r.m.s Ebene um (d), Phe149 (Leu in menschlichen GCSH) und (e) Lys125 (Asp in menschlichen GCSH). Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Vollautomatische Beamlines bieten automatisierte Charakterisierung und Datenerfassung von großen Zahlen von makromolekularen Kristalle ohne die Anwesenheit eines Wissenschaftlers, entweder auf das Strahlrohr oder aus der Ferne, erforderlich. Mit vollständig automatisierten Beamlines hat viele Vorteile im Vergleich zum manuellen Betrieb. Für Beispiel, die automatisierte Probe zentrieren, auf x-ray Netz basierend und Linie scannt, ist genauer als das mit dem menschlichen Auge als wird es nicht durch thermische oder optische Effekte beeinflusst. In der Tat bieten diese Masche und Linie Scans Zusatzdaten (z.B. detaillierte Abmessungen des Kristalls und die besten Interferenzgitters Region des Kristalls) sind wichtig bei der Bestimmung der richtigen Strahlgröße zur Datenerfassung nutzen – vor allem für kleine Kristalle 18– und oft eine bessere Qualität der erhaltenen Beugung Daten führen. Darüber hinaus können unter Ausnutzung der benutzerdefinierten Parameter in der Einrichtung von automatischen Experimenten die Schritte in spezifischen Arbeitsabläufe zugeschnitten werden am besten das System untersucht, damit die Erfolgsrate Experiment weiter zu optimieren.
Nimmt man zusammen, die Zuverlässigkeit der Workflows zur Verfügung, der einfache Zugriff auf das Strahlrohr (Benutzer selbst planen, unter Verwendung eines Kalenders [siehe oben]) und der voll automatisierten Ansatz von massiv-1 bietet einen strengen, hohem Durchsatz und zeitsparende Alternative zu klassischen MX experimentieren und das Potenzial, fortgeschrittene Verfahren und Anwendungen in die automatischen Abläufe zu implementieren. In naher Zukunft wird Kristall Kartographie in 3D19 implementiert werden, um die Genauigkeit der Röntgen-Zentrierung, während komplexere Protokolle wie Kristall Dehydrierung Experimente20, automatisiert werden. Es ist zu hoffen, dass völlig autonom Datenerhebung eine Standardmethode in MX, klein-Molekül Fragment Bildschirme, qualitativ hochwertige Daten zur Optimierung der Vorführung einer großen Anzahl von schlecht Interferenzgitters Kristalle und automatisch die Phase werden Informationen zu Crystal Strukturen de Novo zu lösen. In Kombination mit den Entwicklungen in der automatisierten Ernte von Kristallen21könnte die Möglichkeit der Proteinlösung Kristall-Struktur als einen automatisierten Service nun Wirklichkeit geworden.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Die Autoren danken der ESRF für Strahlzeit.
Materialien
| Name | Company | Catalog Number | Comments |
| Beamline MASSIF-1 | ESRF | ||
| BL21DE3 | New England Biolabs | C2527I | |
| chloramphenicol | Roth | 3886.1 | |
| Concentrators: Amicon Ultra-4 Ultracel -30K | Merck Millipore | UFC803024 | |
| Dialyzing membrane | Spectrumlabs | 132655 | |
| DMSO | Sigma-Aldrich | D8418 | |
| Dnase | Roche | 11284932001 | |
| DTT | Euromedex | EU0006-B | |
| EDTA- free protease inhibitors | Roche | 4,693,159,001 | |
| glycerol | VWR Chemicals Prolabo | 14388.29T | |
| His-trap HP | GE healthcare | 17-5247-01 | |
| imidazole | Sigma-Aldrich | 56750-500G | |
| IPTG | Euromedex | EU0008-B | |
| LB medium | Sigma-Aldrich | L3022 | |
| lipoic acid | Sigma-Aldrich | T5625 | |
| loop | Hampton Research | HR8-124 | |
| lysozyme | Roche | 10 837 059 001 | |
| MonoQ 5/50 GL | GE healthcare | 17-5166-01 | |
| NaCl | Fisher Chemical | S/3160/60 | |
| Sonicator vibra cell 75/15 | SONICS | ||
| SPINE pucks | MiTeGen | SKU: M-CSM003-0001A | |
| Tris base | Euromedex | 26-128-3094-B | |
| Sodium Formate | Sigma-Aldrich | 1064430500 | |
| GCSH purification buffer | 20 mM TRIS pH 8, 200 mM NaCl | ||
| GCSH cryo-protection buffer | 0.25 M Sodium Formate pH 4, 30% glycerol | ||
| Programs: | |||
| MxCube | Gabadinho, J. et al. MxCuBE : a synchrotron beamline control environment customized for macromolecular crystallography experiments. Journal of Synchrotron Radiation. 17 (5), 700-707, doi: 10.1107/S0909049510020005 (2010) | local development | |
| ISPyB | ESRF | Solange Delagenière, Patrice Brenchereau, Ludovic Launer, Alun W. Ashton, Ricardo Leal, Stéphanie Veyrier, José Gabadinho, Elspeth J. Gordon, Samuel D. Jones, Karl Erik Levik, Seán M. McSweeney, Stéphanie Monaco, Max Nanao, Darren Spruce, Olof Svensson, Martin A. Walsh, Gordon A. Leonard; ISPyB: an information management system for synchrotron macromolecular crystallography, Bioinformatics, Volume 27, Issue 22, 15 November 2011, Pages 3186-3192, https://doi-org.remotexs.ntu.edu.sg/10.1093/bioinformatics/btr535 | local development |
| MXCube2 | ESRF | Gabadinho, J. et al. MxCuBE : a synchrotron beamline control environment customized for macromolecular crystallography experiments. Journal of Synchrotron Radiation. 17 (5), 700-707, doi: 10.1107/S0909049510020005 (2010). De Santis, D., Leonard, G. Notiziario Neutroni e Luce di Sincrotrone,Consiglio Nazionale delle Ricerche. (19), 24-226 (2014). | local development |
| BES workflow server | Brockhauser, S. et al. The use of workflows in the design and implementation of complex experiments in macromolecular crystallography. Acta Crystallographica Section D Biological Crystallography. 68 (8), 975-984, doi: 10.1107/S090744491201863X (2012). | ||
| DOZOR | ESRF | Bourenkov and Popov, unpublished | local development |
| BLISS beamline control | Guijarro, M. et al. BLISS - Experiments Control for ESRF EBS Beamlines. Proceedings of the 16th Int. Conf. on Accelerator and Large Experimental Control Systems, ICALEPCS2017, Barcelona, Spain. doi: 10.18429/jacow-icalepcs2017-webpl05 (2018). | local development | |
| AUTO processing of images | Monaco, S. et al. Automatic processing of macromolecular crystallography X-ray diffraction data at the ESRF. Journal of Applied Crystallography. 46 (3), 804-810, doi: 10.1107/S0021889813006195 (2013) | local development | |
| BEST and EDNA | Incardona, M.-F., Bourenkov, G.P., Levik, K., Pieritz, R.A., Popov, A.N., Svensson, O. EDNA : a framework for plugin-based applications applied to X-ray experiment online data analysis. Journal of Synchrotron Radiation. 16 (6), 872-879, doi: 10.1107/S0909049509036681 (2009). | local development | |
| CCP4 | Winn, M.D. et al. Overview of the CCP 4 suite and current developments. Acta Crystallographica Section D Biological Crystallography. 67 (4), 235-242, doi: 10.1107/S0907444910045749 (2011). | ||
| Phaser MR | McCoy, A.J., Grosse-Kunstleve, R.W., Adams, P.D., Winn, M.D., Storoni, L.C., Read, R.J. Phaser crystallographic software. Journal of Applied Crystallography. 40 (4), 658-674, doi: 10.1107/S0021889807021206 (2007). | ||
| Coot | Emsley, P., Cowtan, K. Coot: model-building tools for molecular graphics. Acta Crystallogr D Biol Crystallogr. 60, 2126-32 (2004). | ||
| refmac5 | Murshudov, G.N., Vagin, A.A., Dodson, E.J. Refinement of Macromolecular Structures by the Maximum-Likelihood Method. Acta Crystallographica Section D. 53, 240--255 (1997). | ||
| Matthews | Matthews, B.W. Solvent content of protein crystals. Journal of Molecular Biology. 33 (2), 491-497 (1968). |
Referenzen
- Hui, R., Edwards, A. High-throughput protein crystallization. Journal of Structural Biology. 142 (1), 154-161 (2003).
- Bowler, M. W., et al. MASSIF-1: a beamline dedicated to the fully automatic characterization and data collection from crystals of biological macromolecules. Journal of Synchrotron Radiation. 22 (6), 1540-1547 (2015).
- Svensson, O., Malbet-Monaco, S., Popov, A., Nurizzo, D., Bowler, M. W. Fully automatic characterization and data collection from crystals of biological macromolecules. Acta Crystallographica Section D Biological Crystallography. 71 (8), 1757-1767 (2015).
- Cipriani, F., et al. CrystalDirect: a new method for automated crystal harvesting based on laser-induced photoablation of thin films. Acta Crystallographica Section D Biological Crystallography. 68, 1393-1399 (2012).
- Nurizzo, D., et al. RoboDiff: combining a sample changer and goniometer for highly automated macromolecular crystallography experiments. Acta Crystallographica Section D Structural Biology. 72 (8), 966-975 (2016).
- Bowler, M. W., Svensson, O., Nurizzo, D. Fully automatic macromolecular crystallography: the impact of MASSIF-1 on the optimum acquisition and quality of data. Crystallography Reviews. 22 (4), 233-249 (2016).
- Brockhauser, S., et al. The use of workflows in the design and implementation of complex experiments in macromolecular crystallography. Acta Crystallographica Section D Biological Crystallography. 68 (8), 975-984 (2012).
- Svensson, O., Gilski, M., Nurizzo, D., Bowler, M. W. Multi-position data collection and dynamic beam sizing: recent improvements to the automatic data-collection algorithms on MASSIF-1. Acta Crystallographica Section D Structural Biology. 74, 433-440 (2018).
- Cipriani, F., et al. Automation of sample mounting for macromolecular crystallography. Acta Crystallographica Section D Biological Crystallography. 62 (10), 1251-1259 (2006).
- Delageniere, S., et al. ISPyB: an information management system for synchrotron macromolecular crystallography. Bioinformatics. 27 (22), 3186-3192 (2011).
- Monaco, S., et al. Automatic processing of macromolecular crystallography X-ray diffraction data at the ESRF. Journal of Applied Crystallography. 46 (3), 804-810 (2013).
- Kikuchi, G., Motokawa, Y., Yoshida, T., Hiraga, K. Glycine cleavage system: reaction mechanism, physiological significance, and hyperglycinemia. Proceedings of the Japan Academy. Series B, Physical and Biological Sciences. 84 (7), 246-263 (2008).
- Sparta, K. M., Krug, M., Heinemann, U., Mueller, U., Weiss, M. S. XDSAPP2.0. Journal of Applied Crystallography. 49 (3), 1085-1092 (2016).
- Vonrhein, C., et al. Data processing and analysis with the autoPROC toolbox. Acta Crystallographica Section D Biological Crystallography. 67 (4), 293-302 (2011).
- Winter, G. xia2: an expert system for macromolecular crystallography data reduction. Journal of Applied Crystallography. 43 (1), 186-190 (2010).
- Higashiura, A., et al. High-resolution X-ray crystal structure of bovine H-protein at 0.88 Å resolution. Acta Crystallographica Section D Biological Crystallography. 66 (6), 698-708 (2010).
- Sanishvili, R., et al. A 7 µm mini-beam improves diffraction data from small or imperfect crystals of macromolecules. Acta Crystallographica Section D Biological Crystallography. 64 (4), 425-435 (2008).
- Bowler, M. W., et al. Diffraction cartography: applying microbeams to macromolecular crystallography sample evaluation and data collection. Acta Crystallographica Section D Biological Crystallography. 66 (8), 855-864 (2010).
- Bowler, M. W., et al. Automation and Experience of Controlled Crystal Dehydration: Results from the European Synchrotron HC1 Collaboration. Crystal Growth & Design. 15 (3), 1043-1054 (2015).
- Zander, U., et al. Automated harvesting and processing of protein crystals through laser photoablation. Acta Crystallographica Section D Structural Biology. 72 (4), 454-466 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten