Method Article
Großtechnische Produktion von rekombinanten RNAs auf einem kreisförmigen Gerüst mit einem Viroid-abgeleitete System bei Escherichia coli
In diesem Artikel
Zusammenfassung
Hier präsentieren wir ein Protokoll, um produzieren große Mengen an rekombinanten RNA in Escherichia coli Co zum Ausdruck bringen, eine Chimäre RNA, die die RNA in ein Viroid-Gerüst und eine Pflanze enthält tRNA-Ligase. Das Hauptprodukt ist ein kreisförmigen Molekül, das erleichtert die Reinigung zur Homogenität.
Zusammenfassung
Mit zunehmendem Interesse an RNA Biology und die Verwendung von RNA-Molekülen in anspruchsvolle biotechnologische Anwendungen, sind die Methoden, um große Mengen von rekombinanten RNAs produzieren beschränkt. Hier beschreiben wir ein Protokoll, um produzieren große Mengen an rekombinanten RNA in Escherichia coli basierend auf Co Ausdruck eines Chimären Moleküls, die die RNA in ein Viroid-Gerüst und eine Pflanze enthält tRNA-Ligase. Viroide sind relativ klein, nicht-kodierenden, hoch Basis gepaart kreisförmige RNAs, die ansteckend für höhere Pflanzen sind. Der Host Pflanzen tRNA Ligase ist ein Enzym rekrutiert durch Viroide, die zu der Familie Avsunviroidae, wie z. B. Auberginen latente Viroid (ELVd) gehören, um RNA-Circularization während der Viroid-Replikation zu vermitteln. Obwohl ELVd nicht in E. Coli repliziert, eine ELVd Vorläufer ist effizient durch die E. Coli -RNA-Polymerase transkribiert und verarbeitet durch die eingebetteten Hammerhai Ribozyme in Bakterienzellen und die daraus resultierende Monomere durch circularized sind die Co zum Ausdruck gebrachten tRNA-Ligase eine bemerkenswerte Konzentration zu erreichen. Das Einfügen einer RNA von Interesse in der ELVd Gerüst ermöglicht die Produktion von zehn Milligramm des rekombinanten RNA pro Liter Bakterienkultur unter normalen Laborbedingungen. Eine Hauptfraktion des RNA-Produkts ist kreisförmig, ein Feature, das erleichtert die Reinigung der rekombinanten RNA zur virtuellen Homogenität. In diesem Protokoll wird die eine komplementäre DNA (cDNA) entsprechend der RNS des Interesses an einer bestimmten Position ELVd cDNA in ein Expressionsplasmid eingefügt, die entlang das Plasmid Aubergine tRNA Ligase Co Ausdrücken verwendet wird, um E. Colizu verwandeln. Co Ausdruck der beiden Moleküle unter der Kontrolle der starken konstitutiven Promoter führt zur Produktion von großen Mengen der rekombinanten RNA. Rekombinante RNA kann der bakteriellen Zellen entnommen und getrennt von der Masse der bakteriellen RNAs unter Ausnutzung seiner Zirkularität.
Einleitung
Im Gegensatz zu DNA und Proteine sind Protokolle für einfache, effiziente und kostengünstige Produktion von großen Mengen an RNA nicht reichlich vorhanden. Jedoch anspruchsvolle Forschung und Industrie verlangen immer mehr diese Biomoleküle ihre einzigartigen biologischen Eigenschaften1zu untersuchen oder eingesetzt werden biotechnologische Anwendungen, einschließlich ihrer Verwendung als hochspezifische Aptameren 2, Therapeutika3oder selektive Schädlingsbekämpfungsmittel4. In vitro Transkription und chemische Synthese werden häufig in der Forschung verwendet, um RNA zu produzieren. Jedoch führen diese Methoden wichtige Einschränkungen, wenn große Mengen der Produkte erforderlich sind. Die logische Alternative ist mit den endogenen Transkriptionsmaschinerie lebender Zellen, gefolgt von einen Reinigungsprozess, der RNAs von Interesse von den zellulären Begleitern zu trennen. Nach dieser Strategie wurden Methoden entwickelt, um rekombinante RNAs in Bakterienzellen, wie z. B. der Lab-freundlich zu produzieren Escherichia coli5 oder die marine lila phototrophe Alpha-Proteobakterium Rhodovolum Sulfidophilum 6. rekombinanter RNA in Bakterien produzieren die meisten Methoden setzen auf die Expression von native hochstabile RNA leski, wie z. B. eine tRNA oder einer rRNA, in denen die RNS von Interesse ist7eingefügt. Damit steigen die Notwendigkeit des Freigebens der RNS von Interesse aus der Chimären Moleküls, wenn das Vorhandensein von zusätzlichen RNA ein Problem für die downstream-Anwendungen-8 ist. Ein weiteres Konzept in rekombinanter RNA Biotechnologie ist die Produktion von rekombinanten Ribonucleoprotein-komplexe, die können das gewünschte Produkt per se oder verwendet als schützende Strategie zur Erhöhung der Stabilität der RNA des Interesse9, 10. Ebenso wurde die Produktion von kreisförmigen RNAs auch als Strategie zur stabileren Produkte11generieren vorgeschlagen.
Vor kurzem haben wir eine neue Methode, um große Mengen von rekombinanten RNA in E. Coli zu produzieren, die in drei der oben genannten Konzepte beteiligt ist: das Einfügen von der RNA von Interesse in eine hochstabile zirkuläre RNA-Gerüst und Co Ausdruck der rekombinante RNA mit einer interagierenden Proteins, wahrscheinlich eine stabile Ribonucleoprotein Komplex zu produzieren, die sammelt sich in beachtlichen Mengen in bakterielle Zellen12. Im Gegensatz zu den bisherigen Entwicklungen verwendeten wir eine RNA-Gerüst E. Coli, nämlich ein Viroid völlig fremd. Viroide sind eine ganz besondere Art von Infektionserregern von höheren Pflanzen, die ausschließlich durch eine relativ kleine (246-401 nt) konstituiert sind höchst Basis gepaart kreisförmige RNA13. Interessanterweise Viroide sind nicht-kodierende RNAs und ohne Hilfe von ihren eigenen Proteine können sie komplexe infektiöse Zyklen in den infizierten Hosts14abschließen. Diese Zyklen sind die RNA-RNA-Replikation in die Zellkerne oder Chloroplasten, abhängig von der Viroid-Familie –Pospiviroidae oder Avsunviroidae, bzw. – Bewegung durch die infizierten Pflanze und Umgehung der defensive Wirtsantwort. Viroide müssen unter der stabilste RNAs in der Natur, als Folge eine nackte Runde RNA und Überleben in der feindlichen Umgebung von Pflanzenzellen infiziert eingestuft werden. Diese Eigenschaft kann Viroide als Gerüste, rekombinante RNA in biotechnologischen Ansätzen zu stabilisieren besonders geeignet machen. Darüber hinaus basiert die neue Methode auf Co Ausdruck die Viroid-Gerüst mit einer interagierenden pflanzlichem Eiweiß. Viroide replizieren durch einen rolling-Circle-Mechanismus in welchem, den Host Enzyme rekrutiert werden, um die verschiedenen Schritte des Prozesses zu katalysieren. Insbesondere einige Viroide, genauer gesagt diejenigen, die zu der Familie Avsunviroidae15, gehören enthalten Ribozyme, die auch bei der Replikation beteiligt sind. Je nach Tierart Viroid wird vom Host RNA-Polymerase II oder die chloroplastic-nuklearen codiert-RNA-Polymerase (NEP) Transkription von Viroid-RNAs vermittelt. Viroid-RNA Verarbeitung scheint von einem Host katalysiert werden Typ-III-RNase, obwohl in Viroide mit Ribozyme Oligomere RNA Zwischenprodukte während der Replikation selbst Spalten. Schließlich sind die daraus resultierenden Viroid-Monomere, abhängig von der Viroid-Familie durch die Host-DNA-Ligase 1 oder chloroplastic Isoform der tRNA-Ligase16,17circularized. Dieses letzte Enzym ist Ligatur der Monomeren Formen der Viroide in der Familie Avsunviroidae, wie z. B. Auberginen latente Viroid (ELVd)18beteiligt.
Im Zuge eines Werkes, die Sequenz und strukturellen Anforderungen der ELVd zu analysieren, die Anerkennung durch die Aubergine (Solanum Melongena L.) bestimmen tRNA-Ligase, wir richten Sie ein experimentelles System basierend auf Co Ausdruck der beiden Moleküle in E. Coli 19. Wir haben festgestellt, dass länger als Einheit ELVd Protokolle effizient in E. Coli Zellen durch die eingebetteten Hammerhai Ribozyme selbst zu Spalten und den daraus resultierenden Viroid Monomeren mit 5'-Hydroxyl und 2', 3'-Phosphodiester-Termini wurden effizient circularized durch die Zusammenarbeit zum Ausdruck gebrachten Aubergine tRNA-Ligase. Noch mehr erreicht die resultierende kreisförmige Viroid-RNA eine unerwartete hohe Konzentration in E. Coli, über denen der endogenen rRNAs12. Abwesenheit von Replikation Zwischenprodukten angegeben Mangel ELVd RNA-RNA Amplifikation in diesen bakteriellen Zellen. Interessanterweise hatten die Einfügung der heterologen RNAs in einer bestimmten Position des Moleküls Viroid eine moderate Wirkung auf Anhäufung der kreisförmigen Viroid-abgeleitete RNAs12. Diese Beobachtungen machte uns vorstellen, eine Methode, um große Mengen von rekombinanten RNAs in Bakterien produzieren. Bei dieser Methode die cDNAs entspricht der RNAs von Interesse sind in der ELVd cDNA eingefügt und die daraus resultierende Chimäre RNA drückt sich in E. Coli durch einen starken konstitutiven Promoter. Damit das System funktioniert muss E. Coli mit einem Plasmid, die Auberginen tRNA-Ligase auszudrücken Co umgewandelt werden. ELVd abgeleitet länger als Einheit Transkript wird durch die eingebetteten Hammerhai Ribozyme verarbeitet und die daraus resultierende Monomere mit den entsprechenden Termini werden erkannt und circularized durch die Zusammenarbeit zum Ausdruck gebrachten tRNA-Ligase. Auf diese Weise wird die RNA von Interesse in eine sehr stabil kreisförmige Gerüst bestehend aus das Viroid kreisförmigen Molekül eingefügt. Diese rekombinanten Chimären RNA ist höchstwahrscheinlich weiter innerhalb der E. Coli -Zellen durch Bildung von ein Ribonucleoprotein durch Interaktion mit tRNA-Ligase Komplex stabilisiert. Mit dieser Methode (siehe Protokoll unten), RNA Aptameren, Haarnadel RNAs und andere strukturierte RNAs leicht in Mengen von 10 Milligramm pro Liter des E. Coli -Kultur unter normalen Laborbedingungen hergestellt und zur Einnahme von Homogenität gereinigt Vorteil der Zirkularität12.
Protokoll
(1) Plasmid Bau
-
Durch PCR (oder durch reverse Transkription PCR, wenn ausgehend von einer RNA-Vorlage) verstärken die cDNA entspricht der RNS mit Primer mit 5' Extension zum Einbau in das Expressionsplasmid zu ermöglichen. Verwenden Sie zur Vermeidung von unerwünschten Mutationen ein High-Fidelity-DNA-Polymerase.
- Die folgenden 5' Erweiterungen hinzufügen um die cDNA von Gibson Montage20in das Expressionsplasmid einfügen, die PCR-Primer: vorwärts, 5'-TctccccctcccaggtactatccccttXXXXXXXXXXXXXXXXXXXXXX-3 "; Revers, 5'-CcctcctagggaacacatccttgaXXXXXXXXXXXXXXXXXXXXXX-3 "; X steht für Nukleotide homologe zu den terminal Enden der RNA von Interesse.
- Inkubieren Sie für 30 s bei 98 ° C, gefolgt von 30 Zyklen von 10 s bei 98 ° C, 30 s bei 55 ° C und 30 s bei 72 ° C und eine letzte Verlängerung von 10 min bei 72 ° C.
- Digest 100 ng des Plasmids pLELVd-BZB mit 10 U Typ IIS Restriktionsenzym Bpi Rohr ich für 1 h bei 37 ° C in einer 20 µL Reaktion in 0,5 mL Puffer G (10 mM Tris-HCl, pH 7,5, 10 mM MgCl2, 50 mM NaCl 0,1 mg/mL BSA).
Hinweis: Alle Plasmide sind auf Anfrage an den entsprechenden Autor. BPI I entspricht Bbs ich. - Trennen Sie die PCR und Verdauung Produkte durch Elektrophorese in einem 1 % Agarose-Gel in TAE Puffer (40 mM Tris, 20 mM Essigsäure, 1 mM EDTA, pH 7,2). Färben Sie das Gel für 15 min durch Schütteln in 200 mL 0,5 µg/mL Interkalation Bromid. Visualisieren Sie die DNA mit Hilfe einer UV-Transilluminator und schneiden Sie der Bänder entsprechend der verstärkten cDNA und der Bpi -verdaut Plasmid (2046 bp) mit einem Skalpellklinge.
Hinweis: BPI Ich Verdauung von pLELVd-BZB auch ein 528 bp-Produkt entspricht der LacZ-blau-weiß-Reporters herausbringt. - Eluieren die DNA aus dem Gel Fragmenten mit Kieselgel Spalten (gel DNA-Recovery-Kit in Table of Materials) und Quantifizierung der DNA-Konzentration durch photometrische Analyse.
- Richten Sie eine Gibson Montage Reaktion unter Verwendung der verstärkten cDNA und das verdaute Plasmid. Verwenden Sie ein 3-fold Molaren Überschuss an Einsatz gegen Vektor20. 1 h bei 50 ° C inkubieren und reinige die Reaktionsprodukte mit einer Silica-Gel-Spalte (DNA-sauber & Konzentrator-Kit in der Table of Materials).
-
Verwenden Sie die gereinigten Produkte der Gibson Montage Reaktion auf Electroporate kompetente E. Coli DH5α Zellen. Mit 1 mm Elektroporation Küvetten, gelten die folgenden Einstellungen: 1.500 V und 5 ms Inkubation für 1 h bei 37 ° C in super optimierte Brühe mit Catabolite Repression (SOC; 20 g/L Tryptone, 5 g/L Hefeextrakt, 0,5 g/L NaCl, 2,5 mM KCl, 10 mM MgCl2 20 mM Glucose, pH 7,0) flüssigen Medium und dann Verbreitung auf Luria-Bertani (LB; 10 g/L Tryptone, 5 g/L Hefeextrakt, 10 g/L NaCl) Agar (1,5 %) Platten mit 50 µg/mL Ampicillin.
- Für entsprechende transformierten E. Coli Kolonien auf dem Bildschirm verteilt Klone, die wahrscheinlich die Einlage, 15 min vor der Beschichtung der elektroporiert Zellen aufgenommen 30 µL von 50 mg/mL 5-bromo-4-chloro-3-indolyl-β-D-galactopyranoside (X-gal) in Dimethylformamid. Platten über Nacht bei 37 ° c inkubieren
- Wählen Sie mehrere weiße Kolonien und wachsen über Nacht bei 37 ° C in Flüssigmedien LB. Reinigen Plasmide mit einem Miniprep kit (siehe Tabelle der Materialien) und ihre Größe durch Elektrophorese in einem 1 % Agarose-Gel in TAE Puffer zu analysieren.
- Wählen Sie das am ehesten rekombinante Plasmid basierend auf elektrophoretische Migration im Vergleich zur pLELVd-BZB-Kontrolle. Bestätigen Sie die Reihenfolge der ausgewählten Plasmid durch Sequenzierung mit Primer 5'-CCTTTTTCAATATTATTGAAGC-3' und 5'-GATGCTCGTCAGGGGGGCGGAG-3', die Flanke der ganze Expressionskassette in pLELVd-BZB.
Hinweis: Denken Sie daran, dass dieses Plasmid enthält ein 528 bp Polylinker mit dem LacZ-Marker, die durch die cDNA von Interesse ersetzt wird. Einschränkung-Mapping kann helfen, rechts rekombinante Plasmide auswählen.
2. RNA-Expression
- Co-Electroporate (siehe 1.6) belasten der ausgewählten E. Coli (E. Coli BL21 oder ein BL21-Derivat) mit dem pLELVd-BZB-Derivat, das die cDNA entspricht der RNS von Interesse und Plasmid p15LtRnlSm Co ausdrücken enthält die Aubergine tRNA-Ligase. Verwenden Sie die E. Coli HT115(DE3), der RNase III21fehlt, RNAs mit langen doppelsträngigen Regionen zum Ausdruck bringen.
Hinweis: Beide die RNA des Interesses und der tRNA-Ligase mRNA durch die E. Coli -RNA-Polymerase transkribiert werden. Keine DE3 Lysogen, ausdrückliche T7-RNA-Polymerase ist in der E.-Coli -Stamm erforderlich, obwohl die Anwesenheit von diesem Lysogen keine schädlichen Auswirkungen hat. - Nach 1 h Inkubation bei 37 ° C im flüssigen Medium SOC Platte elektroporiert Bakterien in solid LB-Medium mit 50 µg/mL Ampicillin und 34 µg/mL Chloramphenicol. Über Nacht bei 37 ° c inkubieren
- Wählen Sie eine Kolonie und impfen eine 1 L verblüfft Erlenmeyerkolben mit 250 mL Flüssigkeit tolle Brühe (TB) Medium (12 g/L Tryptone, 24 g/L Hefe-Extrakt, 0,4 % Glycerin und 0,17 M KH2PO40,72 M K2HPO4), mit 50 µg/mL Ampicillin und 34 µg/mL Chloramphenicol. Inkubation bei 37 ° C mit kräftig schütteln mit 180 Umdrehungen pro Minute (u/min). Ernten Sie Bakterien zwischen 12 und 16 Uhr nach der Inokulation Kultur.
(3) RNA-Extraktion und Reinigung
-
Für analytische Zwecke 2 mL Aliquots der Kultur zu den gewünschten Zeitpunkten und Zentrifugieren bei 14.000 X g für 2 min. überstand verwerfen und Aufschwemmen der Zellen in 50 µL TE-Puffer (10 mM Tris-HCl, pH 8.0 und 1 mM EDTA) durch aufschütteln.
- Fügen Sie ein Volume (50 µL) aus einer Mischung von 1:1 (V/V) von Phenol (mit Wasser gesättigt und equilibriert bei pH 8,0 mit Tris-HCl, pH 8,0) und Chloroform. Brechen Sie die Zellen durch kräftig aufschütteln und trennen Sie die wässrigen und organische Phasen durch Zentrifugation für 5 min bei 14.000 X g.
- Die wässrigen Phasen (oben), die gesamte bakterielle RNA enthält sorgfältig zu erholen.
Hinweis: Vorbereitungen können bei-20 ° C für die spätere Analyse gespeichert werden.
- Gießen Sie für präparative Zwecke Kultur in eine Zentrifuge 250 mL Flasche und Spin-down Zellen bei 14.000 X g für 10 min. verwerfen überstand. Waschen Sie die Zellen mit resuspending in 30 mL Wasser. Übertragen auf ein Zentrifugenröhrchen und spin-down Zellen wieder unter den gleichen Bedingungen.
- Den Überstand verwerfen und Aufschwemmen der Zellen in 10 mL Chromatographie Puffer (50 mM Tris-HCl, pH 6,5, 150 mM NaCl, 0,2 mM EDTA) durch aufschütteln.
Hinweis: Zu diesem Zeitpunkt können die Zellen bei-20 ° C zum Fortsetzen der Reinigung zu einem anderen Zeitpunkt eingefroren werden. - Mit einem frisch oder aufgetaut bakterielle Vorbereitung, brechen Zellen durch Hinzufügen von 1 Volumen (10 mL) von Phenol: Chloroform (siehe Punkt 3.2) und vortexen kräftig.
- Für 10 min bei 12.000 X gZentrifugieren, die wässrige Phase zu erholen und wieder mit 1 Volumen (10 mL) von Chloroform unter den gleichen Bedingungen zu extrahieren.
Hinweis: Die RNA-Vorbereitung kann an dieser Stelle bei-20 ° C gespeichert werden. -
Weiter zu reinigen insgesamt bakterielle RNA durch Anionen-Austausch-Chromatographie. Filtern Sie die RNA-Vorbereitung durch eine 45 µm Spritze Filter und Last auf eine 1 mL Diethylethanolamine (DEAE) Spalte.
- Zur Reinigung von Chromatographie verwenden Sie ein Flüssigchromatographie-System bei einer Durchflussmenge von 1 mL/min. Vor der Probe laden, equilibrate die Spalte mit 10 mL der Chromatographie Puffer (siehe Punkt 3.3 für Komposition).
- Laden Sie die Probe und waschen Sie die Spalte mit 10 mL der Chromatographie Puffer. Eluieren Sie RNA mit 20 mL Elution Buffer (50 mM Tris-HCl, pH 6,5, 1 M NaCl, 0,2 mM EDTA) und sammeln Sie 1 mL Aliquote zu.
Hinweis: RNA elutes schnell in den ersten Sekundenbruchteilen. Die Spalte kann für weitere Reinigungen wiederverwendet werden. Hierzu die Spalte mit 10 mL Wasser zu waschen und Lagerung bei 4 ° C in 20 % Ethanol.
- Da ein Großteil der rekombinanten, die RNA in E. Coli in einer Kreisform ansammelt, kann diese Eigenschaft zur Reinigung auf Homogenität12profitierte. Trennen Sie kreisförmige RNAs durch zweidimensionale Polyacrylamid Gelelektrophorese (2D Seite) nicht Denaturierung und Denaturierung (8 M Harnstoff) Bedingungen12,22kombiniert die linearen Gegenstücke.
(4) RNA-Analyse
- Bereiten Sie eine 5 % Polyacrylamid-Gel (37.5:1 Acrylamyde:N, N'- Methylenebisacrylamide, Massenverhältnis) in TBE-Puffer (89 mM Tris, 89 mM Borsäure, 2 mM EDTA) mit 8 M Harnstoff.
- Mix-20 µL RNA-Zubereitungen mit 1 Volumen (20 µL) von Ladepuffer (98 % Formamid, 10 mM Tris-HCl, pH 8.0, 1 mM EDTA, 0,0025 % Bromophenol Blue und 0,0025 % Xylol Cyanol), 1,5 min bei 95 ° C in einem Heizblock inkubieren und snap Cool auf Eis.
- Laden Sie die Proben in das Polyacrylamid-Gel und laufen Sie die Elektrophorese zu angemessenen Bedingungen je nach Abmessungen (z.B.200 V 140 x 130 x 2 mm-Gele für 2,5 h Laufzeit) Gel. Das Gel für 15 min in 0,5 µg/mL Interkalation Bromid zu beflecken, waschen mit Wasser und RNA unter UV-Licht zu visualisieren.
Ergebnisse
Zur Herstellung rekombinanter RNA in E. Coli mit ELVd abgeleitete System12ist die RNA von Interesse in eine ELVd RNA-Gerüst veredelt. Diese Chimären RNA ist Co ausgedrückt entlang der Aubergine tRNA-Ligase in E. Coli. Nach der Bearbeitung, gespalten und circularized, bildet die Chimäre kreisförmige RNA, aus der RNA des Interesses ragt, wahrscheinlich ein Ribonucleoprotein Komplex mit dem Co ausgedrückt Aubergine Enzym, das bemerkenswerte Konzentration in den bakteriellen Zellen ( erreicht Abbildung 1). Folglich besteht der erste Schritt zur Herstellung rekombinanter RNA mit diesem System des Einsetzens der cDNA entspricht der RNS von Interesse in einer bestimmten Position in der ELVd cDNA, T245-T246 in der ELVd Reihenfolge Variante AJ536613. Plasmid pLELVd-BZB, enthält ein Polylinker auf diesem Lageerkennung mit zwei Bpi ich Websites und ein blau/weiß-Markergen LacZ erleichtert diese Einfügung durch die sehr effiziente Gibson Montage Methode20. Auswahl von transformierten E. Coli -Klone mit die gewünschten rekombinanten Plasmiden wird durch das blau/weiß-Screening des LacZ Marker erleichtert. Plasmide in dem doppelten Bpi ich Verdauung unvollständig war und konnte nicht einfügen wahrscheinlich produzieren blauen Kolonien im Beisein von X-gal zu übernehmen. Abbildung 2 zeigt die elektrophoretische Analyse der verschiedenen rekombinanten Plasmide in denen verschiedenen cDNAs eingefügt wurden. Diese Plasmide zeigen verschiedene Migrationen im Vergleich zu pLELVd-BZB. Beachten Sie, dass pLELVd-BZB enthält den LacZ-Marker (528 bp), wird ersetzt durch die cDNA entspricht der RNS von Interesse. Also, obwohl dies hängt von der Größe des eingefügten cDNA, die rekombinanten Plasmiden in der Regel schneller als die Kontrolle Plasmid (Abbildung 2) migrieren.
Die pLELVd-BZB-Derivate, die die cDNAs entspricht der RNAs Interesse enthalten sind entlang p15LtRnlSm, co-Electroporate E. Coliverwendet. Co transformierte Bakterien werden auf Platten mit Ampicillin und Chloramphenicol ausgewählt. Dann ausgewählt E. Coli Klone werden bei 37 ° C mit starkem rühren in das Reich TB-Medium angebaut. Unter diesem Kulturbedingungen maximiert die Produktion von rekombinanten, die RNA ist12. Das Vorhandensein der rekombinanten RNA in den Bakterien kann leicht durch brechen die Zellen mit einer Mischung aus Phenol und Chloroform in Gegenwart eines Puffers und Analyse der RNS, welche Partitionen im wässrigen Puffer durch Denaturierung Seite überwacht werden. Abbildung 3 zeigen Interkalation Bromid gebeizt Polyacrylamid-Gele, die in der gesamten, die RNAs aus unterschiedlichen E. Coli Klone voneinander getrennt waren. Die Bilder zeigen starke Bands, die leere ELVd entsprechen, und Chimären ELVd Formen in denen verschiedenen RNAs Interesse eingefügt wurden. Interessanterweise finden wir einen großen Bruchteil der rekombinanten RNA als Kreisform. Mit einer Kombination von zwei Seiten unter denaturierenden Bedingungen bei hoher und niedriger Ionenstärke, kann die Kreisförmigkeit der Hauptfraktion des rekombinanten RNA leicht (Abbildung 4) beobachtet werden.
Abhängig von der nachgeschalteten Anwendung total E. Coli RNA Vorbereitung, die die Chimäre ELVd-RNA von Interesse enthält möglicherweise das Ziel des derzeitigen Protokolls. Jedoch im Bedarfsfall kann die RNA-Vorbereitung weiter durch Anionen-Austausch-Chromatographie gereinigt werden. Chromatogramm in Abbildung 5 zeigt die effiziente Speicherung von E. Coli RNA über die Spalte bei geringer Ionenstärke (150 mM NaCl) und anschließende Elution bei hohen Ionenstärke (1 M NaCl). Die meisten der RNA wird gesammelt, in Sekundenbruchteilen 2 und 3. Die rekombinante RNA zur Homogenität zu reinigen, kann Zirkularität genutzt werden. Kreisförmige RNAs werden in Bezug auf ihre linearen Gegenstücke in denaturierenden Bedingungen22verzögert. Diese RNAs können nach der Interkalation Bromid Färbung aus dem Gel eluiert. Die Verwendung von ein solubilizable Polyacrylamid-Gel, wie vernetzt mit N, N'-BIZ (Acryloyl) Cystamine23, erleichtert die Reinigung von großen Mengen von rekombinanten RNAs (Abbildung 6).

Abbildung 1: Schematische Darstellung der Viroid-basiertes System zur Herstellung rekombinanter RNA in e. coli. Die cDNA entspricht der RNS von Interesse (die 98 nt-lange RNA Aptamer Spinat im Schema) in Plasmid pLELVd-BZB eingefügt wird. E. Coli ist Co verwandelt mit der pLELVd-BZB-Derivat und Plasmid p15LtRnlSm Aubergine tRNA-Ligase Co zum Ausdruck zu bringen. In E. coli, ist die Chimäre ELVd Spinat RNA-Abschrift selbst gespalten (roten Pfeilspitzen) durch die Viroid Hammerhai Ribozyme. Das daraus resultierende Monomer ist von Co ausgedrückt tRNA-Ligase erkannt und circularized. Die rekombinante RNA besteht aus eine kreisförmige Viroid Gerüst aus der RNA des Interesses (in grün ragt). Ansammlung von rekombinanten RNA in großen Mengen in E. Coli wahrscheinlich resultiert aus Stabilisierung der Komplex mit tRNA-Ligase Ribonucleoprotein. Neben dem Split ELVd cDNA, Plasmid pLELVd-BZB enthält einen pUC Replikation Ursprung (pUC Ori), einem Ampicillin-Auswahl-gen (AmpR), der E. Coli murinen Lipoprotein (Lpp) Veranstalter und rRNA (RrnC) Terminator, und die LacZ Marker. Plasmid p15LtRnlSm enthält einen p15A Replikation Ursprung (p15A Ori), eine Chloramphenicol-Resistenz-Gen (CmR), die E. Coli Lpp -Promotor und ein Phagen T7 Transkription Terminator, neben der Aubergine tRNA Ligase cDNA. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2: elektrophoretische Analyse der pLELVd-BZB-abgeleitete Ausdruck Plasmide, die von Interesse verschiedener cDNAs enthalten. Plasmide wurden durch Elektrophorese in einem 1 % Agarosegel getrennt, die mit Interkalation Bromid gefärbt wurde. Bahn 1, DNA-Marker mit Größen in Kbp von einige der Komponenten auf der linken Seite; Bahn 2, pLELVd-BZB; Bahn 3, pLELVd-BZB-Derivat, eine leere ELVd auszudrücken; Bahnen 4 bis 7, pLELVd-BZB Derivate der RNA-Aptamer Spinat (Bahn 4), Streptavidin bindende Aptamer (Bahn 5), eine RNA-Haarnadel mit einer zusammenhängenden 42 bp Doppelstrang-Region (Bahn 6) und die 3' GAP-unabhängige Übersetzung Enhancer (CITE) einer Anlage zum Ausdruck bringen Virus (Spur 7). Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Rekombinanter RNA in E. Coli mit dem Viroid-basierte System produziert. Total RNAs aus unterschiedlichen E. Coli Klone wurden durch die Behandlung mit Phenol: Chloroform und Aliquote durch Denaturierung Seite getrennt extrahiert. Gele mit Interkalation Bromid befleckt werden angezeigt. Rekombinante RNAs entstanden in E. Coli (A) BL21(DE3) und (B) HT115(DE3). (A und B) Bahnen 1, RNA-Marker mit Größen im nt auf der linken Seite; Fahrspuren 2, RNA aus E. Coli Klone eine leere ELVd ausdrücken (333 nt). (A) Bahnen 3 und 4, RNAs aus E. Coli Klone eine Chimäre ELVd Ausdrücken bilden mit Aptamer Spinat (98 nt) und Streptavidin bindende Aptamer (42 nt), beziehungsweise. (B) Bahn 3, RNAs aus einem E. Coli -Klon, eine Chimäre ELVd Form, enthält eine 42 bp langen Doppel stranded RNA auszudrücken. Rote Pfeile zeigen auf die kreisförmige rekombinante RNAs. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: Zirkularität der rekombinanten RNA in E. Coli mit dem Viroid-basierte System produziert. Total RNAs aus einem E. Coli -Klon eine Chimäre ELVd zum Ausdruck bringen, die die Aptamer Spinat enthält von 2D Denaturierung Seite zuerst unter hoher und dann unter geringer Ionenstärke getrennt wurden. Die Gele wurden mit Interkalation Bromid gebeizt. Die blauen Pfeile zeigen Richtungen elektrophoretische Migration. Der rote Pfeil zeigt auf den kreisförmigen ELVd-Spinat, die selektiv aus der linearen Gegenstücke der gleichen Größe während der zwei Trennungen verzögert wird. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 5: Chromatographische Reinigung von einem rekombinanten RNA in E. Coli mit dem Viroid-basierte System produziert. Total RNA aus einem E. Coli -Klon, der ein chimärer ELVd-3 produziert "CITE (55 nt) auf eine DEAE-Chromatographie-Spalte, die in Anwesenheit von 150 mM NaCl gewaschen wurde geladen wurden. RNA wurde im Beisein von 1 M NaCl eluiert. Das Chromatogramm zeigt Extinktion bei 254 nm (blaue Linie) und Leitfähigkeit in mS/cm (orange Linie) im Vergleich zu Volumen. Die verschiedenen Schritte der chromatographischen Trennung (Gleichgewichtherstellung, Injektion, Wash, Elution und Spalte Regeneration) sind angegeben. Gesammelte Fraktionen werden ebenfalls angezeigt. Bitte klicken Sie hier für eine größere Version dieser Figur.
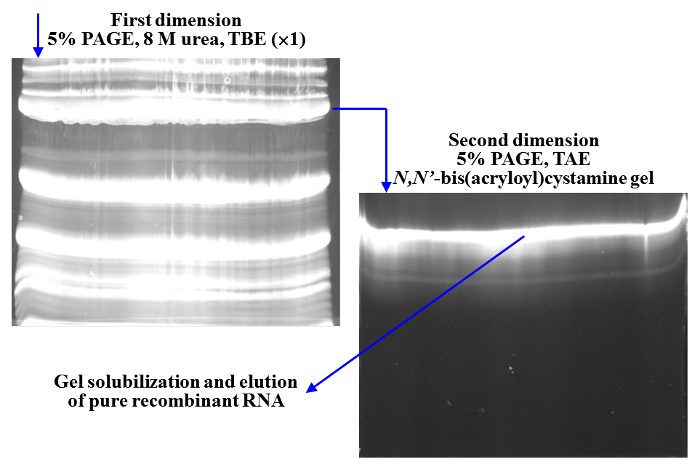
Abbildung 6: Reinigung zur Homogenität des ein rekombinanter RNA in E. Coli mit dem Viroid-basierte System produziert. Total RNA aus einem E. Coli -Klon, der ein chimärer ELVd-3 produziert "CITE wurden zuerst durch Chromatographie mit einem DEAE-Sepharose-Säule gereinigt und dann durch 2D Seite getrennt. Ersten Gel lief unter denaturierenden Bedingungen (8 M Harnstoff, TBE Puffer). Nach der Färbung mit Interkalation Bromid, übertrug die Gel-Band, die die Runde Form der rekombinanten RNA enthält an die Spitze der zweiten Gel, das unter nicht denaturierenden Bedingungen (TAE Puffer) ausgeführt wurde. Die Acrylamid das zweite Gel war vernetzt mit N, N'-BIZ (Acryloyl) Cystamine, die Solubilisierung des Fragments Gel erlaubt, die die reine rekombinante RNA enthalten. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Bei der Recherche die ELVd Sequenz und Strukturanforderungen an die Anerkennung durch die Auberginen tRNA-Ligase beteiligt, bemerkten wir, dass Co Ausdruck der beiden Moleküle in nicht-Host E. Coli führte zu einer unerwarteten großen Anhäufung von Viroid kreisförmige Formen in Bakterienzellen19. Wir verstanden, dass die große Ansammlung von Viroid-RNA in E. Coli sehr wahrscheinlich die Folge Co auszudrücken ein hochstabiles RNA-Molekül, wie die relativ kleinen (333 nt), hoch Basis gepaart, kreisförmige Viroid und ein Protein für die diese bestimmten Viroid zeigt hohen Affinität. ELVd RNA muss der Host tRNA Ligase um seine Circularization während der Replikation in der infizierten Pflanze17vermitteln rekrutieren. Obwohl wir keine experimentelle Bestätigung haben, erwarten wir, dass beide Moleküle ein Ribonucleoprotein Komplex in E. Coli bilden , weiter stabilisiert die Viroid-RNA und ermöglicht die bemerkenswerte Konzentration von 150 mg / l der bakteriellen erreichen Kultur, die stark denen der endogenen RNAs, wie z. B. rRNAs12überschreitet. Da ELVd nicht in E. Colizu replizieren, begründete wir, dass die Einfügung einer heterologen RNA wollte nicht dramatisch beeinflussen Anhäufung der RNA Viroid abgeleitet und in der Tat, das war der Fall. Diese Beobachtung ist die Grundlage der Methode, um große Mengen von rekombinanten RNA in E. Coli zu produzieren, die wir hier beschreiben. Die Methode verwendet die kreisförmige RNS-Molekül des ELVd als ein Gerüst, auf dem die RNA von Interesse vorliegt. Um einen kreisförmigen ELVd Gerüst in E. Colizu produzieren, müssen wir eine Vorstufe RNA mit der doppelten Domäne die Viroid-Hammerhead-Ribozym ausdrücken. ELVd Ribozyme cleave selbst sehr effizient in E. Coli und die daraus resultierenden Viroid Monomeren werden erkannt und circularized durch die Zusammenarbeit zum Ausdruck gebrachten tRNA-Ligase. Co Ausdruck der tRNA-Ligase ist ein wesentliches Merkmal des Systems. ELVd RNA ist kaum in E. Coli entdeckt, sei dieses bestimmte Enzym Co ausgedrückt12.
In unserem Protokoll ermöglicht Plasmid pLELVd-BZB, die cDNA entspricht der RNS des Interesses durch einfache und effiziente Gibson Montage zwischen den Positionen U245 und U246 der ELVd einzufügen. Diese Seite entspricht der Kehrschleife eine lange Haarnadel in der am ehesten ELVd Konformation24,25vorhanden. Eingliederung in diese besondere Lage muss fördern, die die RNA von Interesse aus dem sehr stabilen Gerüst durch ELVd gebildet ragt und, intramolekulare Wechselwirkung zwischen der RNA von Interesse und ELVd (Abbildung 1 vermeiden muss). Wir haben nicht die Wirkung des Einsetzens der RNS des Interesses an alternativen Positionen des Moleküls ELVd erforscht. Plasmid p15LtRnlSm ermöglicht Co Ausdruck der Aubergine tRNA-Ligase. RNAs sind in beiden Plasmide unter der Kontrolle von einem konstitutiven starke E. Coli transkribiert Promotor, nämlich Murein Lipoprotein (Lpp). Dadurch wird das System ohne Induktion konstitutiv. Jedoch bauen wir auch eine ähnliche Plasmid (p15tRnlSm) um die tRNA-Ligase unter der Kontrolle von den Phagen T7-RNA-Polymerase in einem Isopropyl-β-D-1-Thiogalactopyranoside (IPTG) induzierbaren System auszudrücken. Mit Hilfe dieses Plasmid, tritt Anhäufung von rekombinanten RNA nur nach Induktion der tRNA-Ligase Ausdruck durch Zugabe von IPTG12. Im aktuellen System drückt sich die RNA von Interesse aus eine hohe Kopie Nummer Plasmid mit pUC Replikation Herkunft, während die tRNA-Ligase aus eine mäßige Kopie Nummer Plasmid mit p15A Replikation Herkunft zum Ausdruck kommt. Ob die Kopienzahl des Änderung beider Plasmide in das System eingebunden verbessert Anhäufung von rekombinanten, die RNA bisher nicht erforscht worden. Der Größenbereich der RNA des Interesses vom System zugelassen wurde nicht systematisch untersucht, obwohl kleine RNAs sollen logisch zu höheren Konzentrationen als größere ansammeln.
Einer der Gründe, warum die Produktion von rekombinanten RNA in vivo nicht einfach ist, wird wahrscheinlich aufgrund der inneren geringe Halbwertszeit von RNA-Molekülen. Unser System ist nicht dieses Problem fremd. In der Tat beginnt bei einer späten E. Coli wächst Phase, Anhäufung von rekombinanten RNA zu verringern um12praktisch verschwinden. Dies zwingt, im Rechte Fenster bis zur Ernte der Zellen zu finden. Wir beobachten aber, dass dieses Fenster groß genug ist, um das System benutzerfreundlicher zu gestalten. Aber wir auch beobachten, dass das optimale Fenster hängt von vielen Faktoren, einschließlich der besonderen E. Coli Stamm, Kulturmedium, Wachstumsbedingungen (Erregung, Volumen des Kolbens, lüften usw..) und die physiologischen Stadium der Bakterien verwendet, um die Flüssigkultur starten. Wir empfehlen Produktionsbeginn rekombinanter RNA mit frischen E. Coli Kolonien von einer Platte am Vortag inokuliert und bei 37 ° c gewachsen Teller in den Kühlschrank lagern macht die Ernte Fenster unberechenbarer. Wir erhalten die konstantesten Ergebnisse von flüssigen Starterkulturen mit Bakterien aus einer Platte mit einem Zahnstocher abgeholt. Die Verwendung von Pre-Flüssigkultur, fährt besonders wenn gesättigt, auch zu Schwankungen in der Produktionsprotokoll.
Zur Reinigung Behandlung von bakteriellen Zellen mit Phenol: Chloroform ist eine sehr effektive Art, die Zellen zu brechen und ermöglicht die quantitative Erholung total bakterielle RNA in der wässrigen Phase. Abhängig von der nachgeschalteten Anwendung rekombinanter RNA könnte dieser wässrigen Phase oder der Vorbereitung, die durch Fällung der RNS aus dieser wässrigen Phase mit einem Alkohol entsteht genug sein. Wenn weitere Reinigung erforderlich ist, empfiehlt es sich um Anionen-Austausch-Chromatographie mit einer DEAE Anion Austausch Spalte (Abbildung 5). Dieser Reinigungsstufe ermöglicht effiziente Entfernung von DNA und bakteriellen Stoffwechselprodukte, die auch in der wässrigen Phase partitioniert. Wenn die nachgeschaltete Anwendung rekombinanter RNA Reinigung zur Homogenität erfordert, bietet das Viroid-abgeleitete System einen klaren Vorteil im Vergleich zu anderen Methoden. Da eine Hauptfraktion der rekombinanten RNA als eine kreisförmige Form produziert wird, kann diese Eigenschaft genutzt werden, von der Masse der bakteriellen RNAs zu trennen. Kreisförmige RNAs Verzögerung elektrophoretische Migration in denaturierenden Bedingungen in Bezug auf die linearen Gegenstücke der gleichen Größe22. Diese Verzögerung ist stärker ausgeprägt, wenn die RNA unter geringer Ionenstärke electrophoresed ist. In der Praxis, kreisförmige RNAs kann auch getrennt durch zweidimensionale denaturierenden Seite unter hohen und niedrigen Ionischen stärken (Abbildung 4). Nach der elektrophoretischen Trennung muss die rekombinante RNA aus dem Gel eluiert. Die Verwendung von einer reversiblen Vernetzer von Acrylamid, wie z. B. N, N'-BIZ (Acryloyl) Cystamine23, ermöglicht Wiederherstellung, insbesondere dann, wenn große Mengen an RNA (Abbildung 6) zu reinigen. Schließlich verlangt nachgeschalteten Anwendung der erzeugten RNA Trennung der RNA des Interesses vom Viroid Gerüst, bietet unser Protokoll keinen bestimmten Vorteil zu bisherigen Methoden. Die RNA von Interesse muss von Chimären Moleküls mit einigen der zuvor beschriebenen Strategien, wie die Verwendung von Ribozymes, DNAzymes herausgeschnitten werden oder möglicherweise am effizientesten, die Verwendung von RNase H von zwei DNA-Oligonukleotide7geleitet. Versucht, diese letzten Aufbereitungsschritt umständlich zu vermeiden, haben wir gearbeitet, zu versuchen, verkleinern die Viroid-Gerüst, obwohl mit Teilerfolg. Wir waren in der Lage, nennenswerte Mengen an rekombinanter RNA mit Viroid gelöscht Formen zu produzieren (175, 215 und 246 nt) aber zunehmende Löschungen die Viroid-Gerüst mit abnehmender Ansammlung12korreliert.
Zusammenfassend lässt sich sagen wird hier ein Protokoll beschrieben, um große Mengen von rekombinanten RNA in E. Coli zu produzieren, die unseres Wissens, den Ertrag der zuvor veröffentlichten Methoden verbessert. Das Protokoll basiert der Co Interessenbekundung in E. Coli der RNA in leska Viroid (ELVd) und die Auberginen tRNA-Ligase, das Enzym, das diese Viroid in infizierten Pflanzen circularizes eingefügt. Eine Besonderheit unseres Protokolls ist, dass rekombinante produzierte RNA vor allem als eine kreisrunde Form, eine Eigenschaft ist, die Reinigung zur Homogenität für downstream-Anwendungen erleichtert. Mithilfe dieses Protokolls zehn Milligramm rekombinanter RNA pro Liter von E. Coli Kultur unter normalen Laborbedingungen leicht hergestellt werden kann.
Offenlegungen
Die Autoren erklären, dass die in diesem Protokoll beschriebene Technik wurde patentiert (uns Patent No. EP14382177.5, PCT/EP2015/060912).
Danksagungen
Diese Arbeit wurde durch Zuschüsse BIO2017-83184-R und BIO2017-91865-EXP von das spanische Ministerio de Ciencia, Innovación y Universidades (kofinanzierten FEDER Mittel) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Phusion High-Fidelity DNA polymerase | Thermo Scientific | F530S | |
| Bpi I | Thermo Scientific | ER1011 | |
| Agarose | Conda | 8010 | D1 low EEO |
| Tris | PanReac AppliChem | A1086,1000 | |
| Acetic acid | PanReac AppliChem | 131008.1214 | |
| EDTA | Sigma-Aldrich | E5134-500G | |
| Ethidium bromide | PanReac AppliChem | A1152,0025 | 1% |
| Zymoclean Gel DNA Recovery | Zymo Research | D4001 | |
| NanoDrop | ThermoFisher Scientific | ND-3300 | |
| NEBuilder HiFi DNA Assembly Master Mix | New England BioLabs | E2621S | |
| DNA Clean & Concentrator | Zymo Research | D4003 | |
| Eporator | Eppendorf | 4309000019 | |
| Escherichia coli DH5α | Invitrogen | 18265-017 | |
| Tryptone | Intron Biotechnology | Ba2014 | |
| Yeast extract | Intron Biotechnology | 48045 | |
| NaCl | PanReac AppliChem | 131659.1211 | |
| Agar | Intron Biotechnology | 25999 | |
| Ampicillin | PanReac AppliChem | A0839,0010 | |
| X-gal | Duchefa | X1402.1000 | |
| N,N-Dimethylformamide | PanReac AppliChem | 131785.1611 | |
| NucloSpin Plasmid | Macherey-Nagel | 22740588.250 | |
| Escherichia coli BL21(DE3) | Novagen | 69387-3 | |
| Escherichia coli HT115(DE3) | Ref. Timmons et al., 2001 | ||
| Chloramphenicol | Duchefa | C 0113.0025 | |
| Glycerol | PanReac AppliChem | 122329.1211 | 87% |
| KH2PO4 | PanReac AppliChem | 131509.1210 | |
| K2HPO4 | PanReac AppliChem | 122333.1211 | |
| HCl | PanReac AppliChem | 131020.1211 | |
| Phenol | Scharlau | FE04791000 | 90% |
| Chloroform | PanReac AppliChem | A3691,1000 | |
| Filtropur S 0.2 | Sarstedt | 83.1826.001 | |
| HiTrap DEAE Sepharose FF column | GE Healthcare Life Sciences | 17-5055-01 | |
| ÄKTAprime plus liquid chromatography system | GE Healthcare Life Sciences | 11001313 | |
| Acrylamyde | PanReac AppliChem | A1089,1000 | 2K |
| N,N’-methylenebisacrylamide | Sigma-Aldrich | M7279-100G | |
| Urea | PanReac AppliChem | 146392.1211 | |
| Boric acid | PanReac AppliChem | A2940,1000 | |
| Formamide | PanReac AppliChem | A0937,2500 | |
| Bromophenol blue | Sigma-Aldrich | B8026-5G | |
| Xylene cyanol | Sigma-Aldrich | X4126-10G | |
| N,N′-Bis(acryloyl)cystamine | Sigma-Aldrich | A4929-5G |
Referenzen
- Wang, J., et al. Current Research on Non-Coding Ribonucleic Acid (RNA). Genes. 8 (12), (2017).
- Hamada, M. In silico approaches to RNA aptamer design. Biochimie. 145, 8-14 (2018).
- Bobbin, M. L., Rossi, J. J. RNA Interference (RNAi)-Based Therapeutics: Delivering on the Promise? Annual Review of Pharmacology and Toxicology. 56, 103-122 (2016).
- Airs, P. M., Bartholomay, L. C. RNA Interference for Mosquito and Mosquito-Borne Disease Control. Insects. 8 (1), 4(2017).
- Ponchon, L., Dardel, F. Large scale expression and purification of recombinant RNA in Escherichia coli. Methods. 54 (2), 267-273 (2011).
- Kikuchi, Y., Umekage, S. Extracellular nucleic acids of the marine bacterium Rhodovulum sulfidophilum and recombinant RNA production technology using bacteria. FEMS Microbiology Letters. 365 (3), fnx268(2018).
- Ponchon, L., Dardel, F. Recombinant RNA technology: the tRNA scaffold. Nature Methods. 4 (7), 571-576 (2007).
- Batey, R. T. Advances in methods for native expression and purification of RNA for structural studies. Current Opinion in Structural Biology. 26, 1-8 (2014).
- El Khouri, M., et al. Expression and Purification of RNA-Protein Complexes in Escherichia coli. Methods in Molecular Biology. 1316, 25-31 (2015).
- Ponchon, L., et al. Co-expression of RNA-protein complexes in Escherichia coli and applications to RNA biology. Nucleic Acids Research. 41 (15), e150(2013).
- Umekage, S., Kikuchi, Y. In vitro and in vivo production and purification of circular RNA aptamer. Journal of Biotechnology. 139 (4), 265-272 (2009).
- Daròs, J. -A., Aragonés, V., Cordero, T. A viroid-derived system to produce large amounts of recombinant RNA in Escherichia coli. Scientific Reports. 8 (1), 1904(1904).
- Daròs, J. A. Viroids: small noncoding infectious RNAs with the remakable ability of autonomous replication. Current Research Topics in Plant Virology. , Springer International Publishing Switzerland. 295-322 (2016).
- Flores, R., Hernández, C., Martínez de Alba, A. E., Daròs, J. A., Di Serio, F. Viroids and viroid-host interactions. Annual Review of Phytopathology. 43, 117-139 (2005).
- Di Serio, F., et al. ICTV Virus Taxonomy Profile: Avsunviroidae. The Journal of General Virology. 99 (5), 611-612 (2018).
- Nohales, M. A., Flores, R., Daròs, J. A. Viroid RNA redirects host DNA ligase 1 to act as an RNA ligase. Proceedings of the National Academy of Sciences of the United States of America. 109 (34), 13805-13810 (2012).
- Nohales, M. A., Molina-Serrano, D., Flores, R., Daròs, J. A. Involvement of the chloroplastic isoform of tRNA ligase in the replication of viroids belonging to the family Avsunviroidae. Journal of Virology. 86 (15), 8269-8276 (2012).
- Daròs, J. A. Eggplant latent viroid: a friendly experimental system in the family Avsunviroidae. Molecular Plant Pathology. 17 (8), 1170-1177 (2016).
- Cordero, T., Ortolà, B., Daròs, J. A. Mutational Analysis of Eggplant Latent Viroid RNA Circularization by the Eggplant tRNA Ligase in Escherichia coli. Frontiers in Microbiology. 9 (635), (2018).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Timmons, L., Court, D. L., Fire, A. Ingestion of bacterially expressed dsRNAs can produce specific and potent genetic interference in Caenorhabditis elegans. Gene. 263 (1-2), 103-112 (2001).
- Schumacher, J., Randles, J. W., Riesner, D. A two-dimensional electrophoretic technique for the detection of circular viroids and virusoids. Analytical Biochemistry. 135 (2), 288-295 (1983).
- Hansen, J. N., Pheiffer, B. H., Boehnert, J. A. Chemical and electrophoretic properties of solubilizable disulfide gels. Analytical Biochemistry. 105 (1), 192-201 (1980).
- Giguère, T., Adkar-Purushothama, C. R., Bolduc, F., Perreault, J. P. Elucidation of the structures of all members of the Avsunviroidae family. Molecular Plant Pathology. 15 (8), 767-779 (2014).
- López-Carrasco, A., et al. The transcription initiation sites of eggplant latent viroid strands map within distinct motifs in their in vivo RNA conformations. RNA Biology. 13 (1), 83-97 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten