Method Article
Eine neuronale Netzwerk-basierte Identifikation der entwicklungsgeschichtlich zuständigen oder inkompetent Maus ausgewachsene Eizellen
In diesem Artikel
Zusammenfassung
Hier präsentieren wir Ihnen ein Protokoll für die nicht-invasive Beurteilung der Eizelle Entwicklungskompetenz während ihre in Vitro Reifung von Germinal Vesicle zur Metaphase II Stufe durchgeführt. Diese Methode verbindet Zeitraffer Bildgebung mit Particle Image Velocimetry (PIV) und neuronale Netzwerkanalysen.
Zusammenfassung
Die Möglichkeit, entwicklungsgeschichtlich zuständigen vs. inkompetent Eizellen mit nicht-invasiven Verfahren, so verbessert das Gesamtergebnis der Schwangerschaft wählen würde Unfruchtbarkeit Kliniken profitieren. Wir vor kurzem entwickelt eine Klassifizierungsmethode basierend auf mikroskopische live Beobachtungen der Maus Eizellen während ihre in Vitro Reifung von Germinal Vesicle (GV) die Metaphase II Stufe, gefolgt von der Analyse der zytoplasmatischen Bewegungen in diesem Zeitraffer Zeitraum auftreten. Hier präsentieren wir Ihnen ausführliche Protokolle dieses Verfahrens. Eizellen sind von ausgewachsenen antrale Follikel isoliert und kultiviert für 15 h in einem Mikroskop für Zeitraffer-Analyse bei 37 ° C und 5 % CO2ausgestattet. In 8 min Abständen fotografiert. Die Bilder werden analysiert, mit der Particle Image Velocimetry (PIV)-Methode, die für jede Eizelle, das Profil der zytoplasmatischen Bewegung Geschwindigkeiten (CMVs) während der gesamten Kultur vorkommende berechnet. Schließlich werden die CMVs jede einzelne Eizelle durch eine mathematische Klassifizierungs-Tool (Feed-Forward künstliche neuronale Netz, FANN) zugeführt, die Wahrscheinlichkeit, dass ein Gamete entwicklungsgeschichtlich zuständigen oder inkompetent mit einer Genauigkeit von 91,03 % prognostiziert. Dieses Protokoll für die Maus einrichten konnten jetzt auf Eizellen anderer Arten, einschließlich des Menschen getestet werden.
Einleitung
Weiblicher Unfruchtbarkeit ist eine Pathologie, die sich eine steigende Zahl von Frauen auswirkt. Nach Angaben der Weltgesundheitsorganisation sind rund 20 % aller Paare unfruchtbar, mit einer 40 % aufgrund von Unfruchtbarkeit bei Frauen. Darüber hinaus ist ein Drittel der Frauen in der Krebs-Behandlungen (300.000/Jahr und 30.000/Jahr in den USA oder Italien, beziehungsweise) vorzeitige Ovarialinsuffizienz zu entwickeln.
Eine Strategie zur Unfruchtbarkeit bei Krebspatienten zu verhindern ist die Isolation und Kryokonservierung von Eibläschen vor der onkologischen Behandlung, gefolgt von in-vitro- Reifung (IVM) der GV Eizellen auf die MII-Bühne (GV-MII-Übergang). Die Verbesserung der Verfügbarkeit von nicht-invasive Marker der Eizelle Entwicklungskompetenz würde die Befruchtung und Entwicklungsprozesse und die gesamte Schwangerschaft Erfolg1,2.
Anhand ihrer Chromatin-Konfiguration nach der Färbung mit der supravital Fluorochrom Hoechst 33342, beobachtet Säugetier-ausgewachsene Eizellen werden entweder als umgeben Nukleolus (SN) oder eine nicht umgeben Nukleolus (NSN)3eingestuft. Neben ihrer unterschiedlichen Chromatin Organisation zeigen diese beiden Arten von Eizellen viele morphologische und funktionelle Unterschiede3,4,5,6,7,8 ,9, einschließlich ihrer meiotische und entwicklungspolitische Kompetenz. Wenn aus dem Eierstock isoliert und in Vitro gereift, Art von Eizellen Reichweite die MII-Bühne, sowohl nach Sperma Besamung, zu entwickeln, um das 2-Zell-Stadium, aber nur diejenigen mit einer SN Chromatin Organisation können zum Begriff9entwickeln. Obwohl gut wie eine Klassifizierungsmethode für die Auswahl der zuständigen vs. inkompetent Eizellen, ist der Hauptnachteil die mutagene Wirkung, die das Fluorochrom selbst und vor allem das UV-Licht verwendet für seine Entdeckung auf die Zellen haben könnte.
Aus all diesen Gründen suchten wir für andere nicht-invasive Marker, die mit der SN oder NSN Chromatin-Konformation, die die Verwendung von Hoechst unter Beibehaltung der gleichen hohen Klassifizierung Genauigkeit ersetzen könnte. Die Time-Lapse Beobachtung der zytoplasmatischen Bewegung Geschwindigkeiten (CMVs) taucht als ein Merkmal Kennzeichen der Status der Zelle. Zum Beispiel demonstriert neuere Studien, dass die Zuordnung zwischen CMVs zum Zeitpunkt der Befruchtung und die Fähigkeit der Maus und menschlichen Zygoten, komplette Präimplantationsdiagnostik und voll ausgetragenen Entwicklung10,11aufgenommen.
Basierend auf diesen früheren Studien, beschreiben wir hier eine Plattform für die Anerkennung von entwicklungspolitisch zuständigen oder inkompetent Maus ausgewachsene Eizellen5,6,7,8. Die Plattform basiert auf drei wesentlichen Schritten: 1) Oozyten isoliert von antrale Follikel werden zuerst eingestuft anhand ihrer Chromatin-Konfiguration entweder als ein umgeben Nukleolus (SN) oder ein nicht-umgeben Nukleolus (NSN); (2) Zeitraffer Bilder von CMVs, die während des GV-MII Übergangs jede einzelne Eizelle entnommen und analysiert mit Particle Image Velocimetry (PIV); und 3) mit PIV gewonnenen Daten werden mit einem Feed-Forward künstliche neuronale Netzwerk (FANN) für blinde Klassifizierung12,13analysiert. Wir geben die wichtigsten Schritte des Verfahrens für die Maus, um es bereit getestet und verwendet werden für andere Säugetierarten (z. B. Rinder, Affen und Menschen) entwickelt.
Protokoll
Alle Verfahren, bei denen Tiere stimmten die institutionellen Animal Care und Nutzung und ethischen Komitees an Universität von Pavia. Tiere wurden beibehalten, unter 22 ° C, 60 % Luftfeuchtigkeit und ein hell-dunkel-Zyklus von 12:12 Uhr.
1. Eierstock Isolation
- Injizieren Sie intraperitoneal 2 vier bis elf Wochen alten CD1 weibliche Mäuse mit 10 U der Follikel-stimulierendes Hormon mit einer sterilen 1 mL Spritze mit Insulin.
- Warten Sie 46-48 h.
- Wiegen der Maus und mit einer intramuskulären Injektion von 50 mg/kg der Zoletil (Tiletamina und Zolazepan Cloridrate) zu betäuben. Einschläfern Sie durch zervikale Dislokation.
- Erfassen Sie die Haut der Bauchdecke mit einer Dissektion Pinzetten. Schneiden Sie Haut und Körper Wand mit einer Schere und ziehen Sie den Schnitt mit der Pinzette.
- Bewegen Sie mit der Pinzette die Eingeweide beiseite, lokalisieren die uterine Hörner und die Eierstöcke ihre Spitze zu lokalisieren.
- Sanft halten Sie den Eierstock mit Uhrmacher Pinzette und mit einer Schere schneiden Sie ihre Bänder in die Gebärmutter. Wiederholen Sie mit den anderen Eierstock.
- Übertragen Sie die gesammelten Eierstöcke in einer 35-mm-Zelle Kultur Petrischale mit 1 mL M2 Isolierung Medium bei 37 ° C, 5 % CO2 in Luft vorgewärmt.
- Entfernen Sie das Fett und die Eileiter mit einer feinen Schere unter einem Stereomikroskop.
2. Isolierung der Cumulus Eizelle komplexe
- Punktion der Eierstöcken Oberfläche mit sterilen Pinzette und eine sterile Insulin-Nadel 1G bis antral Cumulus Eizelle komplexe (CoC) passiv freigegeben werden.
- Kok mit mehr als drei Zellschichten Cumulus mit einer Mund-gesteuerte Hand zog Pasteur Mikropipette (200 µm im Durchmesser) zu sammeln und in einem 300 µL Tropfen frisches M2 Medium überführen.
- Pipettieren sanft ein-und für einige Sekunden, um die Kumuluszellen rund um die Eizellen mit einer Hand zog Pasteur Mikropipette (80 µm im Durchmesser) zu entfernen,
- Immer wieder übertragen Sie Eizellen aus einem Tropfen 20 µL M2 Medium auf ein anderes, bis Kumuluszellen vollständig beseitigt sind.
(3) Chromatin Organisationsbasis Eizelle Klassifizierung
- Bereiten Sie Hoechst 33342 Färbelösung auf eine Endkonzentration von 0,05 µg/mL in M2 mittlerer ab einer Mutter-Lösung von 5 mg/mL in 1 X PBS verdünnt.
- Platzieren Sie 3,5 µL micro Tröpfchen von Hoechst Färbelösung am unteren Rand ein 35 mm-Petrischale-Deckel.
- Jede einzelne Eizelle in ein Hoechst Färbung Drop übertragen, die Schale auf einer Heizplatte vorgewärmt (37 ° C) und bedecken Sie es mit einem dunklen Deckel, Licht Exposition zu vermeiden.
- Beobachten Sie nach 10 min Inkubation bei Raumtemperatur die Eizellen mit einem Fluoreszenzmikroskop bei 10facher Vergrößerung unter UV-Licht (330-385 nm), für nicht mehr als 1-2 s.
- Klassifizieren Sie Eizellen als ein umgeben Nukleolus (SN) (Abbildung 1A), wenn sie einen Ring von Hoechst-positiven Chromatin die Nukleolus Umgebung präsentieren.
- Klassifizieren Sie Eizellen als eine nicht umgeben Nukleolus (NSN), wenn ihre Nukleolus nicht von Hoechst-positiven Chromatin umgeben ist und zeigt heterochromatischen Flecken innerhalb des Zellkerns (Abbildung 1 b zerstreuen).
(4) neuronale Netzwerk-basierte Eizelle Klassifizierung
- Bereiten Sie vier 2 µL Tröpfchen von α-MEM Medium mit 5 % Hitze-inaktivierten fötalen Rinderserum, 2 mM L-Glutamin, 5 mM Taurin, 100 U/mL Penicillin, 75 µg/mL Streptomycin und 23,5 µg/µL Natrium Pyruvat in ein 35-mm-Glas ergänzt unten Petrischale und bedecken Sie sie mit vorgewärmte (37 ° C) und Pre-äquilibriert Mineralöl, mittlere Verdunstung zu vermeiden.
Hinweis: Wenn eine live-Zelle-Screening-System verwenden, halten Sie die Tropfen in einem bestimmten Abstand mit einem gezeichneten Raster von 7 x 7 mm als Leitlinie. - Transfer 3-4 Eizellen zu jedem Tropfen.
- Legen Sie den Glasboden Petrischale in ein live Cell-Screening-System, ausgestattet mit Zeitraffer-Aufnahme-Software (siehe Tabelle der Materialien), wodurch die Beobachtung der Eizellreifung in einer Umgebung mit stabilen Temperatur (37 ° C) und CO2 Konzentration (5 %).
- Öffnen Sie die Zeitraffer-Aufnahmesoftware. Auf dem Bildschirm erscheint ein Fenster mit eine live Aufnahme von der Eizelle auf der linken Seite und die Einstellung-Tasten auf der rechten Seite.
- Wählen Sie die Mitte der Eizelle in der Bildschirmmitte positionieren. Stellen Sie die Schärfe, wenn nötig.
- Wählen Sie die Schaltfläche " Ph " und legen Sie die DIA-Lampe auf 192; die Belichtungszeit für 1/125 s; der zu gewinnen um 1,99; und die Auflösung auf 1600 x 1200.
- Wählen Sie FL2 und DIA Lampe auf 5gesetzt; die Belichtungszeit , 3 s; die gewinnen zu 2,37; und die Auflösung auf 1600 x 1200.
- Wählen Sie die Schaltfläche " New point " um die Eizelle Position innerhalb der Kultur-Tropfen zu erfassen. Wiederholen Sie die gleiche Routine für jede Eizelle innerhalb der Kultur-Tropfen.
- Wählen Sie Uhrzeit und den Erwerb Zyklus auf 8 min. und die Gesamtzeit bis 15 h.
- Wählen Sie Zeitraffer starten , um die Aufnahme-Zyklus zu starten.
- Öffnen Sie die MATLAB Software und schreiben >>Cell_piv; und wählen Sie dann die PIV-Tool.
- Wählen Sie den Ordner mit den aufgezeichneten Film und .jpeg-Dateien, die gesamte Bildsequenz zu laden. Wählen Sie für jede Eizelle die Region of Interest (ROI) durch Klicken auf den Bereich auswählen.
- Wählen Sie Prozess PIV in das Menü "Datei". Wählen Sie die Schaltfläche " Viewer-Tool ".
Hinweis: Das Programm berechnet die Geschwindigkeitsvektoren innerhalb des ROI und produziert automatisch eine Datendatei als .mat Datei gespeichert. - Wählen Sie die zu öffnende Datei .mat. Wählen Sie die Schaltfläche Wählen Sie ROI-Tool und zeichnen Sie einen Kreis um den Umriss der Eizelle. Klicken Sie im Menü Datei auf speichern.
Hinweis: Die Vektoren in dieser Region werden jetzt angezeigt und die mittlere Größe-Daten im Fenster "Graph" für diese neue ROI aktualisiert. - Offen die gesammelten mittlere Größe-Daten der gesamten Zeitraffer Frames aufgezeichnet in ein Arbeitsblatt, mit, auf die Zeilen, die Bildfolge und an den Säulen, jede einzelne Eizelle analysiert.
- Organisieren Sie die Daten von NSN und SN Eizellen in 10 Untergruppen.
- Verwenden Sie 9 Untergruppen, um das Feedforward künstliche neuronale Netz iterativ zu trainieren (FANN) und 1 Untergruppe blind zu Testzwecken.
- Wiederholen Sie die anderen 9 Mal, ändern die blinde Teststichprobe (10-divisibel Kreuzvalidierung).
- Öffnen Sie MATLAB maßgeschneiderte Skript Haupt und, auf Anfrage, geben Sie den Namen der Datei mit den Trainingsdaten (train.txt), die Anzahl der NSN und SN Eizellen verwendet für Training und das Time-Lapse Intervall (in der Regel von analysiert werden # 1-2 Rahmen, Rahmen # 112-113).
- Drücken Sie, geben Sie das Training durchführen.
- Geben Sie den Namen der Datei mit Testdaten (test.txt).
- Öffnen Sie den Vektor Test_outputs_cyt im MATLAB Workspace.
Hinweis: Es erscheint ein Fenster auf dem Bildschirm zeigt, in der ersten Zeile, die Wahrscheinlichkeit, richtig positive Ergebnisse (TP oder wahre NSN) und False Positives (FP oder falsche NSN) bzw. für NSN und SN Eizellen zu erhalten; stattdessen in der zweiten Reihe, die Wahrscheinlichkeit falsch negative (FN oder falsche SN) und richtig negativ (TN oder wahre SN) für NSN und SN Eizellen, bzw. zu erhalten. - Verwenden Sie die Formel: (TP+TN)/(TP+FP+FN+TN), um die FANN Genauigkeit, ausgedrückt als Prozentsatz zu berechnen.
Ergebnisse
Abbildung 2 zeigt eine repräsentative entwicklungsgeschichtlich kompetente und inkompetente Eizelle bzw. am Anfang (GV) und Ende (MII) das IVM-Verfahren. IVM ausgewachsene Maus Eizellen tritt während 15 h Kultur. Die Time-Lapse Beobachtung zeichnet den Verlauf der Meiose und erkennt meiotische Großveranstaltungen, darunter die GVBD und die Extrusion der erste Polkörper.
Die Analyse und den Vergleich von mehr als zweihundert entwicklungsgeschichtlich zuständigen vs. inkompetent Eizellen zeigten Zeitunterschiede in den Verlauf der Meiose. Insbesondere in NSN Eizellen, GVBD deutlich von 1-2 Zeitraffer Frames, verzögert während die Extrusion PB1 mit einer Verzögerung von 15 Frames erfolgt. Trotz dieser Unterschiede ist die Variabilität zu hoch, um einzelne Eizelle Klassifizierung ermöglichen. Aus diesem Grund wurde das Verfahren mit einer mathematischen Klassifizierungs-Tool realisiert.
Abbildung 3 zeigt ein Schema des Werkzeugs mathematische Klassifikation verwendet, benannte Feedforward künstliche neuronale Netz (FANN). FANN gibt für jede Eizelle durch zugeführt gleichzeitig die Wahrscheinlichkeit, dass die Keimzelle einer entwicklungsgeschichtlich zuständig ist und die Wahrscheinlichkeit, dass es eine inkompetente Eizelle ist. In unseren Experimenten die FANN Maus Eizellen mit eine mittlere Genauigkeit von 91,03 % klassifiziert.
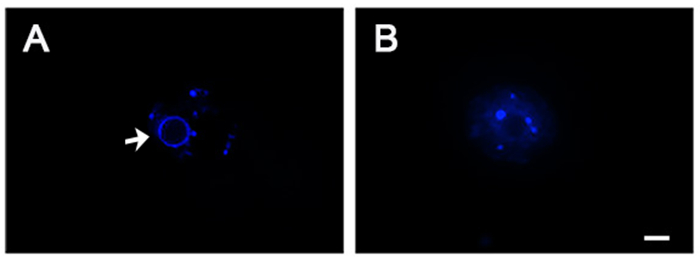
Abbildung 1 . Repräsentative SN und NSN Eizellen gebeizt mit Hoechst 33342. Nach Isolation und Färbung mit Fluorochrom Hoechst 33342, ausgewachsene Eizellen eingestuft werden entweder als umgeben Nukleolus (SN) (A) oder nicht umgeben Nukleolus (NSN) (B), je nach dem Vorhandensein (Pfeil) oder fehlen, bzw. aus einem Ring aus Hoechst-positiven Heterochromatin rund um die Nukleolus. Maßstab: 5 µm Klicken Sie bitte hier, um eine größere Version dieser Figur.

Abbildung 2 . Particle Image Velocimetry Analyse der Zeitraffer Bilder. Die Ergebnisse der PIV-Analyse sehen Sie unten das Hellfeld Bild für jede der 113 Zeitraffer Frames analysiert. Die Abbildung zeigt den Anfang (GV) und Ende (MII) des Brennvorgangs. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3 . Schematische Darstellung des künstlichen neuronalen Netzes Feedforward. FANN ist das mathematische Werkzeug verwendet, um Eizellen als entwicklungsgeschichtlich zuständigen oder inkompetent zu klassifizieren. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Es gibt mehrere wichtige Schritte, die eine kümmern sollte während der Durchführung dieses Protokolls Maus Eizellen sowie mit anderen Arten. Einmal aus den Follikeln isoliert, Eizellen übertragen werden sollte sofort in die Aufnahme sinkt, wie die Trennung von der Begleiter Cumulus löst Zellen Anfang des GV-MII Übergangs. Eine mögliche Änderung dieses Protokolls konnte die Zugabe von 3-Isobutyl-1-Methylxanthin (IBMX) M2 Medium für COCs Isolierung verwendet werden. IBMX verhindert die sofortige Auslösung des GV-MII Übergangs und ermöglicht eine Synchronisierung der gesamte Versuchsgruppe Eizellen.
Das live Cell-Screening-System verwendet in unseren Experimenten (siehe Tabelle der Materialien) begrenzt die Anzahl der Reifung Tropfen, 4 mit 4 Eizellen pro Tropfen, somit auf insgesamt 16 Eizellen pro Versuch. Diese Einschränkung konnte überwunden werden, mit anderen Zeitraffer live-Zelle-Screening-Systeme, kommerziell verfügbar und mit mehreren Kultur Kammern ausgestattet.
Das Aufzeichnungsintervall zwischen einem Frame und den folgenden ist basierend auf unserer Erfahrung, ein kritischer Punkt. Nach einer Reihe von Vorversuchen nahmen wir Bilder alle 8 min weil dies die minimale Zeitfenster war, dass die klare Beobachtung wichtige Ereignisse während des GV-MII-Übergangs, wie die germinal Vescicle Aufteilung und die Extrusion erlaubt der erste Polkörper. Dieses Zeitfenster müssen aufgewertet werden, wenn Sie Eizellen anderer Arten zu beobachten.
Ein Nachteil dieser Methode ist die Kultur der Eizellen in Ermangelung ihrer umgebenden Kumuluszellen, da letztere bekannt sind, um den GV-MII-Übergang weiter zu verbessern. In dieser Hinsicht ist unsere jetzt eine leichte Modifikation des Protokolls von gemeldeten Labortests durch die Einbeziehung der Kultur der Cumulus-freie Eizellen für eine Cumulus Zellen Feeder Schicht.
Durch seine Natur die FANN hat eine feste Anzahl von Input (die Anzahl der analysierten CMV-Module) und Ausgabe (entweder ein entwicklungsgeschichtlich zuständigen oder inkompetent Eizelle) Neuronen, aber die Anzahl der versteckten Neuronen wird gewählt durch den Prüfer durch eine Prüfung und Korrektur Annäherung an die beste Vorhersage Leistung zu erzielen. In unserem Fall Wir experimentierten mit einer unterschiedlichen Anzahl von versteckten Neuronen und festgestellt, dass drei der besten Ergebnisse gab. Außerdem erfordern FANN Analysen eine große Anzahl von Eingabedaten (in unseren Experimenten 112 CMV-Modulen), um eine robuste Statistiken zu erhalten. Eine mögliche Verbesserung, die die Anzahl der notwendigen Eingabedaten reduzieren könnte ist der Einsatz von alternativen Klassifizierung Tools, z. B. Support Vector Machine, Baum-Bag oder KNN Klassifikatoren. Wenn die FANN Analyse seiner Robustheit zeigt, könnten Schritt 1 aus dem Verfahren beseitigt werden.
Dieses Protokoll, eingerichtet für die Maus könnte auf Eizellen anderer Arten, einschließlich des Menschen getestet werden. Darüber hinaus Aufnahme die Kombination von Zeitraffer-mit PIV und neural Network, die Analysen für die Beobachtung von CMVs in Zygoten10,11 und Präimplantationsdiagnostik Embryonen für die Bewertung von ihren weiteren ausgenutzt werden kann Entwicklungspotenzial.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde möglich dank der Unterstützung von: Universität von Pavia BRD 2016; Universität Parma FIL 2014, 2016; und Kinesis für die Versorgung der Kunststoffprodukte notwendig, diese Studie durchzuführen. Wir danken Dr. Shane Windsor (Faculty of Engineering, University of Bristol, UK) für die Bereitstellung der Cell_PIV-Software.
Materialien
| Name | Company | Catalog Number | Comments |
| Folligon | Intervet | A201A02 | Hormonal treatment |
| Hoechst 33342 | Sigma-Aldrich | B2261 | For oocyte heterochromatin staining |
| Cell culture Petri-dish 35 mm x 10 mm | Corning | 430165 | For COCs isolation |
| EmbryoMax M2 Medium (1X), Liquid, with phenol red | Merck-Millipore | MR-015-D | For COCs isolation |
| MEM Alpha medium (1X) + Glutamax | Sigma-Aldrich | M4526 | For oocyte in vitro maturation |
| Cell culture Petri-dish 35 mm glass-bottom | WillCo | GWSt-3522 | For imaging experiments |
| BioStation IM-LM | Nikon | MFA91001 | Live cell screening system |
| Pasteur pipette | Delchimica Scientific Glassware | 6709230 | For follicles manipulation |
| Mineral oil | Sigma-Aldrich | M8410 | To prevent contamination and medium evaporation |
| Penicillin / Streptomycin | Life Technologies | 15070063 | To prevent medium contamination |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | ML16141079 | For making up αMEM medium |
| L-Glutamine | Life Technologies | 25030 | For making up αMEM medium |
| Taurine | Sigma-Aldrich | T0625 | For making up αMEM medium |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A3310 | For making up αMEM media |
| Sodium pyruvate | Sigma-Aldrich | P4562 | For making up αMEM media |
| Zoletil (Tiletamina and Zolazepan cloridrate) | Virbac Srl | QN01AX9 | For mice anesthesia |
| Cell_PIV sofware | Kindly provided by Dr. Shane Windsor, University of Bristol, UK | - | - |
| MATLAB | The MathWorks, Natick, MA | - | For multi-paradigm numerical computing |
Referenzen
- Patrizio, P., Fragouli, E., Bianchi, V., Borini, A., Wells, D. Molecular methods for selection of the ideal oocyte. Reprod. Biomed. Online. 15 (3), 346-353 (2007).
- Rienzi, L., Vajta, G., Ubaldi, F. Predictive value of oocyte morphology in human IVF: a systematic review of the literature. Human. Reprod. Update. 17 (1), 34-35 (2011).
- Tan, J. H., et al. Chromatin configurations in the germinal vesicle of mammalian oocytes. Mol. Hum. Reprod. 15 (1), 1-9 (2009).
- Vigone, G., et al. Transcriptome based identification of mouse cumulus cell markers that predict the developmental competence of their enclosed antral oocytes. BMC Genomics. 14, 380(2013).
- Bui, T. T., et al. Cytoplasmic movement profiles of mouse surrounding nucleolus and not-surrounding nucleolus antral oocytes during meiotic resumption. Mol. Reprod. Dev. 84 (5), 356-362 (2017).
- Zuccotti, M., Piccinelli, A., Giorgi Rossi, P., Garagna, S., Redi, C. A. Chromatin organization during mouse oocyte growth. Mol. Reprod. Dev. 41 (4), 479-485 (1995).
- Zuccotti, M., Garagna, S., Merico, V., Monti, M., Redi, C. A. Chromatin organisation and nuclear architecture in growing mouse oocytes. Mol. Cell. Endocrinol. 234 (1-2), 11-17 (2005).
- Zuccotti, M., Merico, V., Cecconi, S., Redi, C. A., Garagna, S. What does it take to make a developmentally competent mammalian egg? Hum. Reprod. Update. 17 (4), 525-540 (2011).
- Inoue, A., Nakajima, R., Nagata, M., Aoki, F. Contribution of the oocyte nucleus and cytoplasm to the determination of meiotic and developmental competence in mice. Hum. Reprod. 23 (6), 1377-1384 (2008).
- Ajduk, A., et al. Rhythmic actomyosin-driven contractions induced by sperm entry predict mammalian embryo viability. Nat. Commun. 2, 417(2011).
- Swann, K., et al. Phospholipase C-ζ-induced Ca2+ oscillations cause coincident cytoplasmic movements in human oocytes that failed to fertilize after intracytoplasmic sperm injection. Fertil. Steril. 97 (3), 742-747 (2012).
- Thakur, A., Mishra, V., Jain, S. K. Feed forward artificial neural network: tool for early detection of ovarian cancer. Sci. Pharm. 79 (3), 493-505 (2011).
- Laudani, A., Lozito, G. M., Riganti Fulginei, F., Salvini, A. On Training Efficiency and Computational Costs of a Feed Forward Neural Network: A Review. Comput. Intell. Neurosci. 2015, 818243(2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten