Method Article
Immunfluoreszenzanalyse der Stressgranulat-Bildung nach bakterieller Herausforderung von Säugetierzellen
In diesem Artikel
Zusammenfassung
Wir beschreiben eine Methode zur qualitativen und quantitativen Analyse der Stressgranulatbildung in Säugetierzellen, nachdem die Zellen mit Bakterien und einer Anzahl unterschiedlicher Belastungen herausgefordert wurden. Dieses Protokoll kann angewendet werden, um die zelluläre Stressgranulat-Antwort in einem breiten Bereich von Wirts-bakteriellen Wechselwirkungen zu untersuchen.
Zusammenfassung
Die Fluoreszenz-Bildgebung von zellulären Komponenten ist ein wirksames Instrument zur Untersuchung von Wirts-Pathogen-Wechselwirkungen. Pathogene können viele verschiedene Merkmale infizierter Zellen beeinflussen, darunter Organellen-Ultrastruktur, zytoskeletale Netzwerkorganisation sowie zelluläre Prozesse wie Stressgranulat (SG). Die Charakterisierung, wie Pathogene Host-Prozesse untergraben, ist ein wichtiger und integraler Bestandteil des Feldes der Pathogenese. Während variable Phänotypen leicht sichtbar sind, ist die genaue Analyse der qualitativen und quantitativen Unterschiede in den zellulären Strukturen, die durch die pathogene Herausforderung induziert werden, für die Festlegung statistisch signifikanter Unterschiede zwischen experimentellen und Kontrollproben unerlässlich. SG-Bildung ist eine evolutionär konservierte Stressreaktion, die zu antiviralen Reaktionen führt und seit langem mit viralen Infektionen untersucht wird 1 . SG-Bildung wirkt sich auch auf Signalkaskaden aus und kann noch andere noch unbekannte Konsequenzen habenUnces 2 Die Charakterisierung dieser Stressreaktion auf andere Pathogene als Viren, wie bakterielle Pathogene, ist derzeit ein aufstrebendes Forschungsgebiet 3 . Dennoch ist die quantitative und qualitative Analyse der SG-Bildung noch nicht routinemäßig, auch in den viralen Systemen. Hier beschreiben wir ein einfaches Verfahren zur Induktion und Charakterisierung der SG-Bildung in nicht infizierten Zellen und in mit einem zytosolischen Bakterienpathogen infizierten Zellen, die die Bildung von SGs als Reaktion auf verschiedene exogene Spannungen beeinflussen. Die Analyse der SG-Bildung und -Anlage wird durch die Verwendung einer Anzahl von verschiedenen SG-Markern und des Spotdetektor-Plugins von ICY, einem Open-Source-Bildanalyse-Tool, erreicht.
Einleitung
Die Visualisierung von Host-Pathogen-Interaktionen auf zellulärer Ebene ist eine leistungsstarke Methode, um Einblicke in pathogene Strategien zu gewinnen und wichtige Zellwege zu identifizieren. In der Tat können Pathogene als Werkzeuge verwendet werden, um wichtige zelluläre Ziele oder Strukturen zu lokalisieren, da sich Pathogene entwickelt haben, um zentrale zelluläre Prozesse als Strategie für ihr eigenes Überleben oder ihre Ausbreitung zu untergraben. Die Visualisierung von zellulären Komponenten kann durch rekombinante Expression von fluoreszenzmarkierten Wirtsproteinen erreicht werden. Während dies eine Echtzeitanalyse ermöglicht, ist die Erzeugung von Zelllinien mit speziell markierten Wirtsproteinen sehr aufwendig und kann zu unerwünschten Nebenwirkungen führen. Zweckmäßiger ist der Nachweis von zellulären Faktoren, die spezifische Antikörper verwenden, da mehrere Wirtsfaktoren gleichzeitig analysiert werden können und einer nicht auf einen bestimmten Zelltyp beschränkt ist. Ein Nachteil ist, dass nur eine statische Ansicht erfasst werden kann, da die Immunfluoreszenzanalyse die Wirtszell-Fixierung erfordertIon. Ein wichtiger Vorteil der Immunfluoreszenz-Bildgebung ist jedoch, dass sie sich sowohl qualitative als auch quantitative Analysen eignet. Dies wiederum kann verwendet werden, um statistisch signifikante Unterschiede zu erhalten, um neue Einblicke in Host-Pathogen-Wechselwirkungen zu liefern.
Fluoreszierende Bildanalyse-Programme sind leistungsstarke analytische Werkzeuge für die Durchführung von 3D- und 4D-Analysen. Allerdings sind die hohen Kosten der Software und ihre Wartung machen Methoden auf freie Open-Source-Software mehr attraktiv. Eine sorgfältige Bildanalyse mit Bioanalyse-Software ist wertvoll, da sie die visuelle Analyse untermauert und bei der Zuweisung statistischer Signifikanzen das Vertrauen in die Richtigkeit eines gegebenen Phänotyps erhöht. Bisher wurden SGs mit der freien ImageJ Software analysiert, was die manuelle Identifikation einzelner SGs 4 erfordert. Hier stellen wir ein Protokoll für die Induktion und Analyse der zellulären SG-Bildung im Rahmen von bac zur VerfügungTerial-Infektionen mit der freien Open-Source-Bio-Bild-Analyse-Software ICY (http://icy.bioimageanalysis.org). Die Bio-Bildanalyse-Software verfügt über ein eingebautes Spot-Detektor-Programm, das sich hervorragend für die SG-Analyse eignet. Es ermöglicht die Feinabstimmung des automatisierten Erkennungsprozesses in bestimmten Regionen von Interesse (ROIs). Dies überwindet die Notwendigkeit einer manuellen Analyse einzelner SGs und beseitigt die Abtastvorspannung.
Viele Umgebungsbelastungen induzieren die Bildung von SGs, die phasen-dichte zytosolische, nicht-membranöse Strukturen von 0,2 - 5 μm im Durchmesser 5 , 6 sind . Diese zelluläre Antwort ist evolutionär in Hefe, Pflanzen und Säugetieren konserviert und tritt auf, wenn die globale Protein-Translation gehemmt wird. Es handelt sich um die Aggregation von blockierten Translationsinitiationskomplexen in SGs, die als Haltungsplätze für translational-inaktive mRNAs betrachtet werden, was eine selektive Translation einer Untermenge von zellulären mRNAs ermöglicht.Nach der Beseitigung der Belastung, SGs auflösen und globale Preise der Proteinsynthese wieder aufnehmen. SGs bestehen aus Translationsdehnungsinitiationsfaktoren, Proteinen, die an dem RNA-Metabolismus beteiligt sind, RNA-bindende Proteine sowie Gerüstproteine und Faktoren, die an der Wirtszell-Signalisierung 2 beteiligt sind , obwohl die genaue Zusammensetzung in Abhängigkeit von der angewandten Belastung variieren kann. Umwelteinflüsse, die die SG-Bildung induzieren, schließen die Aminosäure-Verhungern, die UV-Bestrahlung, den Hitzeschock, den osmotischen Schock, den endoplasmatischen Retikulumstress, die Hypoxie und die Virusinfektion 2 , 7 , 8 ein . Es wurde viel Fortschritte gemacht, um zu verstehen, wie Viren die SG-Bildung induzieren und auch untergraben, während wenig darüber bekannt ist, wie andere Pathogene wie Bakterien-, Pilz- oder Protozoenpathogene diese zelluläre Stressreaktion 1 , 7 beeinflussen .
ShigeLla flexneri ist ein gram-negatives fakultatives zytosolisches Pathogen des Menschen und der Erreger von schweren Durchfall oder Shigellosis. Shigellosis ist eine große öffentliche Gesundheit Belastung und führt zu 28.000 Todesfälle jährlich bei Kindern unter 5 Jahren im Alter von 9 , 10 . S. flexneri infiziert das Kolon-Epithel und verbreitet Zelle-zu-Zelle durch Entführung der Zytoskelettkomponenten des Wirts 11 , 12 . Die Infektion des Epithels unterstützt die Replikation von S. flexneri innerhalb des Cytosols, aber infizierte Makrophagen sterben durch einen entzündlichen Zelltodprozess namens Pyroptose. Infektion führt zu einer massiven Rekrutierung von Neutrophilen und einer schweren Entzündung, die von Hitze, oxidativem Stress und Gewebezerstörung begleitet wird. Während also infizierte Zellen internen Belastungen durch Infektion induziert werden, wie z. B. Golgi-Störung, genotoxischer Stress und zytoskeletale UmlagerungS, infizierte Zellen sind auch Umweltbelastungen durch den entzündlichen Prozess unterworfen.
Die Charakterisierung der Wirkung von S. flexneri- Infektion auf die Fähigkeit von Zellen, auf Umweltbelastungen mit einer Anzahl von SG-Markern zu reagieren, hat gezeigt, dass die Infektion zu qualitativen und quantitativen Unterschieden in der SG-Zusammensetzung führt 3 . Allerdings ist wenig über andere bakterielle Pathogene bekannt. Hier beschreiben wir eine Methodik für die Infektion von Wirtszellen mit dem zytosolischen Pathogen S. flexneri , die Beanspruchung von Zellen mit unterschiedlichen Umweltbelastungen, die Markierung von SG-Komponenten und die qualitative und quantitative Analyse der SG-Bildung und Zusammensetzung im Kontext von infizierten Und nicht infizierten Zellen. Diese Methode ist weithin anwendbar auf andere bakterielle Pathogene. Zusätzlich kann die Bildanalyse der SG-Bildung für Infektionen durch Viren oder andere Pathogene verwendet werden. Es kann verwendet werden, um SG zu analysierenBildung bei Infektion oder die Wirkung einer Infektion auf die SG-Bildung als Reaktion auf exogene Spannungen.
Protokoll
1. Vorbereitung von Bakterien und Wirtszellen
- Übertragen Sie 12 oder 14 mm Glasdeckel in entweder 24- oder 12-Well-Platten, jeweils mit steriler Pinzette und zählen eine Vertiefung pro experimentellen Zustand. Sterilisieren Sie diese durch UV-Behandlung in einer Gewebekulturhaube für 15 min.
HINWEIS: Kontrollieren Sie die Brunnen für die bakterielle Herausforderung und für die SG-Induktion durch exogene Spannungen. - Für eine 24-Well-Platte mit 12 mm Deckgläsern, Saatgut 1 x 10 5 Säugetierzellen ( zB HeLa-Zellen) auf Deckgläser; Die Deckgläser mit einer sterilen Pipettenspitze nach unten drücken. Die Zellen haften über Nacht bei 37 ° C in einem 5% CO 2 Gewebekulturinkubator in Kulturmedium, das für jeden Zelltyp geeignet ist.
HINWEIS: Tafeln Sie Zellen, so dass der Zusammenfluss der Zellen zwischen 40 - 60% am folgenden Tag ist.- Für HeLa-Zellen inkubieren in Dulbecco's Modified Eagle Medium (DMEM) mit nicht essentiellen Aminosäuren (NEAA) und 10% dekomplementiertem Fetal Calf Serum (FCS).Zersetzung FCS durch Erhitzen für 30 min in einem 56 ° C Wasserbad, Wirbeln des Serums einmal nach 15 min.
- Um Bakterien auszurotten, verwenden Sie eine sterile Pipettenspitze, um eine kleine Menge aus einem bakteriellen Glycerin-Material (20% Glycerin) zu entfernen, der in einem Gefrierschrank von -80 ° C aufbewahrt und auf eine beschriftete Platte gelegt wird. Verwenden Sie eine sterile Schleife, um die Bakterien über etwa 10% der Platte zu verbreiten. Benutze eine neue Sterilschleife und ziehe sie durch den Abstrich, um 3 parallele Linien zu machen, eine halbe Platte lang. Streifen Sie durch diese Linien, die den Rest der Platte bedecken. Inkubation der Platte O / N bei 37 ° C.
- Wählen Sie eine einzelne Bakterienkolonie ( zB S. flexneri ) aus der frisch gestreiften Platte. Vermeiden Sie die Arbeit mit Bakterienkolonien älter als 1 Woche.
- Für S. flexneri- Stamm M90T, eine rote Kolonie aus einer frisch gestreiften Tryptic Soy Agar-0,1% Congo-Rot-Agar-Platte in 8 ml Tryptic Soy-Brühe in einem 15-ml-Röhrchen inokulieren; Ziehen Sie den Deckel fest und inkubieren Sie das Schütteln bei 222 U / min O / N bei 306; C.
ACHTUNG: Verwenden Sie keine großen weißen Kolonien, da sie das Virulenzplasmid verloren haben und nicht infektiös sind.
2. Bakterielle Herausforderung der Wirtszellen
- Bakterien vorbereiten, um das Infektionspotential zu maximieren.
- Für S. flexneri wurden Subkulturbakterien durch Zugabe von 150 & mgr; l O / N-Kultur zu 8 ml Tryptin-Soja-Agar und Inkubieren von Schütteln von 222 U / min bei 37 ° C zu einer späten exponentiellen Phase (~ 2 h), um die Expression von Virulenzgenen zu induzieren und die Infektion zu verbessern.
- Wenn die Kultur eine optische Dichte zwischen 0,6 und 0,9 (gemessen bei 600 nm) aufweist, wird 1 ml Kultur in ein 1,5 ml-Röhrchen überführt. Pellet-Bakterien für 2 min bei 8000 xg und Resuspend in Infektionsmedium (DMEM mit NEAA, ohne FCS) bei OD 600 = 1. Wenn also die OD 600 0,85 ist, resuspendieren sie in 850 μL.
ANMERKUNG: Für S. flexneri, bei OD 600 = 1, gibt es 8 x 10 8 Bakterien pro ml, wennKultiviert in 8 ml Medium. Eine Multiplizität der Infektion (MOI) von 20 ist geeignet. Für andere Pathogene muss die Anzahl der Bakterien pro ml bei einer OD 600 und die entsprechende MOI empirisch bestimmt werden.
- Wenn die Kultur eine optische Dichte zwischen 0,6 und 0,9 (gemessen bei 600 nm) aufweist, wird 1 ml Kultur in ein 1,5 ml-Röhrchen überführt. Pellet-Bakterien für 2 min bei 8000 xg und Resuspend in Infektionsmedium (DMEM mit NEAA, ohne FCS) bei OD 600 = 1. Wenn also die OD 600 0,85 ist, resuspendieren sie in 850 μL.
- Zur Verbesserung der Bakterien-Wirts-Wechselwirkungen, Mantel Bakterien mit Poly-L-Lysin (PLL).
HINWEIS: Die PLL-Behandlung von S. flexneri hatte keinen Einfluss auf die SG-Dynamik. Andere Pathogene müssen für ihre Vermeidung von PLL-Behandlung beurteilt werden.- Um Bakterien mit PLL zu beschichten, 1 ml Bakterienkultur für 2 min bei 8000 xg zentrifugieren und dann das Pellet in 10 μg / ml PLL in Phosphat-gepufferter Kochsalzlösung (PBS) bei einer OD 600 = 1 resuspendieren.
- Fügen Sie die Bakterienlösung zu einer kleinen Schale hinzu, wie z. B. einer Vertiefung innerhalb einer 12-Well-Platte, und inkubieren Sie bei RT (~ 20 ° C) für 10 min unter leichtem Rühren auf einem Plattenschüttler.
- Pellet Bakterien für 2 min bei 8.000 xg, entfernen Sie die überschüssige PLL. Resplosion des Pellets in 1 mL PBS anD wiederholen Sie diesen Waschschritt zweimal. Nach der letzten Wäsche in Infektionsmedium bei OD 600 = 1 resuspendieren.
HINWEIS: Hier ist das Infektionsmedium für S. flexneri Wirtszellmedium mit 20 mM HEPES ohne FCS.
- Für S. flexneri wurden Subkulturbakterien durch Zugabe von 150 & mgr; l O / N-Kultur zu 8 ml Tryptin-Soja-Agar und Inkubieren von Schütteln von 222 U / min bei 37 ° C zu einer späten exponentiellen Phase (~ 2 h), um die Expression von Virulenzgenen zu induzieren und die Infektion zu verbessern.
- Bereiten Sie Zellen für bakterielle Herausforderung vor.
- Medium aus Säugetier-HeLa-Zellen mit einer Saugpumpe entfernen. Füge 500 μl RT HeLa-Zellmedium hinzu, um die Zellen zu waschen, zu strudeln, Medium mit einer Saugpumpe zu entfernen und mit 500 μl RT geeigneten Infektionsmedium ( zB Wirtszellmedium mit 20 mM HEPES ohne FCS) zu ersetzen.
- HINWEIS: Für alle Waschschritte, schnell arbeiten und nur bis zu 4 Deckgläser verarbeiten, um ein Austrocknen der Zellen zu vermeiden.
- Challenge vorbereitet HeLa Zellen mit Bakterien zu infizieren 20 - 50% der Zellen.
ANMERKUNG: Für jede Bakterienart muss die geeignete Zeit und das MOI bestimmt werden. Im Allgemeinen ist ein guter Start 30 min bei 37° C mit einem MOI von 10.- Für S. flexneri die HeLa-Zellen nach einer der beiden Methoden herausfordern (Schritt 2.3.1.1 oder 2.3.1.2).
- Spin Nicht-PLL-behandelten Bakterien (20 & mgr; l OD 600 = 1 / Vertiefung von 24-Well-Platte in 500 & mgr; l Medium) auf Zellen für 10 min bei 13000 × g
- Addieren Sie PLL-beschichtete Bakterien (15 & mgr; l OD 600 = 1 / Vertiefung von 24-Well-Platte in 500 & mgr; l Medium) für 15 min bei RT, um zu ermöglichen, dass sich Bakterien auf Zellen absetzen.
- Lassen Sie die Infektion auftreten, indem Sie Zellen mit abgesetzten Bakterien in einen 37 ° C Gewebekulturinkubator für 30 min stellen.
HINWEIS: Für kurze Zeitpunkte synchronisieren Sie die S. flexneri- Infektion, indem Sie die Platten in einem 37 ° C-Wasserbad für 15 min anstelle des Gewebekulturinkubators platzieren.
- Für S. flexneri die HeLa-Zellen nach einer der beiden Methoden herausfordern (Schritt 2.3.1.1 oder 2.3.1.2).
- Stoppen Sie die Infektion durch Waschen von Zellen.
- Um Zellen zu waschen und überschüssige Bakterien zu entfernen, entfernen Sie das Medium mit einer Saugpumpe, fügen Sie 1 ml hinzuFrisches vorgewärmtes (37 ° C) HeLa Kulturmedium (DMEM, 10% FCS, NEAA). Stirpeln Sie das Medium, entfernen Sie es und ersetzen Sie es mit frischem Medium. Wiederholen Sie zweimal und lassen Sie frisches Medium auf die Zellen.
- Für S. flexneri oder andere intrazelluläre Bakterien, nach Infektion der HeLa-Zellen und der Waschungen, ersetzen Sie das Medium mit Standard-Gewebekulturmedium, das 50 μg / ml Gentamicin enthält, um extrazelluläre Bakterien zu töten und eine weitere Zellinvasion zu verhindern.
HINWEIS: Fügen Sie keine Antibiotika im Medium für extrazelluläre Bakterien hinzu.
- Für S. flexneri oder andere intrazelluläre Bakterien, nach Infektion der HeLa-Zellen und der Waschungen, ersetzen Sie das Medium mit Standard-Gewebekulturmedium, das 50 μg / ml Gentamicin enthält, um extrazelluläre Bakterien zu töten und eine weitere Zellinvasion zu verhindern.
- Um Zellen zu waschen und überschüssige Bakterien zu entfernen, entfernen Sie das Medium mit einer Saugpumpe, fügen Sie 1 ml hinzuFrisches vorgewärmtes (37 ° C) HeLa Kulturmedium (DMEM, 10% FCS, NEAA). Stirpeln Sie das Medium, entfernen Sie es und ersetzen Sie es mit frischem Medium. Wiederholen Sie zweimal und lassen Sie frisches Medium auf die Zellen.
- Inkubieren von Bakterien mit Zellen für die gewünschte Zeitdauer in einem Gewebekulturinkubator.
HINWEIS: Für S. flexneri kann die Infektion je nach Zelltyp so lang wie 6 h sein. Für HeLa-Zellen, lassen Sie die Infektion für 1,5 - 2 h vor der Zugabe von exogenen Spannungen, um SG-Bildung zu induzieren. Für Caco-2-Infektionen, bei denen Shigella Zell-Zelle verbreitet, infizieren Sie für 30 Minuten bis 6 Stunden, bevor Sie exogenen Stress hinzufügen. CeKann an dieser Stelle fixiert werden, ohne exogene Spannungen hinzuzufügen, um die Bildung von SGs als Folge der bakteriellen Infektion zu beurteilen (in diesem Fall geht es weiter zu Abschnitt 4).
3. Induktion der Stressgranulat-Bildung durch die Zugabe von exogenen Stressoren
- Entfernen Sie das Medium aus infizierten und nicht infizierten HeLa-Zellen mit einer Saugpumpe, ersetzen Sie das Medium mit 500 μl des geeigneten Mediums mit oder ohne Stressor und inkubieren Sie es.
HINWEIS: Stressinduzierende Zustände umfassen die folgenden (siehe unten). Für weitere Behandlungen siehe Kedersha et al . 13- Für die Hitzeschockbehandlung verwenden Sie regelmäßiges Medium mit 20 mM HEPES und legen Sie die Platte 30 Minuten lang in ein 44 ° C Wasserbad.
- Für Clotrimazol (induziert mitochondrialen Stress) Behandlung, verwenden Sie regelmäßige Medium ohne FCS mit 20 μM Clotrimazol und inkubieren in einem Gewebekultur-Inkubator für 1 h.
HINWEIS: Einmalige Aliquots von 20 m speichernM-Clotrimazol-Stamm in Dimethylsulfoxid (DMSO) bei -20 ° C für bis zu 4 Monate. DMSO-Fahrzeug für Kontrollzellen verwenden. - Für Arsenit (induziert oxidative Stress) Behandlung, verwenden Sie regelmäßige Medium mit 0,5 μM Arsenit und inkubieren in Gewebe Kultur Inkubator für 1 h.
HINWEIS: Einmalige Aliquots von 0,5 mM Arsenit-Lager bei -20 ° C für bis zu 6 Monate aufbewahren.
ACHTUNG: Clotrimazol und Arsenit sind gesundheitsschädlich und gefährlich und müssen entsprechend entsorgt werden. - Für die Des-Methyl-Des-Amino (DMDA) -Pateamin-Behandlung (hemmt die eukaryotische Translationsinitiierung), verwenden Sie regelmäßiges Medium mit 50 - 200 nM DMDA-Pateamin und inkubieren Sie im Gewebekulturinkubator für 1 h.
HINWEIS: DMDA-Pateamin bei -80 ° C aufbewahren. Reagenzien als kleine Aliquote aufbewahren, um Einfrieren Auftauen zu vermeiden.
4. Fixierung und Immunfluoreszenzanalyse der Stressgranulatbildung
HINWEIS: Verarbeiten Sie die Steuerung undExperimentelle Deckgläser gleichzeitig zu vermeiden, um Unterschiede zu vermeiden, die die Bildanalyse in nachfolgenden Schritten beeinflussen können. Die Kontrollproben umfassen keine Infektion mit und ohne SG-induzierende Behandlung und infizierte Proben mit und ohne SG-induzierende Behandlung.
- Entfernen Sie das Medium vollständig mit einer Saugpumpe und fixieren Sie Proben durch Zugabe von 0,5 ml RT 4% Paraformaldehyd (PFA) in PBS. 30 min bei RT inkubieren
ACHTUNG: PFA ist für den Handler und die Umwelt schädlich. Geeignete Maßnahmen ergreifen, um den Handler zu schützen und mit chemischen Abfällen zu entsorgen.
HINWEIS: Alternativ für 15 min fixieren und dann mit -20 ° C vorgebratenem Methanol für 10 min ersetzen, um die zytoplasmatische Lokalisation von SG-Markern 13 zu behalten. - Entfernen Sie die PFA mit einer Pipette und entsorgen Sie die Flüssigkeit in einem geeigneten Abfallbehälter. Waschen des Deckglases mit 1 ml Tris-gepufferter Kochsalzlösung (TBS: 25 mM Tris, pH 7,3, 15 mM NaCl). Das Deckglas für 2 min mit sanftem Schütteln inkubierenAuf einem Plattenschüttler beim Waschen.
- Entfernen Sie TBS vollständig mit einer Saugpumpe und fügen Sie 500 μl 0,3% Triton-X100 in TBS für 10 min hinzu, um die Zellen zu permeabilisieren.
- Permeabilisierungslösung mit einer Saugpumpe entfernen. Ersetzen Sie es mit 1 mL TBS, schwenken Sie die Platte vorsichtig, entfernen Sie TBS durch Absaugen und fügen Sie 1 ml Blockierungslösung (5% FCS in TBS) für 1 h bei RT hinzu.
- Bereiten Sie die primäre Antikörperlösung (1: 300 Verdünnung) in 5% FCS in TBS vor. Berechnen Sie 50 μl pro 12 mm Deckglas und machen Sie genug für alle Deckgläser in einer Charge.
HINWEIS: Die üblichen primären Antikörper, die Ziel G3BP1, eIF3b und TIA1 verwendet werden. - Spot 50 μl des primären Antikörper-Mixes auf einen Plastik-Paraffin-Film (Parafilm) fest auf die Bank oder Kammer geklebt.
HINWEIS: Eine Liste der kanonischen SG-Marker finden Sie in der Tabelle der Materialien , aber die Zusammensetzung der SGs kann von der angewandten Belastung abhängen. Andere SG-Marker sind in Kedersha et al. 13 Die erwarteten Ergebnisse für eine Reihe von SG-Markern für die S. flexneri- Infektion sind in Tabelle 1 aufgelistet. - Heben Sie das Deckglas mit Pinzette auf, tupfen Sie den Rand des Deckglases auf das Gewebe, kurz die Pinzette loslassen, um überschüssige Flüssigkeit zu entfernen, und legen Sie das Gesicht vorsichtig auf den Tropfen. Inkubieren Sie Deckgläser für 1,5 h bei RT oder über Nacht bei 4 ° C in einer feuchten Kammer.
HINWEIS: Um eine feuchte Kammer vorzubereiten, legen Sie vorverdrahtete Gewebe an den Kanten einer fest verschlossenen Kammer, die mit Plastikparafilm ausgekleidet ist. - Übertragen Sie jedes Deckglas mit Pinzette in einen Brunnen einer neuen 24-Well-Platte, die mit 1 ml TBS gefüllt ist. Mit einem sanften Schütteln auf einem Plattenschüttler für 5 min inkubieren. TBS in jeder Vertiefung mit einer Saugpumpe abziehen, mit 1 mL TBS ersetzen und 5 min auf den Plattenschüttler zurücksetzen. Wiederholen Sie noch einmal waschen.
HINWEIS: Wiederholen Sie den Parafilm, indem Sie die überschüssige Flüssigkeit mit einem Gewebe entfernen und die Oberfläche mit Wasser waschen. Stellen Sie sicher, dass der Paraffinfilm vor dem Gebrauch vollständig trocken istG für den nachfolgenden Färbungsschritt. - Die sekundäre Antikörperlösung in TBS (Verdünnung 1: 500 - 1: 2000) vorbereiten. Um den Kern und die Bakterien zu färben, füge 2 μg / ml DAPI zu der Mischung hinzu. Um Aktin zu färben, füge fluoreszierendes Phalloidin hinzu. Pipettieren Sie 50 μl sekundäre Antikörpermischung auf den Plastikparaffinfilm.
HINWEIS: Die Verwendung von hochgereinigten Kreuz-absorbierten Esel-Sekundärantikörpern wird für Co-Lokalisierungsstudien verschiedener SG-Marker empfohlen, wenn G3BP1 (Ziegen-Antikörper) verwendet wird. Wählen Sie fluoreszenzmarkierte Sekundärantikörper mit nicht überlappenden Spektren. EIF3b deutlich das Cytoplasma der Zellen und ist daher ein guter Marker, um Zellen abzugrenzen. Der Aktin-Fleck hilft weiter, Zellen an Zell-zu-Zell-Kontaktstellen und Bereichen des Bakterieneintrags abzugrenzen. - Heben Sie das Deckglas mit Pinzette auf, tupfen Sie die Kante des Deckglases auf das Gewebe, lösen Sie kurz die Pinzette, um überschüssige Flüssigkeit zu entfernen. Legen Sie das Deckglas vorsichtig nach unten auf den Tropfen und inkubieren Sie die DeckgläserIm Dunkeln für 1 h bei RT.
- Verwenden Sie eine Pinzette, um das Deckglas wieder in die 24-Well-Platten zu legen, die mit frischem 1 ml PBS gefüllt sind. Stellen Sie sicher, dass das Deckglas vollständig von der PBS abgedeckt ist. Die Zellen 3x mit sanftem Schütteln für jeweils 5 min waschen.
HINWEIS: Halten Sie die Platte während der Waschungen unter einer Abdeckung, um vor Licht zu schützen, da die Fluorophore des sekundären Antikörpers lichtempfindlich sind. - Tauchen Sie das Deckglas kurz in entionisiertes Wasser, um das Salz zu entfernen, trocknen Sie es von der Kante auf ein Gewebe und montieren Sie das Deckglas auf 5 μl Fluoreszenz-Montagemedium auf einer Glasrutsche. Das Deckglas vor der Bildgebung mit Nagellack abdichten. Halten Sie die Folien bei 4 ° C für kurzfristige (Woche) oder bei -20 ° C für Langzeitlager (Monat) auf.
HINWEIS: Vermeiden Sie Luftblasen beim Abdichten, da dies die Bildqualität beeinträchtigt.
5. Fluoreszenzbildgebung
HINWEIS: Zur Optimierung des Aufbaus siehe Bedienungsanleitung des Mikroskops.
- Erfassen Sie Bilder mit einem konfokalen Mikroskop mit einem 40 oder 63X Öl Immersionsobjektiv. Wählen Sie die gewünschten Fluoreszenzkanäle für die Bilderfassung aus. Wenn Sie mehrere fluoreszierende Kanäle verwenden, wählen Sie den sequentiellen Erfassungsmodus.
ANMERKUNG: Beginnen Sie mit den experimentellen Proben, bei denen SGs am stärksten induziert wurden, um eine Überbelichtung bei nachfolgenden Proben zu vermeiden. Achten Sie darauf, nicht überbelichtete SGs über die gesamte Tiefe der Zelle zu haben.- Stellen Sie die Bitgröße von 12 oder höher innerhalb der Mikroskopeinstellungen ein, um die Genauigkeit der Bildanalyse zu gewährleisten.
- Setzen Sie die Bildstapel auf die gesamte Tiefe der Zelle. Überprüfen Sie in den verschiedenen Kanälen und für verschiedene SG-Marker, um sicherzustellen, dass die gesamte Palette enthalten ist. Setzen Sie den Anfang des Stapels an der Basis der Zellen und die Oberseite der Stapel in der Höhe an der Oberseite der Zellen, wo keine SG-Marker beobachtet werden.
- Erwerben Sie den Stapel. Halten Sie die Stapelhöhe für alle Bilder gleich.
HINWEIS: NichtÄndern Sie die Erfassungseinstellungen zwischen den Kontroll- und den experimentellen Proben, die für den nachfolgenden Vergleich verwendet werden.
6. Bildanalyse
HINWEIS: Hier wird die SG-Analyse auf zusammengebrochenen Stacks mit Freeware ICY beschrieben. Bildanalyse von 3D-Rekonstruktionen kann auch mit anderen spezialisierten Software erfolgen. Die SG-Erkennung kann über einen vollautomatischen Workflow durchgeführt werden, der für dieses Protokoll eingerichtet wurde (verfügbar unter http://icy.bioimageanalysis.org/protocol/Stress_granule_detection_in_fluorescence_imaging). Um dieses Protokoll zu verwenden, müssen die Kerne mit einem DNA-Fleck für die Software gefärbt werden, um das Zentrum jeder Zelle zu finden, und die Zellkanten müssen entweder durch einen zytoplasmatischen Marker (wie z. B. eIF3b) oder Aktinfleck für die Software markiert werden Um die Zellgrenzen zu identifizieren. Der automatische Workflow kann verwendet werden, um manuell abgeleitete Ergebnisse zu validieren oder direkt für die Analyse, wenn Zellgrenzen mit hoher Zuverlässigkeit erkannt werden, was mo wirdSt hängen von der Dichte der Zellen und dem Marker ab, der verwendet wird, um die Zellgrenzen zu erkennen.
- Mit ImageJ oder Fidschi kollabieren Sie die Bildstapel (Bild> Stapel> Z-Projektion) und speichern als .tiff-Dateien.
HINWEIS: Erstellen Sie einen neuen Ordner für jedes Bild, da die Bildanalyse-Software ihre Ergebnisse mit der Seite des Originalbildes verknüpft. - Laden Sie eine Kontrolle und experimentelle .tiff Bild auf die Bildanalyse-Plattform (Datei> Öffnen). Gehen Sie zum Sequenzfenster. Scrollen Sie nach unten in der "Lookup-Tabelle" zu den Kanalparametern.
- Deaktivieren Sie alle Kanäle, indem Sie auf das Kontrollkästchen aller Kanalregisterkarten klicken. Klicken Sie auf Kanal 0 und wählen Sie dann die bevorzugte Farbe aus, indem Sie auf die Farbleiste klicken. Erhöhe die Intensität des Kanals nach Bedarf, um alle Strukturen zu sehen, indem du die Zeile im Intensitätsfeld klickst und verschiebst. Wiederholen Sie dies für alle Kanäle.
HINWEIS: Deaktivieren Sie Kanäle, die für eine bestimmte Aufgabe nicht benötigt werden, um die Vorspannung in der Probenanalyse zu minimieren. - ScrollenUp und innerhalb der Canvas-Registerkarte, vergrößern Sie auf ca. 10 Zellen pro Feld, indem Sie die Zoom-Registerkarte anpassen.
- Verwenden Sie die Polygon-Funktionstools (in der oberen Leiste), um Zellgrenzen von Zellen im Bild abzugrenzen. Verwenden Sie den Aktin-Fleck oder einen cytosolischen Marker für die Zellgrenzenabgrenzung ( zB eIF3b).
- Klicken Sie in den "File & ROI" -Tools auf die Polygon-Funktion. Starten Sie die Abgrenzung, indem Sie auf die Zelle klicken und dann die Linie erweitern, indem Sie Anker durch wiederholtes Klicken um die Zellgrenze machen. Um einen ROI zu beenden, klicken Sie erneut auf die Polygon-Funktion in den "File & ROI" -Tools.
HINWEIS: Identifizieren Sie die Unterpopulationen der abgegrenzten Zellen, die infiziert sind. Die Probe besteht aus einer Mischung von infizierten und nicht infizierten Zellen. Um Bakterien zu identifizieren, verwenden Sie entweder den DAPI-Fleck oder fluoreszierende Bakterien (wie GFP-exprimierende Bakterien). Erhöhe die Intensität, um sicherzustellen, dass alle Bakterien gesehen werden. - Um einen ROI zu nennen, geh zum ROI-Fenster rechts und cliCk auf der Zelle von Interesse in das Bild. Dadurch wird der entsprechende ROI auf der Registerkarte ROI hervorgehoben. Doppelklicken Sie auf den ROI-Namen und ändern Sie den Namen ( zB infizierte Zelle # 1).
HINWEIS: Wenn ein Bild verwendet werden soll, um sowohl infizierte als auch nicht infizierte Zellen zu analysieren, ist es einfacher, das Bild in zwei separaten Ordnern zu speichern und die infizierten und nicht infizierten Zellen separat zu beschreiben und zwei verschiedene Spreadsheets zu erzeugen, die die Analyse erleichtern später.
- Klicken Sie in den "File & ROI" -Tools auf die Polygon-Funktion. Starten Sie die Abgrenzung, indem Sie auf die Zelle klicken und dann die Linie erweitern, indem Sie Anker durch wiederholtes Klicken um die Zellgrenze machen. Um einen ROI zu beenden, klicken Sie erneut auf die Polygon-Funktion in den "File & ROI" -Tools.
- Öffnen Sie die Spot-Detektor-Applikation im Register "Detection & Tracking" (obere Leiste). Innerhalb der Registerkarte Eingabe wird das aktuelle Bild ausgewählt. Ändere nichts Wählen Sie auf der Registerkarte Vorverarbeitung den zu analysierenden Kanal aus.
HINWEIS: Nur ein Kanal kann zu einem beliebigen Zeitpunkt analysiert werden. Hier kann eine Batch-Analyse ausgewählt werden, wenn man die vollautomatische Analysenfolge verwendet, deren Parameter auf befriedigende Ergebnisse überprüft wurden.- Klicken Sie auf der Registerkarte Detektor auf "DeHelle Flecken über dunklem Hintergrund ", dann wählen Sie die entsprechende Skala und Empfindlichkeit aus. Testen Sie dies durch Testen verschiedener Einstellungen und Überprüfen der Fleckerkennung sowohl in der Steuerung als auch in den experimentellen Proben.
HINWEIS: Für ein Bild mit einer Auflösung von 2.048 x 2.048 und einer Pixelgröße von 0,12 μm 2 ist eine Stufe 2 Schwelle mit einer Empfindlichkeit von 25 bis 100 allgemein geeignet, aber jeder SG-Marker muss empirisch getestet werden. Stellen Sie die Spoterkennung so ein, dass bei der unbehandelten Kontrollprobe keine Spuren erkannt werden, während in der experimentellen Probe mit SGs alle kleinen und großen SGs gezählt werden. - Wählen Sie auf der Registerkarte ROI den vorgeschlagenen ROI aus der Sequenz aus. Wählen Sie auf der Registerkarte Filterung keine Filterung aus. Wählen Sie auf der Registerkarte Ausgabe die entsprechende Ausgabe aus.
HINWEIS: Die Option Enable Specific File ist hilfreich, um verschiedene Erkennungsbedingungen oder verschiedene Kanäle zu speichern. Die Auswahl des Binärbildes und des Originalbildes mit ROIs und der Erkennung ist helPful für die Qualitätskontrolle Analyse. - Wählen Sie "Vorherige Spots entfernen, die als ROIs gerendert werden". Wählen Sie den Ordner, um die Datei zu speichern, indem Sie auf die Schaltfläche "Keine Datei ausgewählt" klicken. Wählen Sie auf der Registerkarte Anzeige die gewünschten Optionen aus. Klicken Sie auf "Start Erkennung".
HINWEIS: Es wird ein Ordner mit dem Namen und dem ausgewählten Standort erstellt, der die exportierten Imaging-Optionen enthält. Diese Bilder werden für jede neue Bedingung überschrieben. Um die Bilder zu behalten, benennen Sie entweder den Ordner um, so dass ein neuer Ordner erstellt oder die Bilder aus dem Speicherordner in einen neuen Ordner übertragen wird. Für die Qualitätskontrolle der Bilder ist es hilfreich, die Anzeige-Erkennungsmarke, die Nummer der Erkennung von ROI und ROI-Nummer und Namen auszuwählen.
- Klicken Sie auf der Registerkarte Detektor auf "DeHelle Flecken über dunklem Hintergrund ", dann wählen Sie die entsprechende Skala und Empfindlichkeit aus. Testen Sie dies durch Testen verschiedener Einstellungen und Überprüfen der Fleckerkennung sowohl in der Steuerung als auch in den experimentellen Proben.
- Konsolidieren und speichern Sie die Daten für die verschiedenen Parameter mit der generierten Kalkulationstabelle für jedes Bild oder Zustand getestet.
HINWEIS: Die zu analysierenden Parameter beinhalten die Anzahl der SGs pro ROI, die SG-Flächen und die SG maxImum, durchschnittliche und minimale Intensitäten. - Übertragen Sie die Daten und analysieren Sie mit einem Analyseprogramm, das in der Lage ist, die statistische Signifikanz zu analysieren, zu graphisieren und zu bestimmen.
Ergebnisse
Um das in diesem Manuskript beschriebene Protokoll zu erläutern und zu demonstrieren, charakterisierten wir das Bild von Clotrimazol-induzierten SGs in HeLa-Zellen, die mit dem cytosolischen Pathogen S. flexneri infiziert waren oder nicht. Eine Skizze des Verfahrens ist in Abbildung 1 dargestellt und umfasst virulente und avirulierende S. flexneri, die auf kongo-rote Platten, Bakterienpräparation, Infektion, Zusatz von Umweltbelastung, Probenfixierung und Färbung, Probenbildgebung und Quantifizierung sowie Bildanalyse gestreift wurden . Eine Anzahl unterschiedlicher Spannungen können verwendet werden, um eine SG-Bildung zu induzieren, und eine Vielzahl von SG-Markern stehen zur Vernehmung zur Verfügung. EIF3b ist ein kanonischer SG-Marker, der auch das zytoplasmatische Kompartiment der Zelle deutlich färbt und zur Identifizierung von Zellkanten verwendet werden kann. G3BP1 ist ein weit verbreiteter SG-Marker, der sich ohne jegliche Hintergrundfärbung in SGs zusammensetzt ( Abbildung 2 A, B & D ). Die Zellen werden am besten mit einem konfokalen Mikroskop abgebildet und die Stapel, die die gesamte Zelltiefe abdecken, werden auf die Bildgebungsanalyse Freeware ICY geladen ( Abbildung 2 A - C ). Um infizierte Zellen besser zu identifizieren und Zellen in die infizierte oder nicht infizierte Gruppe für eine spätere Analyse zu bringen, ist es sinnvoll, die Intensität des Nukleinsäurefarbstoffs zu erhöhen ( Abbildung 2 D ). Innerhalb der Imaging-Analysesoftware wird der Spotdetektor dann verwendet, um SGs innerhalb jedes der bezeichneten ROIs zu identifizieren ( Fig. 2E), und es wird ein binäres Bild erzeugt, das alle identifizierten SGs anzeigt ( Fig. 2F ).
SG-Analysen müssen auf jeden analysierten SG-Marker zugeschnitten werden und die entsprechende Analyseanalyse muss sorgfältig ausgewählt werden. Fokussierung auf die SG maRker G3BP1, die Änderung der Größenanforderung des erkannten Punktes oder die Änderung der Empfindlichkeit der Erkennungsparameter führt zu unterschiedlichen Ergebnissen, wie in Abbildung 3 A - C gezeigt wird . Die Skalenauswahl (1 - 3, obwohl Skalen hinzugefügt werden können) basiert auf Pixelgröße und damit bei höheren Skalen, kleinere SGs werden nicht gezählt und die Anzahl der SGs wird abnehmen ( Abbildung 3 C ). Die Empfindlichkeit wird von 1 - 100 gemessen, wobei 100 die empfindlichste ist. Durch Erhöhung der Empfindlichkeit erhöht sich die Anzahl der erkannten SGs ( Abbildung 3 C ). So müssen die richtigen Einstellungen gefunden werden, um falsche Positive und falsche Negative zu minimieren. Dies geschieht am besten durch sorgfältige Analyse der für jede Einstellung erhaltenen Visuals. Für G3BP1 ergab eine Skala von 2 mit einer Empfindlichkeit von 100 (2 - 100, rot hervorgehoben) das beste Ergebnis. Eine Skala von 2 mit einer niedrigeren Empfindlichkeit (50 oder 25) links kleinSGs unzählbar (siehe orange Pfeile). Ähnlich wurde eine Skala von 3 linken kleinen SG nicht berücksichtigt, während eine Skala von 1 überabgetastet wurde. Wichtig ist, dass zusätzliche Skalen hinzugefügt werden können, um sich nur auf große SGs zu konzentrieren, falls dies gerechtfertigt ist. Für eIF3b gab eine Skala von 2 mit einer Empfindlichkeit von 55 (2 - 55) das beste Ergebnis für eIF3b (rot markiert), obwohl rote Pfeile entweder unter oder überabtasten in einer Skala 2 - 50 & 2 - 55 hervorheben.
Sobald die Parameter für die SG-Analyse richtig eingestellt sind, können die Daten in vielerlei Hinsicht analysiert werden ( Abbildung 4 ). Der Spotdetektor gibt die Anzahl der SGs, die in jedem ROI detektiert werden, so dass die nicht infizierten und infizierten Zellen verglichen werden können ( Abbildung 4 A ). Ähnlich ist die Größe, in diesem Fall die Anzahl der Pixel, gegeben, aus denen die Oberfläche berechnet werden kann ( Abbildung 4 B ). Frequenzverteilung plOts sind auch nützlich, um Verschiebungen in der Größe von SGs hervorzuheben, die in verschiedenen Zellpopulationen gefunden wurden. Hier wird eine vollständige Abwesenheit von großen SGs in S. flexneri infizierten Zellen sowie eine definitive Verschiebung der Verteilung klarer ( Abbildung 4 C ). Darüber hinaus gibt die Intensität (hier gezeigt ist minimale und maximale Intensität) Informationen über die Qualität der analysierten SGs. Für S. flexneri infizierte Zellen sind SGs deutlich weniger intensiv. Diese Analysen liefern statistisch relevante Informationen über die qualitative und quantitative Natur von SGs, die als Reaktion auf exogenen Stress gebildet werden, wenn Zellen mit oder ohne S. flexneri infiziert sind.

Abbildung 1 : Überblick über die experimentelle Prozedur zur Infektion von Zellen mit S. Flexneri undUm die Wirkung der Infektion SG Formation zu analysieren. Eine kongo-rote Kolonie und eine nichtvirulent Kongo-Rot-negative Kolonie werden in der tryptischen Sojabohnenbrühe gezüchtet und gewachsen. Die Übernachtkultur wird in die späte exponentielle Phase subkultiviert, bevor Bakterien mit PLL beschichtet werden, um die Adhärenz zu fördern. Wirtszellen, die auf Glasdeckgläschen in 12-Well-Platten gezüchtet wurden, werden mit Bakterien für 30 min infiziert. Kontrollzellen sind nicht infiziert. Die Zellen werden gewaschen und mit Stress-Induktoren behandelt, um die SG-Bildung zu induzieren, indem entweder das Medium durch spannungshaltiges Medium ersetzt wird oder die Zellen Stressbedingungen wie Wärme ausgesetzt werden. Die Zellen werden dann fixiert, permeabilisiert, mit immunfluoreszierenden SG-spezifischen Markern gefärbt und mit konfokaler Mikroskopie abgebildet. Z-Projektionen werden mit ImageJ durchgeführt und mit dem Spot-Detektor der Bildanalyse-Software analysiert. Die Daten werden dann analysiert. Bitte klicken Sie hier um eine größere Ansicht zu sehenVersion dieser Figur.
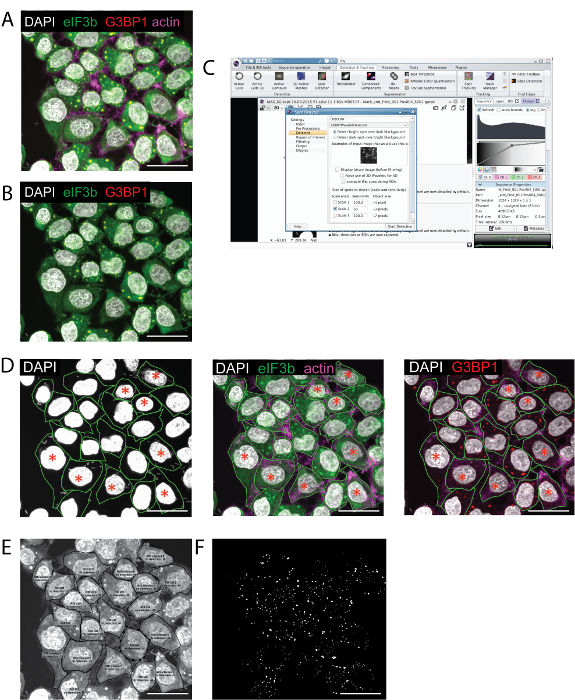
Abbildung 2 : Spot-Detektor-Analyse von HeLa-Zellen infiziert mit S. Flexneri für 1,5 h vor der Zugabe von Clotrimazol für 1 h. A. Immunfluoreszenzbild einer z-Projektion, die Zellen zeigt, die mit den SG-Markern eIF3b und G3BP1, DAPI und Actin gefärbt sind. B. Gleiches Bild wie in (A), aber ohne den Aktin-Fleck, um die Verwendung von eIF3b hervorzuheben, um Zellgrenzen abzugrenzen. C. Screenshot der Bildanalyse-Software mit dem Spot-Detektor-Modul. D. Gating der infizierten und nicht infizierten Zellen, wie sie mit verschiedenen Kanälen zu sehen sind. Um alle Bakterien zu sehen, wird die Intensität erhöht. Zellen mit mehr als 1 Bakterium in der Zelle sind mit einem roten Stern markiert. E Ausgabe des SpotdetektorsDie Analyse der einzelnen ROIs, die den ROI-Namen, die Anzahl der Spots und ihren Standort angeben. F. Bild von Spots in den ROIs erkannt. Maßstab = 30 μm. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.

Abbildung 3 : Vergleiche von verschiedenen Skalen- und Empfindlichkeitseinstellungen des Spot-Detektors für die Erkennung von SGs durch verschiedene SG-Marker. A. Zoom-in HeLa-Zellen infiziert oder nicht mit S. flexneri und gefärbt mit DAPI und dem SG-Marker G3BP1 und analysiert auf verschiedenen Skalen (Pixelgröße abgeschnitten, 1 - 3) und Empfindlichkeit (1 - 100). Grafische Darstellung von SG-Zahlen in nicht infizierten und infizierten Zellen, die auf verschiedenen Skalen analysiert wurden ( > B) und Empfindlichkeiten ( C ). Visuals ( D ) und grafische Darstellung ( E ) der SG-Nummernerkennung des SG-Markers eIF3b für das gleiche Bild bei Änderung der Empfindlichkeitsgrenze ohne Änderung der Spotgrößenabschaltung. Rotschreiben und rote Symbole zeigen die besten Parameter an. Rote Pfeile zeigen auf falsche Positives und orange Pfeile auf einen Mangel an SG-Erkennung. Werte sind Mittelwert mit Standardabweichung. Die besten Ergebnisse sind rot markiert. Ausgewählte statistische Analysen enthalten zur Klarheit mit dem Wilcoxon Rang Summe Test P-Werte auf der linken Seite und die Varianz F-Test auf der rechten Seite. * = <0,05, ** = <0,01, *** = <0,001, ns = nicht signifikant. Maßstab = 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Es / ftp_upload / 55536 / 55536fig4.jpg "/>
Abbildung 4 : Analyse von Spot Detector Generierte SG-Daten aus Clotrimazol-behandelten HeLa-Zellen, die mit S. Flexneri infiziert wurden . A. Anzahl der G3BP1-SGs pro Zelle in infizierten und nicht infizierten HeLa-Zellen. B. Oberfläche von G3BP1-SGs in infizierten und nicht infizierten Zellen und deren prozentuale Verteilungshäufigkeit ( C ). D. Minimaler und maximaler Intensitätswert (beliebig) von G3BP1-SGs. Werte sind Mittelwert mit Standardfehler. Ausgewählte statistische Analysen enthalten zur Klarheit mit dem Wilcoxon Rang Summe Test P-Werte auf der linken Seite und die Varianz F-Test auf der rechten Seite. * = <0,05, ** = <0,01, *** = <0,001, ns = nicht signifikant. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Diskussion
Das hier beschriebene Protokoll beschreibt die Induktion, Lokalisierung und Analyse von SGs in nicht-infizierten Zellen und Zellen, die mit dem cytosolischen Pathogen S. flexneri in Gegenwart oder Abwesenheit von exogenem Stress infiziert sind. Mit Hilfe der kostenlosen Imaging-Software erlauben die Protokolle die präzise qualitative und quantitative Analyse der SG-Bildung, um die Unterschiede in den gegebenen Phänotypen zu identifizieren und statistisch zu adressieren.
Es gibt mehrere kritische Schritte innerhalb des Protokolls für die Infektion, SG-Induktion und Bildgebung Teile. Für die Infektion ist es wichtig, dass die bakterielle Interaktion mit Wirtszellen, wie zB Invasion als skizziertes Protokoll, so weit wie möglich synchronisiert wird. Dies ist besonders wichtig bei der Untersuchung der Auswirkungen der bakteriellen Herausforderung früh während des Infektionsprozesses. Wir haben zuvor gezeigt, dass einige Phänotypen, wie die eIF3b-Lokalisation nach S. flexneri- Infektion, frühzeitig im Wildtyp gestört werdenS. flexneri- Herausforderung, während andere Phänotypen, wie Aggregation von G3BP1-haltigen SGs, zu späteren Zeitpunkten 3 stärker betroffen sind. Um die zeitliche Wirkung der bakteriellen Herausforderung genau zu beschreiben, ist die Synchronisation der Infektion ratsam. Um die SG-Bildung zu analysieren, sollten die Zellen in der exponentiellen Phase des Wachstums liegen, und daher ist die Zelldichte zum Zeitpunkt der Stressaddition ebenfalls ein wichtiger Parameter. Es beschränkt auch die SG-Analyse auf nicht konfluierende Zellinfektionsmodelle. Ein weiterer kritischer Aspekt ist die Co-Verarbeitung von Kontroll- und Versuchsmustern sowohl bei der Verarbeitung der Proben für die Bildgebung als auch bei der Bildaufnahme. Für die Immunfluoreszenzverarbeitung sollten alle Proben mit den gleichen Antikörper-Herstellungsverdünnungen für die gleiche Zeitspanne und unter den gleichen Bedingungen ( dh RT Vs. 4 ° C) gefärbt werden, während auch darauf geachtet wird, jedes Deckglas ähnlich während der Waschstufen zu behandeln Montage oDie Deckgläser. Damit werden vergleichbare immunfluoreszierende Färbungen sichergestellt, die sowohl qualitativ als auch quantitativ analysiert werden können. Unterschiede im Montagemedium können bei der Bildaufnahme erhebliche Auswirkungen auf das Bleichen der Proben haben und sollten daher konstant gehalten werden. Ebenso sollte die Bildaufnahme für alle zu analysierenden Proben in einer Sitzung durchgeführt werden und mit den gleichen Einstellungen, um Unterschiede, die sich aus der Laserfestigkeit oder dem Probenaufbau ergeben, zu minimieren.
Bei der Verwendung von exogenen Belastungen ist es wichtig, das Reagenz ordnungsgemäß zu lagern und häufig neue Arbeitsbestände zu machen. Verlängerte Perioden (> 4 Monate für Clotrimazol zum Beispiel) führen zu einer verminderten Potenz des Medikaments und beeinflussen die SG-Bildung. Reagenzien sollten dem Kulturmedium unmittelbar vor der Zugabe zu den Zellen hinzugefügt werden; Wenn eine Verringerung der SG-Bildung in Kontrollzellen beobachtet wird oder die Aggregation von SGs als fehlerhaft erscheint, sollten Reagenzien auf ihre ac getestet werdenUnd neue Bestände sollten gemacht werden.
Eine Einschränkung ist, dass die SG-Identifikation unter Verwendung des Spotdetektors weniger zuverlässig wird, wenn zu viel Hintergrundfluoreszenz vorhanden ist. Während dies für viele SG-Marker kein Problem darstellt, sind einige, wie z. B. eIF3b, die ein klarer zytosolischer Marker unter SG-induzierenden und nicht-induzierenden Zuständen sind, schwieriger zu analysieren, wie in Abbildung 3 gezeigt . Darüber hinaus müssen Skalen und Empfindlichkeiten empirisch für jeden SG-Marker bestimmt werden. Eine weitere Einschränkung ist, dass die Analyse durch die Bildanalyse-Software am besten mit 2D-Bildern ist und daher gut auf optischen Scheiben oder zusammengebrochenen Stapeln funktioniert, was jedoch zu einem Verlust von 3D-Informationen führt. Die Analyse auf z-Projektionen neigt daher dazu, die Größe von SGs zu übertreiben und die Anzahl der SGs zu unterschätzen. Die 3D-Analyse von SGs kann mit Imaris durchgeführt werden, um eine noch genauere räumliche Charakterisierung zu ermöglichen, wenn dies für einen bestimmten Phänotyp notwendig ist. Die SG-Charakterisierung kann schnell und standardisiert mit dem automatisierten Spotdetektor der Bildanalyse-Software durchgeführt werden. Im Gegensatz dazu, manuelle Methoden zur Identifizierung und Abgrenzung von SGs wie zuvor durchgeführt 4 , lassen Sie die Analyse offen für mehr Bias und ist erheblich zeitaufwendiger. SG-Analyse mit Spot-Detektor kann auch maßgeschneidert werden, um kleine SG-Aggregate einzuschließen oder auszuschließen, indem sie unterschiedliche Empfindlichkeits- und Größenschwellen auswählen und dadurch Flexibilität bei der Bewertung verschiedener Aspekte der SG-Bildung ermöglichen. Die SG-Analyse kann auch vollständig automatisiert mit einem etablierten automatisierten Workflow durchgeführt werden 3 . Innerhalb dieses Workflows wird das Zentrum der Zelle durch einen DAPI-Fleck identifiziert, und das Programm lässt dann eine verformbare Sphäre nach außen wachsen, um die Zellgrenzen auf der Basis eines zytoplasmatischen Flecks (wie z. B. eIF3b) oder Aktin abzugrenzen. Zur gleichen Zeit, mit einer halbautomatischen Analyse vonManuelles Abgrenzen einzelner Zellen für die Analyse, kann vorteilhaft sein, wenn Zellen in Clustern wachsen und Zellgrenzen sind schwierig für das automatisierte Programm klar abgrenzen. Unter diesen Umständen kann die Definition von Zellgrenzen manuell falsch positive oder negative Werte beseitigen.
Das Protokoll kann an andere SG-induzierende Zustände und an andere Pathogene einschließlich Viren, Bakterien, Hefe und Protozoen angepasst werden. Von besonderem Interesse können andere zytosolische Pathogene wie Rickettsia spp., Francisella tularensis , Burkholdieria pseudomallei und Listeria spp. 14 Für jedes infektiöse Agens und ggf. auch verschiedene Zelllinien muss das Infektionsprotokoll angepasst und die Probenzeiten exogener Spannungen empirisch bestimmt werden. Darüber hinaus kann die SG-Charakterisierung mit Bildanalyse-Software auch zukünftig auf die Live-Zell-Bildgebung erweitert werden. Echtzeit-SG-Analyse wäre notwendigDie Expression eines oder mehrerer fluoreszenzmarkierter SG-Marker in den Wirtszellen zu vermitteln, aber damit die Frage nach der zeitlichen und speziellen Dynamik der SG-Bildung unter verschiedenen experimentellen Bedingungen adressieren zu können. Fluoreszenzmarkierte Bakterien würden dann benötigt, um die Echtzeit-SG-Analyse in infizierten und nicht infizierten Zellen zu ergänzen.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
PS ist ein Empfänger der Bill und Melinda Gates Grand Challenge Grant OPP1141322. PV wurde von einem Schweizerischen National Science Foundation Early Postdoc Mobility Stipendium und einem Roux-Cantarini Postdoktorandenstipendium unterstützt. PJS wird von einem HHMI-Stipendium und ERC-2013-ADG 339579-Entschlüsselung unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Primary Antibodies | |||
| eIF3b (N20), origin goat | Santa Cruz | sc-16377 | Robust and widely used SG marker. Cytosolic staining allows cell delineation. Dilution 1:300 |
| eIF3b (A20), origin goat | Santa Cruz | sc-16378 | Same target as eIF3b (N20) and in our hands was identical to eIF3b (N20). Dilution 1:300 |
| eIF3A (D51F4), origin rabbit (MC: monoclonal) | Cell Signaling | 3411 | Part of multiprotein eIF3 complex with eIF3b . Dilution 1:800 |
| eIF4AI, origin goat | Santa Cruz | sc-14211 | Recommended by (Ref # 13). Dilution 1:200 |
| eIF4B, origin rabbit | Abcam | ab186856 | Good stress granule marker in our hands. Dilution 1:300 |
| eIF4B, origin rabbit | Cell Signaling | 3592 | Recommended by Ref # 13. Dilution 1:100 |
| eIF4G, origin rabbit | Santa Cruz | sc-11373 | Widely used SG marker. (Ref # 13): may not work well in mouse cell lines. Dilution 1:300 |
| G3BP1, origin rabbit (MC: monoclonal) | BD Biosciences | 611126 | Widely used SG marker. Dilution 1:300 |
| Tia-1, origin goat | Santa Cruz | sc-1751 | Widely used SG marker. Can also be found in P bodies when SG are present (Ref # 13). Dilution 1:300 |
| Alexa-conjugated Secondary Antibodies | |||
| A488 anti-goat , origin donkey | Thermo Fisher | A-11055 | Cross absorbed. Dilution 1:500 |
| A568 anti-goat, origin donkey | Thermo Fisher | A-11057 | Cross absorbed. Dilution 1:500 |
| A488 anti-mouse, origin donkey | Thermo Fisher | A-21202 | Dilution 1:500 |
| A568 anti-mouse, origin donkey | Thermo Fisher | A10037 | Dilution 1:500 |
| A647 anti-mouse, origin donkey | Thermo Fisher | A31571 | Dilution 1:500 |
| A488 anti-rabbit, origin donkey | Thermo Fisher | A-21206 | Dilution 1:500 |
| A568 anti-rabbit, origin donkey | Thermo Fisher | A10042 | Dilution 1:500 |
| Other Reagents | |||
| Shigella flexneri | Available from various laboratories by request | ||
| Tryptone Casein Soya (TCS) broth | BD Biosciences | 211825 | Standard growth medium for Shigella, application - bacterial growth |
| TCS agar | BD Biosciences | 236950 | Standard growth agar for Shigella, application - bacterial growth |
| Congo red | SERVA Electrophoresis GmbH | 27215.01 | Distrimination tool for Shigell that have lost the virulence plasmid, application - bacterial growth |
| Poly-L-Lysine (PLL) | Sigma-Aldrich | P1274 | Useful to coating bacteria to increase infection, application - infection |
| Gentamicin | Sigma-Aldrich | G1397 | Selective killing of extracellular but not cytosolic bacteria, application - infection |
| HEPES | Life Technologies | 15630-056 | PH buffer useful when cells are incubated at RT, application - cell culture |
| Dulbecco's Modified Eagle Medium (DMEM) | Life Technologies | 31885 | Standard culture medium for HeLa cells, application - cell culture |
| Fetal Calf Serum (FCS) | Biowest | S1810-100 | 5% supplementation used for HeLa cell culture medium, application - cell culture |
| Non-Essential Amino Acids (NEAA) | Life Technologies | 11140 | 1:100 dilution used for HeLa cell culture medium, application - cell culture |
| DMSO | Sigma-Aldrich | D2650 | Reagent diluent, application - cell culture |
| Sodium arsenite | Sigma-Aldrich | S7400 | Potent stress granule inducer (Note: highly toxic, special handling and disposal required), application - stress inducer |
| Clotrimazole | Sigma-Aldrich | C6019 | Potent stress granule inducer (Note:health hazard, special handling and disposal required), application - stress inducer |
| Paraformaldehyde (PFA | Electron Microscopy Scences | 15714 | 4% PFA is used for standard fixation of cells, application - fixation |
| Triton X-100 | Sigma-Aldrich | T8787 | Used at 0.03% for permeabilizationof host cells before immunofluorescent staining, application - permeabilization |
| A647-phalloidin | Thermo Fisher | A22287 | Dilution is at 1:40, best added during secondary antibody staining, application - staining |
| DAPI | Sigma-Aldrich | D9542 | Nucleid acid stain used to visualize both the host nucleus and bacteria, application - staining |
| Parafilm | Sigma-Aldrich | BR701501 | Paraffin film useful for immunofluorescent staining of coverslips, application - staining |
| Prolong Gold | Thermo Fisher | 36930 | Robust mounting medium that works well for most fluorophores , application - mounting |
| Mowiol | Sigma-Aldrich | 81381 | Cheap and robust mounting medium that works well for most fluorophores, application - mounting |
| 24-well cell culture plate | Sigma-Aldrich | CLS3527 | Standard tissue culture plates, application - cell culture |
| 12 mm glass coverslips | NeuVitro | 1001/12 | Cell culture support for immunofluorescent applications, application - cell support |
| forceps | Sigma-Aldrich | 81381 | Cheap and robust mounting medium that works well for most fluorophores, application - mounting |
| Programs and Equipment | |||
| Prism | GraphPad Software | Data analysisand graphing program with robust statistical test options, application - data analysis | |
| Leica SP5 | Leica Microsystems | Confocal microsope, application - image acquisition | |
| Imaris | Bitplane | Professional image analysis program, application - data analysis | |
| Excel | Microsoft | Data analysis and graphing program, application - data analysis |
Referenzen
- Reineke, L. C., Lloyd, R. E. Diversion of stress granules and P-bodies during viral infection. Virology. 436 (2), 255-267 (2013).
- Kedersha, N., Ivanov, P., Anderson, P. Stress granules and cell signaling: more than just a passing phase. Trends Biochem Sci. 38 (10), 494-506 (2013).
- Vonaesch, P., Campbell-Valois, F. -. X., Dufour, A., Sansonetti, P. J., Schnupf, P. Shigella flexneri modulates stress granule composition and inhibits stress granule aggregation. Cell Microbiol. 18 (7), 982-997 (2016).
- Kolobova, E., Efimov, A., et al. Microtubule-dependent association of AKAP350A and CCAR1 with RNA stress granules. Exp Cell Res. 315 (3), 542-555 (2009).
- Anderson, P., Kedersha, N. Stress granules: the Tao of RNA triage. Trends in biochemical sciences. 33 (3), 141-150 (2008).
- Anderson, P., Kedersha, N. Stressful initiations. J Cell Sci. 115, 3227-3234 (2002).
- Mohr, I., Sonenberg, N. Host translation at the nexus of infection and immunity. Cell Host Microbe. 12 (4), 470-483 (2012).
- Hu, S., Claud, E. C., Musch, M. W., Chang, E. B. Stress granule formation mediates the inhibition of colonic Hsp70 translation by interferon- and tumor necrosis factor. Am J Physiol Gastointest Liver Physiol. 298 (4), 481-492 (2010).
- Barry, E. M., Pasetti, M. F., Sztein, M. B., Fasano, A., Kotloff, K. L., Levine, M. M. Progress and pitfalls in Shigella vaccine research. Nat Rev Gastroenterol Hepatol. 10 (4), 245-255 (2013).
- Lanata, C. F., Fischer-Walker, C. L., et al. Global Causes of Diarrheal Disease Mortality in Children. PloS One. 8 (9), 72788 (2013).
- Ashida, H., Ogawa, M., et al. Shigella deploy multiple countermeasures against host innate immune responses. Curr Opin Microbiol. 14 (1), 16-23 (2011).
- Ashida, H., Ogawa, M., Mimuro, H., Kobayashi, T., Sanada, T., Sasakawa, C. Shigella are versatile mucosal pathogens that circumvent the host innate immune system. Curr Opin Immunol. 23 (4), 448-455 (2011).
- Kedersha, N., Anderson, P. Mammalian stress granules and processing bodies. Methods Enzymol. 431, 61-81 (2007).
- Ray, K., Marteyn, B., Sansonetti, P. J., Tang, C. M. Life on the inside: the intracellular lifestyle of cytosolic bacteria. Nat Re. Micro. 7 (5), 333-340 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten