Method Article
Bestimmung des Zetapotentials über Nanoparticle Translokation Geschwindigkeiten durch eine Tunable Nanopore: Die Verwendung von DNA-modifizierten Partikel als Beispiel
In diesem Artikel
Zusammenfassung
Hier verwenden wir ein Polyurethan abstimmbaren Nanopore in eine resistive Pulserfassungstechnik integriert Nanoteilchen Oberflächenchemie über die Messung der Partikelgeschwindigkeiten Translokation zu charakterisieren, die verwendet werden können, das Zeta-Potential der einzelnen Nanopartikel zu bestimmen.
Zusammenfassung
Nanopore Technologien, die zusammen als Resistive Pulssensoren (RPS) bekannt ist, werden verwendet, um Proteine, Moleküle und Nanopartikel zu erkennen, zu quantifizieren und zu charakterisieren. Tunable resistive Pulserkennung (TRPS) ist eine relativ neue Anpassung an RPS, die einen abstimmbaren Poren enthält, die in Echtzeit verändert werden kann. Hier wir TRPS verwenden , um die Translokation Zeiten von DNA-modifizierten Nanopartikel zu überwachen , da sie die abstimmbaren Porenmembran als Funktion der DNA - Konzentration und Struktur (dh einzelsträngig zu doppelsträngiger DNA) durchqueren.
TRPS basiert auf zwei Ag / AgCl Elektroden durch eine elastomere Porenmembran getrennt, die eine stabile Ionenstrom auf ein angelegtes elektrisches Feld aufbaut. Im Gegensatz zu verschiedenen optisch-basierten Partikelcharakterisierung Technologien können TRPS charakterisieren einzelnen Partikel unter einer Probenpopulation, so dass für multimodale Proben mit Leichtigkeit analysiert werden. Hier zeigen wir, Zetapotentialmessungenüber Partikel Translokation Geschwindigkeiten von bekannten Standards und wenden diese Analyten Translokation mal abzutasten, wodurch sich das Zetapotential dieser Analyten zu messen.
Sowie Mittel zeta Potentialwerte Erfassen werden die Proben alle unter Verwendung eines Partikel-by-Teilchen Perspektive zeigen weitere Informationen auf einer gegebenen Probe durch die Probenpopulation Verteilungen, zum Beispiel. Von solchen, zeigt dieses Verfahren Potential in Sensoranwendungen für beide medizinischen und ökologischen Bereich.
Einleitung
Funktionalisierte Nanopartikel werden immer beliebter als Biosensoren sowohl in medizinischen und ökologischen Bereich. Die Fähigkeit , eine Nanopartikel der Oberflächenchemie, die mit DNA, beispielsweise zu verändern, erweist sich nützlich für die gezielte Arzneimittelverabreichungssysteme 1 und Überwachung DNA-Protein - Wechselwirkungen 2-4. Eine zunehmend verbreitete Nanopartikel Eigenschaft wird in Biotests verwendet und bei der Bereitstellung von Therapeutika ist Superpara 5. Superparamagnetische Partikel (SPPS) sind äußerst nützlich bei der Identifizierung und spezifischen Analyten aus komplexen Mischungen zu entfernen und kann so mit der einfachen Verwendung eines einzigen Magneten tun. Einmal entfernt, kann der Analyt-gebundenen Teilchen charakterisiert und analysiert werden, für den Zweck geeignet.
Frühere Verfahren verwendet zum Nachweis und zur Charakterisierung von Nanoteilchen umfassen optische Techniken, wie beispielsweise dynamische Lichtstreuung (DLS), die auch als Photonenkorrelationsspektroskopie bekannt. Obwohl ein hallogh Durchsatz Technik wird DLS beschränkt zu sein , eine Mittelungs basierte Technik und bei der Analyse von multimodalen Proben ohne die Zugabe von Spezialsoftware, die größeren Teilchen eine viel dominanter Signal, produzieren einige der kleineren Teilchen völlig unbemerkt 6,7 zu verlassen. Particle-by-Teilchen sind Charakterisierungstechniken daher wesentlich günstiger Nanopartikel und funktionalisierte Nanopartikel-Systeme zu analysieren.
RPS basierten Technologien basieren auf ein elektrisches Feld an eine Probe Aufbringen und Überwachen des Transportmechanismus der Partikel durch einen synthetischen oder biologischen Nanopore. Eine relativ neue Nanopartikel - Detektion und Charakterisierung Technik basiert auf RPS ist abstimmbaren Widerstandspulserkennung (TRPS) 16.08. TRPS ist ein Zwei-Elektrodensystem durch ein elastomeres, abstimmbaren Porenmembran getrennt. Abstimmbarer Poren Verfahren ermöglicht Analyten aus einer Reihe von Form 17 und eine Größe über ihre trans zu messPort-Mechanismen durch die Pore. Abstimmbaren Poren wurden zuvor für die Detektion von kleinen Partikeln (70-95 nm Durchmesser) Herstellung vergleichbare Ergebnisse zu anderen Techniken , wie beispielsweise Transmissionselektronenspektroskopie (TEM) 10 verwendet. Wenn ein elektrisches Feld angelegt wird, wird ein Ionenstrom beobachtet und als Partikel / Moleküle durch die Poren passieren, sie vorübergehend die Poren blockieren, eine Verringerung der Strom zu verursachen, die als "Blockade Ereignis" definiert werden kann. Jede Blockade Ereignis ist repräsentativ für ein einzelnes Teilchen , so dass jedes Teilchen in einer Probe einzeln auf der Blockade Größe basierend charakterisiert werden kann, Δ 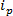 Halb Maximum und voller Breite, FWHM, sowie andere Blockade Eigenschaften. Analysieren von einzelnen Teilchen, wie sie durch eine Nanopore passieren ist vorteilhaft für die multimodale Proben als TRPS erfolgreich und effektiv eine Reihe unterscheiden von Partikelgrößen amongst eine einzelne Probe. Tunable resistive Pulserfassungs vervollständigt Größe 10, Zetapotential 12,18 und Konzentration 15 Messungen gleichzeitig in einem einzigen Durchlauf und kann daher noch unterscheiden Proben ähnlich, wenn nicht die gleiche Größe durch ihre Oberflächenladung 19; ein Vorteil gegenüber alternativen Sizing-Techniken.
Halb Maximum und voller Breite, FWHM, sowie andere Blockade Eigenschaften. Analysieren von einzelnen Teilchen, wie sie durch eine Nanopore passieren ist vorteilhaft für die multimodale Proben als TRPS erfolgreich und effektiv eine Reihe unterscheiden von Partikelgrößen amongst eine einzelne Probe. Tunable resistive Pulserfassungs vervollständigt Größe 10, Zetapotential 12,18 und Konzentration 15 Messungen gleichzeitig in einem einzigen Durchlauf und kann daher noch unterscheiden Proben ähnlich, wenn nicht die gleiche Größe durch ihre Oberflächenladung 19; ein Vorteil gegenüber alternativen Sizing-Techniken.
Zeta - Potential ist als das elektrostatische Potential an der Scherebene 20 definiert ist , und wird aus Partikelgeschwindigkeiten berechnet , da sie eine Pore 19 durchqueren. Zeta-Potential-Messungen einzelner Partikel gibt damit einen Einblick in die Translokation Mechanismen und das Verhalten der Nanopartikel-Systemen in Lösung, wertvolle Informationen für die Zukunft von Nanopartikel-Assay-Designs für eine Vielzahl von Anwendungen. Particle-by-Partikelanalyse solcher Art, ermöglicht auch die Verbreitung und Verteilung von Zetapotentialwerte unter einer Stichprobe untersucht werden, um weitere Informationen erlauben on Reaktionskinetik (einzelsträngig zu doppelsträngiger DNA, zum Beispiel) und Partikelstabilitäten in Lösung erreicht werden.
Hier beschreiben wir eine Technik, und charakterisiert Oberflächen sowohl unmodifizierte und DNA-modifizierten SPP erkennt. Das beschriebene Protokoll ist hier anwendbar auf eine Reihe von anorganischen und biologischen Nanopartikel, aber wir demonstrieren das Verfahren DNA-modifizierten Oberflächen aufgrund ihrer breiten Palette von Anwendungen. Die Technik ermöglicht es dem Benutzer, zwischen einzelsträngigen und doppelsträngigen DNA-Ziele auf einer Nanopartikeloberfläche zu unterscheiden, basierend auf Partikel Translokation Geschwindigkeiten durch ein Porensystem und somit deren Zeta-Potentiale.
Protokoll
1. Herstellung der Phosphat-gepufferte Saline mit Tween-20 (PBST) Puffer
- Auflösen einer Tablette PBS (0,01 M Phosphatpuffer, 0,0027 M Kaliumchlorid, 0,137 M Natriumchlorid, pH 7,4) in 200 ml deionisiertem Wasser (18,2 M & OHgr; cm).
- Füge 100 & mgr; l (0,05 (v / v)%) Tween-20 in die 200 ml Pufferlösung als Tensid.
2. Vorbereitung Carboxylende Polystyrolteilchengröße Standards
- Vortexen die Kalibrierungspartikeln für 30 sec vor der Beschallung für 2 min bei 80 Watt Monodispersität der Teilchen zu erzeugen.
- Verdünnen Sie die Kalibrierung Partikel 1 in 100 bis zu einer Konzentration von 1x10 10 Teilchen / ml in PBST - Puffer und Vortex für 30 Sekunden.
3. Vorbereitung Streptavidin beschichtete Teilchen
- Vortexen die Partikel für 30 sec vor der Beschallung für 2 min bei 80 Watt Monodispersität zu gewährleisten.
- Verdünnen Sie die Streptavidin-beschichteten Teilchen 1 in 100 in PBST Puffer achieve eine resultierende Konzentration von 1x10 9 Teilchen / ml und Vortex für 30 Sekunden.
Hinweis: Ein typisches Probenvolumen 200 ul ist. Wenn beispielsweise die Untersuchung von fünf DNA-Konzentrationen vorzubereiten 1 ml verdünntes Streptavidin beschichteten Teilchen.
4. Herstellung von Oligonucleotiden
- Rekonstituieren Oligonukleotiden mit deionisiertem Wasser bis zu einer resultierenden Konzentration von 100 uM.
5. Die Zugabe von Fänger-Sonde (CP) DNA an die Streptavidin beschichtete Teilchen
- Vor der DNA-Bindung, vortex die Streptavidin-beschichteten Teilchen (200 & mgr; l Probenvolumen) für 30 sec mit einem 80 Watt Beschallung 2 min gefolgt.
- Auf der Basis der Bindungskapazität des Lieferanten (4.352 pmol / mg) versehen ist, fügt die entsprechende Konzentration an DNA auf die Teilchen zur resultierenden Konzentrationen von 10, 20, 30, 40, 47, 95, 140 und 210 nM DNA.
- Vortex, um die Proben für 10 Sekunden und auf ein Drehrad bei Raumtemperaturfür 30 min für die DNA zu erlauben, über eine Streptavidin-Biotin-Wechselwirkung an die Partikeloberflächen zu binden.
- Sobald der Fänger-DNA mit den Streptavidin-beschichteten Teilchen wurde zugegeben und inkubiert, das überschüssige DNA in Lösung durch magnetische Trennung, indem die Proben auf einem Magnetzahnstange Plazieren für 30 min.
- Entfernen Sie den Überstand, kümmert sich nicht um die neu gebildete Gruppe von Teilchen der Nähe des Magneten zu stören, und ersetzen mit dem gleichen Volumen an neuen PBST-Puffer.
6. Hybridisierende Complementary DNA an die CP-Teilchen
- Die erforderliche Menge an Ziel-DNA (im Überschuß bei 500 nM), um die maximal mögliche Ziel sicherzustellen Bindung erreicht wurde.
- Vortex die Proben 10 sec und auf ein Drehrad bei Raumtemperatur für 30 min.
- Sobald die Hybridisierung abgeschlossen ist, entfernen Sie das überschüssige Ziel-DNA über magnetische Trennung, indem die Proben auf einem Magnet Rack platzieren für 30 min.
- Entfernen Sie den Überstand, Kümmert sich nicht um die neu gebildete Gruppe von Teilchen der Nähe des Magneten, und ersetzen mit dem gleichen Volumen neuer PBST Puffer zu stören.
- Wiederholen Sie die Schritte 6.1 bis 6.4 für Doppelproben und legen diese Proben auf einem Drehrad bei Raumtemperatur für 16 Stunden DNA Hybridisierungszeiten zu untersuchen.
7. TRPS-Setup
- Stecken Sie das Gerät in ein Computersystem mit Software vorhanden.
- Kalibrieren Sie die Anfangsstrecke mit einer Schieblehre.
- Den Abstand zwischen der Außenseite von zwei parallelen Klemmbacken.
- Eingang in die Software, indem Sie die Strecke in der "Strecke" Feld in der "Geräteeinstellungen" Registerkarte und klicken Sie "Kalibrieren Stretch 'unter der Registerkarte.
- Lateral passen ein Polyurethan Nanopore Membran geeigneter Dimensionierung für die Analyse auf die Backen mit der Nanopore-ID-Nummer nach oben. Dann strecken die Backen auf der Strecke benötigt für die Analyse unter Verwendung derstretch Einstellgriff an der Seite des Instruments. Strecken Sie die Backen zwischen 43 und 48 mm.
Hinweis: Der genaue Wert der Strecke entlang der angelegten Spannung bestimmt wird, so dass eine Kalibrierung Partikel Blockaden mindestens 0,3 nA groß sind. Die Strecke ist bereits in die Software in Schritt eingegeben 7.2 und werden automatisch angepasst, wie die Backen gespannt sind. - Legen Sie 80 ul PBST-Puffer in der unteren Fluidzelle unterhalb der Nanopore, um sicherzustellen, gibt es keine Blasen vorhanden, die die Messung beeinflussen können. Wenn es Blasen gesehen sind, zu entfernen und den Puffer zu ersetzen.
- Klicken Sie auf die obere Flüssigkeitszelle in Position und platzieren 40 ul Puffer hinein, wieder zu gewährleisten gibt es keine Blasen vorhanden. Wenn Blasen in der oberen Fluidzelle sind, entfernen Sie sie durch die Flüssigkeit zu ersetzen.
- Wenn eine reproduzierbare Basisstrom hat sich von Ersetzen der oberen Flüssigkeitszelle mit Puffer erreicht, dann werden 40 ul der Probe in die obere Fluidzelle und messen, indem Sie auf "stKunst "in der" Data Acquisition "Tab auf der Software-Bildschirm.
Hinweis: Die Datenerfassung bei einer Frequenz von 50 kHz mit einer Blockade Größenordnung untere Grenze von 0,05 nA abgeschlossen ist, obwohl dies geändert werden kann die Software über die "Analyse Daten" Reiter (unter "Analyseeinstellungen" und "Resistive Sperrungen ') . - Platzieren einen Faradayschen Käfig über die Oberseite des Fluidzellensystems auf elektrische Hintergrundrauschen auf die Messung reduzieren.
- Verwenden Sie einen variablen Druckmodul (VPM) einen Druck oder ein Vakuum auf die Proben anzuwenden.
- Um sich ein äußerer Druck die Düse in die obere Fluidzelle verbinden, drehen Sie dann den Druck Arm und einrasten (je nachdem, ob ein positiver Druck (PRE) oder ein Vakuum (VAC) wird angewendet werden).
- Wenden Sie Druck in einem "cm" oder "mm" Skala der Druckstufe Knopf auf der Oberseite des VPM gelegen verwenden. Drücken Sie den Knopf nach unten Druck auf die 'cm' Maßstab anwenden und ziehen Sie sie nach oben to gelten Druck auf die "mm" Skala.
8. Vorbereiten von Beispielen für TRPS Analyse
- Vortex Proben 30 sec und beschallen für 2 min bei 80 Watt vor TRPS Analyse.
9. Kalibrieren des Nanopore für Zeta-Analyse
- 40 ul Eichpartikel (1x10 10 Teilchen / ml) in die obere Fluidzelle Nach der Platzierung, füllen Sie eine TRPS Messung (Setup wie in Kapitel 7) bei 3 Spannungen angelegt. Ändern Sie die Spannung, die durch einen Klick auf das "+" und "-" Tasten auf der Spannungsskala in der Registerkarte "Geräteeinstellungen" auf der Software.
- Überprüfen Sie, ob die drei Spannungen Hintergrund Ströme von ca. 140, 110 und 80 nA zurück. Sicherzustellen, dass die Kalibrierungs Partikel erzeugen eine durchschnittliche Blockade Größenordnung von mindestens 0,3 nA an der Mittelspannung.
- Einen Druck so die durchschnittliche Halbwertsbreite (FWHM) Dauer der Kalibrierungs Partikel zumindest0,15 ms. Tun Sie dies manuell den Druckarm mit auf den variablen Druckmodul befestigt. Wählen Sie den Druck (PRE) oder Vakuum (VAC) durch Drehen des Armes, bis es in der gewünschten Position einrastet und entsprechend Anweisungen in Schritt 7.8.2 einrichten gelten nach. Sobald diese Bedingungen erreicht wurden, den Lauf starten, indem Sie "Start" auf der Software in der "Data Acquisition" Registerkarte klicken.
- Füllen Sie den Lauf von "Stop" in der "Data Acquisition" Tab drücken, wenn mindestens 500 Teilchen gemessen wurden (siehe "Particle Count 'am unteren Rand der Software-Bildschirm während der Messung) und der Lauf wurde 30 Sekunden überschritten wird (siehe' Run Time 'auch in Richtung der Unterseite des Bildschirms).
- Kalibrieren Sie das System durch einen Kalibrierungslauf abgeschlossen, wie jedes Mal, wenn ein neues Nanopore beschrieben eingeführt wird oder für jeden neuen Tag der Analyse von 9,1-9,4 Schritt vervollständigt.
10. eine Probe Lauf
- Führen Sie die Proben mit der höchsten oderzweithöchste Spannung wie die Kalibrierungsproben auf einem ähnlichen (± 10 nA) gewährleistet, wenn sie nicht gleich sind, Basisstrom.
- Sobald die geeignete Basisstrom erreicht wird, ersetzen Sie den Elektrolyt in der oberen Fluidzelle mit 40 ul der Probe. Wenn eine Probe eingeführt wird, Blockaden auf der Signalspur zu sehen. Starten Sie das Beispiel ausführen, indem Sie auf "Start" in der "Data Acquisition" Tab und notieren Sie mindestens 500 Teilchen (check 'Particle Count' unter der Signalspur befindet) und sorgen für die Laufzeit mindestens 30 sec ist (siehe 'Run Time 'auch unterhalb der Signalspur befindet).
- Um die Messung abgeschlossen ist, klicken Sie auf "Stopp" in der "Data Acquisition Registerkarte 'und die Datendatei speichern.
- die Datei, geben Sie die Dateiinformationen in folgendem Format zu speichern; "Untersuchung" ist der Ordner, die Datei gespeichert werden soll in "Nanopore ID" ist die Seriennummer der Pore verwendet wird, 'Part #' is die Art der Poren (dh NP150 / NP200), "Proben - ID" der Name der Probe vorhanden ist, "Kalibrierung oder Probe" Details , ob es sich um eine Kalibrierung oder Probenmessung ist, "Verwässerung" wird verwendet, wenn die Probe wurde verdünnt ( Typ 100, wenn die Probe verdünnt wurde 100-fach), "Druck" ist der ausgeübte Druck auf die Probe (in cm - siehe Abschnitt 7.8), 'Electrolyte-ID' ist der Name des Puffers wird die Probe aus in und ' Hinweise 'sind jegliche persönliche Notizen über die Probe oder laufen.
- Zwischen jedem Probelauf, waschen Sie das System mit 40 ul PBST Platzierung mehrmals Puffer in die obere Fluidzelle und verschiedene Drücke (in der Regel bei -10, -5 cm (Vakuum), und 5 und 10 cm (Überdruck)) Aufbringen bis nicht mehr Blockade Ereignisse vorhanden sind, um sicherzustellen, gibt es im System verbleiben keine Restpartikel sind und daher keine Kreuzkontamination zwischen den Proben. Führen Proben in dreifacher Ausfertigung mit diesem Waschschritt zwischen jeder Wiederholung Probe abgeschlossen als wel laufenl zwischen verschiedenen Proben.
Ergebnisse
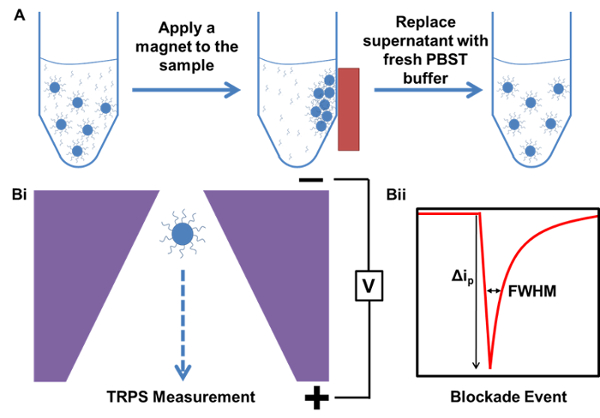
Abbildung 1 : Schematische Darstellung der Prozesse der magnetischen Reinigung und einer TRPS Messung. A) Beispiel für magnetische Reinigung von Probe mit einer Probe ausgehend enthält überschüssige, nicht gebundene Einfang - Sonde DNA. B) trpS Meßbeispiel i) Partikel durch die Nanopore vorbei und ii) Blockade Ereignis von Teilchen vorübergehend verschließenden Ionen in der Pore erzeugt eine vorübergehende Abnahme verursacht in Strom; Informationen aus der verwendet wird , Teilchen Translokation Geschwindigkeiten zu berechnen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Die Entfernung von überschüssigem DNA, die nicht aus den Proben, um die Partikel Oberfläche gebunden hat, ist wichtig, vor der TRPS Analyse als nicht zu melden"Falsch-positiven" Ergebnisse. Die Fähigkeit , einen Magneten zu verwenden , um die SPPs extrahieren und waschen ist ein großer Vorteil für TRPS (1A). 1B beschreibt ein einfaches Beispiel einer TRPS Messung und ein Beispiel "Blockade Ereignis" als Partikel erreicht durchquert die Pore. Erstens haben wir gezeigt, dass TRPS ein Hochdurchsatz-Verfahren ist, die zwischen den Proben von ähnlicher Größe, aber einer wesentlich unterschiedlichen Ladungs unterscheiden kann. Seine Fähigkeit , sowohl die Größe und Ladung Analyse zu vervollständigen gleichzeitig in einer einzigen Messung ist in Abbildung 2 zu sehen ist. Abbildung 2 ist ein Beispiel für eine) Größe und b) Zeta - Potential - Analyse von Streptavidin - beschichteten Teilchen ohne Modifikationen (hellrosa Datensatz) und Streptavidin beschichtete Teilchen mit Einzelstrang-DNA auf der Oberfläche gesättigt (blau Datensatz). Obwohl beide Proben eine ähnliche Größe waren, wurde das Zetapotential signifikant unterschiedliche und viel größer als DNA functi waronalized auf die Oberfläche des Partikels.

Abbildung 2. Größe und Zeta - Potential - Analyse von DNA-modifizierten und unmodifizierten Streptavidin beschichtete Nanopartikel. Die rosa Balken stellen unmodifizierte Streptavidin beschichteten Teilchen und die blauen Balken repräsentieren DNA-modifizierten Partikel. A) Frequenz (%) versus Partikeldurchmesser (nm). B) Frequenz (%) vs Zetapotential (mV). Abbildung angepasst von Zusatzdaten in Blundell et al. 19. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Nicht nur kann die Technik unterscheiden zwischen Teilchen unmodifizierten und modifizierten DNA kann TRPS auch zwischen Proben mit verschiedenen Konzentrationen von DNA unterscheiden zum par hybridisiertentikel Oberfläche. Abbildung 3 zeigt die Größe und Zeta - Potential für Proben zeigten Daten mit der niedrigsten (10 nM, hellgrün Datensatz) und höchsten (210 nM, blau Datensatz) Konzentrationen von DNA an die Streptavidin - beschichteten Partikeln hybridisiert. Eine größere Zeta-Potential-Wert wird für Partikel aufgezeichnet mit einer höheren Konzentration an DNA hybridisiert.

Abbildung 3. Gleichzeitige Größe und Zetapotential Daten aus einer einzigen TRPS Messung erfasst. Die blauen Balken / Datenpunkte repräsentativ sind von Streptavidin-beschichteten Teilchen mit 210 nM CP-DNA hybridisiert und die hellgrünen Balken / Datenpunkte repräsentieren Streptavidin beschichtete Teilchen mit 10 nM CP-DNA hybridisiert. Figur angepasst von Blundell et al. 19. Bitte klicken Sie hier um zu seheneine größere Version dieser Figur.
Es ist sinnvoll , dass jeder Datenpunkt in dem Streudiagramm zu beachten , ein einzelnes Partikel unter einer Probenpopulation darstellt, so dass für in der Tiefe Partikel-by-Partikelanalyse mit jeder Probe. Abbildung 4 die Technik des unterstützt Wirksamkeit bei geringen Änderungen in der DNA - Struktur zu bestimmen ( einsträngige und doppelsträngige DNA), sowie Unterschiede in den Proben mit Ziel - DNA von der gleichen Größe, aber gebunden in einen anderen Bereich der Einfangsonde den Nachweis der hohen Empfindlichkeit zu identifizieren (dh Mittel Bindung und End verbindliche Ziele gezeigt in Abbildung 4). Die Teilchen in Figur 4 gezeigt sind diejenigen mit den folgenden Oberflächenmodifikationen, von links nach rechts; Fänger-Sonde (CP) DNA nur, CP und einer vollständig komplementären DNA-Target, CP und eine mittlere Bindung DNA-Target, CP und ein Ende bindende DNA-Target, CP und eine überhängende DNA-Ziel.

. Figur 4. Relative Veränderung des Zetapotential gemessen für DNA-modifizierten Teilchen mit einer Reihe von DNA - Targets ändern in Zeta - Potential, mV, aus i) CP Teilchen auf einen Bereich von DNA - Targets funktionalisiert; ii) vollständig komplementär, iii) Mittelbindung iv) End Bindung, v) Surplombant Ziel. Die Fehlerbalken stellen die Standardabweichung, wobei n = 3. Abbildung von Blundell angepasst et al. 19. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Diskussion
Die Berechnung für das Zeta - Potential verwendet , um eine Kalibrierung basierte Methode verwandt von Arjmandi arbeiten et al. 21. Die Dauer der Translokation von Teilchen, wie sie eine Nanopore durchqueren, als eine Funktion der angelegten Spannung gemessen wird, eine mittlere elektrische Feld und Partikelgeschwindigkeiten über die Gesamtheit eines regelmäßigen konischen Pore verwenden. Die elektrophoretische Mobilität ist die Ableitung von 1 / T in Bezug auf die Spannung (wobei T die Dauer Blockade ist), durch das Quadrat der Erfassungszonenlänge multipliziert, l. Durchschnittsgeschwindigkeiten an mehreren Referenzpunkten durch die Sensorzone gemessen werden für minimale Fehler bei der Berechnung Zetapotential mit dieser Methode zu ermöglichen.
Die Kalibrierung der Poren basiert auf der Linearität von 1 / T vs Spannung, V, an jedem Referenzpunkt in der Sensorzone. Die elektroPartikelGeschwindigkeiten von Eich- und Probenpartikel,upload / 54577 / 54577eq2.jpg "/> und  jeweils auf ihre Zeta-Potentiale im Zusammenhang,
jeweils auf ihre Zeta-Potentiale im Zusammenhang,  und
und  Wie in Gleichung 1, unter der Annahme einer linearen Beziehung zwischen den beiden wie 12,20 in der Smoluchowski Näherung dargestellt. Die Netto-Zeta-Potential-Werte sowohl für die Kalibrierung und Probe sind die Unterschiede in der Partikel Zeta-Potential und die Membran Zeta-Potential,
Wie in Gleichung 1, unter der Annahme einer linearen Beziehung zwischen den beiden wie 12,20 in der Smoluchowski Näherung dargestellt. Die Netto-Zeta-Potential-Werte sowohl für die Kalibrierung und Probe sind die Unterschiede in der Partikel Zeta-Potential und die Membran Zeta-Potential,  . Verwendung Strömungspotentialverfahren 12,18 als -11 mV in PBS für diese Studie das Zeta - Potential des Polyurethans Pore gemessen.
. Verwendung Strömungspotentialverfahren 12,18 als -11 mV in PBS für diese Studie das Zeta - Potential des Polyurethans Pore gemessen.
 (1)
(1)
Das Zeta - Potential der einzelnen Teilchen, i,  , Wird aus den jeweiligen Zeta-Potentiale gemessen bei verschiedenen Referenzpunkten innerhalb der Pore berechnet (Gleichung 2), wobei
, Wird aus den jeweiligen Zeta-Potentiale gemessen bei verschiedenen Referenzpunkten innerhalb der Pore berechnet (Gleichung 2), wobei 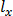 ist die Position des Partikels innerhalb der Pore nach der Zeit t = T x, und
ist die Position des Partikels innerhalb der Pore nach der Zeit t = T x, und  ist die Partikelgeschwindigkeit einzelner Probenteilchens i bei einer relativen Positionen l x;
ist die Partikelgeschwindigkeit einzelner Probenteilchens i bei einer relativen Positionen l x;  .
. 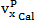 , P und V sind elektroGeschwindigkeit pro Einheit Spannung, Konvektionsgeschwindigkeit pro Einheit Druck, ausgeübten Druck und Spannung für die Probe jeweils ausgeführt wird , kann eine vollständige Ableitung dieser Gleichung in der Arbeit von Blundell et al. 19.
, P und V sind elektroGeschwindigkeit pro Einheit Spannung, Konvektionsgeschwindigkeit pro Einheit Druck, ausgeübten Druck und Spannung für die Probe jeweils ausgeführt wird , kann eine vollständige Ableitung dieser Gleichung in der Arbeit von Blundell et al. 19.
 (2)
(2)
Wenn die Fangsonde DNA an die Streptavidin-beschichteten Nanopartikel bindet, ist es wichtig, dass die Forscher in Lösung überschüssige, nicht gebundene Einfang-Sonde DNA links entfernt. Dies geschieht ganz einfach den SPPs und einen einfachen Magneten mit zu einer schnellen und einfachen Austausch des Überstandes mit neuen PBST-Puffer. Wenn überschüssige capture DNA in Lösung belassen wird, und Ziel-DNA zugegeben, wobei die Ziel-DNA in die freie capture DNA in Lösung binden kann, und nicht als die auf der Oberfläche SPP. Eine Änderung der Partikelgeschwindigkeit und Zeta-Potential wird nur dann beobachtet werden, wenn die Ziel-DNA auf der Partikeloberfläche an die Fangsonde vorhanden bindet.
Analyse und den Vergleich einer großen Anzahl von Proben über viele Tage TRPS Verwendung kann die Verwendung von mehr als einer Porenmembran erfordern. Einige Poren einige kleinere Unterschiede in ihrer Größe haben können aufgrund des Herstellungsprozesses und in diesen Fällen muss der Benutzer den Basisstrom sicherzustellen bleibt identisch in allen runs. Wenn die gleiche Basisstrom beobachtet wird, die erhaltenen Ergebnisse sind vergleichbar zwischen den Poren. Sobald die Basislinie die gleiche wie vorhergehende Durchläufe ist, ist es zwingend notwendig, dass der Benutzer die Dehnungs unverändert zwischen Kalibrier- und Probenläufen hält für eine genaue Bestimmung der Partikel Translokation Geschwindigkeiten zu ermöglichen, da sie die Poren passieren.
Die TRPS-Technologie hat eine relativ einfache Einrichtung, die einfach und schnell während eines Experiments zerlegt werden kann. Wenn Beheben von Problemen kann dies der Prozess sehr viel einfacher machen. Zum Beispiel ist es wichtig, keine Blasen in der unteren Fluidzelle oder oberen Flüssigkeitszelle zu ermöglichen, wenn die Analyse Bezeichnung. Dies wird zu einer instabilen Basisstrom führen. Wenn Blasen in der oberen Fluidzelle sind, kann die Probe entfernt und ersetzt werden. Wenn Blasen in der unteren Fluid Zelle erscheinen, sollte der Puffer mit frischem Puffer entfernt und ersetzt werden. Wenn die Blasen ein hartnäckiges Problem sind, dann kann es zu viel Tensidin der Lösung , so kann dies 16 reduziert werden müssen (wir verwenden nur 0,05% Tween-20). Einige Proben können die Poren blockieren, wenn ihre Größe überschreitet, die Porengröße oder wenn die Konzentration der Probe zu hoch ist. Um dies zu beheben, kann die Porengröße durch Erhöhen der Dehnung oder der Probe erhöht werden kann auf eine niedrigere Partikelkonzentration 16 verdünnt werden. Für Einzelpartikelanalyse kann die Probe auch die Poren blockieren, wenn es eine Menge große Aggregate vorhanden sind, ist es wichtig, Wirbel und beschallen die Probe, bevor es durch TRPS läuft.
Unter anderen Verfahren hat TRPS verschiedene Vorteile, einschließlich der Fähigkeit, Größe und Ladungsmessungen einzelner Teilchen zu vervollständigen gleichzeitig; so dass für multimodale Proben effektiv analysiert unter Verwendung dieser Methode werden. Ein Vorteil ist , das Signal / Blockaden erzeugt wird, kann durch einfaches in Minuten für eine bestimmte Probe optimiert werden Ändern der Dehnung und Spannung eine Blockade Größenordnung zu erhalten, Δ </ Em> 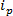 Deutlich größer als das Hintergrundrauschen (Blockaden sind nA Maßstab im Vergleich zum Hintergrundrauschen <10 pA). Die Möglichkeit, die Strecke von der Pore macht das Verfahren vielseitiger über Festkörperporen Techniken zu verändern als die Porengröße in Bezug auf die Größe des Analyten in Frage eingestellt werden; besonders nützlich, wenn die Größen in Analytbindungspartners Effekte wie Aggregation und DNA-Protein-Untersuchung ergeben können die ursprüngliche Festkörperporengrößenbereich zu überschreiten. Ein weiterer vorteilhafter Aspekt der TRPS ist das Niveau der Empfindlichkeit von der Technik. Die Fähigkeit, feine Unterschiede in DNA zu detektieren Bindung (wobei die gleiche Menge an DNA hinzugefügt wurde (gleiche Menge an zugesetztem Ladungs) und die Proben werden von der gleichen Größe), basierend auf der Position der Ziel-DNA-Bindung ist sehr tiefe in diesem Bereich der Analyse und wird für zukünftige Nanopartikel-Test-Design-Plattformen von großem Nutzen sein. Jede subtil unterscheidenrenz kann mit einem Partikel-by-Teilchen Natur der TRPS Technologie erkannt und isoliert werden. Diese Analyse übertrifft die von ensemble Techniken wie dynamische Lichtstreuung oder Photonenkorrelationsspektroskopie , die lediglich ein Durchschnitt der Probenpopulation wird gage analysiert und kann in den Fällen von Proben multimodal 6,7 nicht differenzieren.
Deutlich größer als das Hintergrundrauschen (Blockaden sind nA Maßstab im Vergleich zum Hintergrundrauschen <10 pA). Die Möglichkeit, die Strecke von der Pore macht das Verfahren vielseitiger über Festkörperporen Techniken zu verändern als die Porengröße in Bezug auf die Größe des Analyten in Frage eingestellt werden; besonders nützlich, wenn die Größen in Analytbindungspartners Effekte wie Aggregation und DNA-Protein-Untersuchung ergeben können die ursprüngliche Festkörperporengrößenbereich zu überschreiten. Ein weiterer vorteilhafter Aspekt der TRPS ist das Niveau der Empfindlichkeit von der Technik. Die Fähigkeit, feine Unterschiede in DNA zu detektieren Bindung (wobei die gleiche Menge an DNA hinzugefügt wurde (gleiche Menge an zugesetztem Ladungs) und die Proben werden von der gleichen Größe), basierend auf der Position der Ziel-DNA-Bindung ist sehr tiefe in diesem Bereich der Analyse und wird für zukünftige Nanopartikel-Test-Design-Plattformen von großem Nutzen sein. Jede subtil unterscheidenrenz kann mit einem Partikel-by-Teilchen Natur der TRPS Technologie erkannt und isoliert werden. Diese Analyse übertrifft die von ensemble Techniken wie dynamische Lichtstreuung oder Photonenkorrelationsspektroskopie , die lediglich ein Durchschnitt der Probenpopulation wird gage analysiert und kann in den Fällen von Proben multimodal 6,7 nicht differenzieren.
Kleine Solid-State - Nanoporen (100-200 nm) wurden ebenfalls Teilchendynamik verwendet worden , um zu überwachen und haben festgestellt , dass Teilchenbeweglichkeit beeinflusst werden können , wenn der Durchmesser des Teilchens , dass der Nanopore 22,23 zu nähern beginnt. Nanopores viel größer als die Partikel analysiert werden (wie in dieser Untersuchung verwendet) haben einen geringeren Effekt auf die Partikelmobilität und somit innerhalb der Pore die Translokation Dynamik. Die Poren in dieser Studie verwendet werden, um ihre Analyten Größenbereiche jedoch begrenzt, hat eine NP150 beispielsweise einen Größenbereich von 60 bis 480 nm so, wenn eine multimodale Probe aus Teilchen innerhalb und überschreitet dieseGrenze, können sie nicht auf der gleichen Poren als Poren analysiert werden kann dann blockiert werden. Es ist auch wichtig zu beachten, dass eine bimodale Probe gemessen, die 60 und 480 nm-Partikel (die an der absoluten unteren und oberen Grenzen der Pore), zum Beispiel, werden unterschiedliche Dehnungs- und Spannungsbedingungen erfordern, obwohl sowohl innerhalb der Größenanalyse Bereich sind der Pore. Dies ist, weil die für die größeren Partikel erforderliche Dehnung in den kleineren Partikeln führen wird eine besonders kleine Blockade Größe hat (basierend auf dem reduzierten Widerstand), die als Hintergrundrauschen angesehen werden könnte und daher nicht notwendigerweise während eines Probelaufs gemessen.
Blasen können ein Problem mit den Probenmessungen sein, wie in der unteren oder oberen Flüssigkeitszelle Blasen eine instabile Basisstrom erzeugen, auf die Probendurchläufe nicht abgeschlossen werden kann. Elektrolyte einer Brause Natur (einige hochkonzentrierten biologischen Medien, zum Beispiel) kann schwierig sein, Proben zu laufen und damit erfuiring Suspension kann in diesen spezifischen Medien als problematisch erweisen. Die meisten Proben kann jedoch sein, stark verdünnt oder in alternative Puffer vor der TRPS Analyse suspendiert.
Das Verfahren ist anpassungsfähig und kann verwendet werden , um eine Reihe von Nanopartikel-Analyten zu analysieren, einschließlich der Analyse von Proteinen, DNA, kleine Moleküle, Aggregationsassays 17,24 und biologisch relevante Partikel. Die Vielseitigkeit der TRPS in eine breite Palette von Analyten zu charakterisieren zeigt die Techniken Potential in einer Reihe von Bereichen wie Drug - Delivery - 1,25, Biosensorik 26-28 und Umweltprüfungen.
Offenlegungen
ELCJB wird von Izon Science Ltd. unterstützt
Danksagungen
Die Autoren danken Izon Science Ltd für ihre Unterstützung. Die Arbeit wurde von der Europäischen Kommission für Forschung (PCIG11-GA-2012-321836 Nano4Bio) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Phosphate buffered Saline (PBS) | Sigma Aldrich, UK | P4417 | 1 tablet dissolved in 200 mL deionised water to make buffer solution. |
| Tween-20 | Sigma Aldrich, UK | P1379 | 0.05% (v/v) in PBS buffer as a surfactant |
| Carboxyl polystyrene nanoparticles | Bangs Laboratories, US | CPC200 | Nominal diamter of 220 nm, raw concentration of 1E12 particles/mL, specific surface charge of 86 µeq/g (equivalent to a surface charge density of 3.2E19 C/nm^2. |
| Streptavidin coated nanoparticles | Ademtech, France | 3121 | Batch had binding capacity of 4352 pmol/mg (188 nM theoretical DNA binding capacity) at a raw concentration of 1.1E11 particles/mL. |
| Biotinylated oligonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Biotin modification at 3' end; Lyophilised powders reconstituted to 100 µM using deionised water, and diluted as required. Sequences: CP 5'ATGGTTAAACCTCAC TACGCGTGGC[Btn]3' |
| Standard olignonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Lyophilised powders reconstituted to 100 µM using deionised water, and diluted as required. Sequences of DNA targets: Fully complementary - 5'GCCACGCGTAGTGAGGTTTAACCAT3', Middle binding - 5'GTAGTGAGGT3', End binding - 5'GTTTAACCAT3', Partially complementary overhanging - 5'GTGAGGTTTAACCAT TTTTTTTTTTTTTTT3'. |
| Izon qNano | Izon Science, NZ | Inherent pressure on system of 47 Pa, | |
| Izon Variable Pressure Module (VPM) | Izon Science, NZ | Each 'cm' of pressure is equivalent to approximately 1000 Pa. | |
| Polyurethane nanopore membranes | Izon Science, NZ | NP150 | Analyte size range 60-480 nm, pore diameter of calculated to be 799 nm at a 45 mm stretch. |
| Magrack 6 | GE Healthcare, UK | 28-9489-64 | |
| Sonic Bath | Fisher Scientific, UK | 10692353 | 80 Watts |
| Vortexer | IKA, Germany | 0003365000 | |
| Rotary Wheel | Labnet International, US | H5500-230 V |
Referenzen
- Alexander, C. M., Maye, M. M., Dabrowiak, J. C. DNA-capped nanoparticles designed for doxorubicin drug delivery. Chem Commun. 47 (12), 3418-3420 (2011).
- Billinge, E. R., Platt, M. Aptamer based dispersion assay using tunable resistive pulse sensing (TRPS). Anal Methods. 7 (20), 8534-8538 (2015).
- Bulyk, M. L. Protein Binding Microarrays for the Characterization of Protein-DNA Interactions. Adv Biochem Eng Biotechnol. 104, 65-85 (2007).
- Platt, M., Rowe, W., Knowles, J., Day, P. J., Kell, D. B. Analysis of aptamer sequence activity relationships. Integr Biol. 1 (1), 116-122 (2009).
- Ruiz-Hernández, E., Baeza, A., Vallet-Regí, M. Smart Drug Delivery through DNA/Magnetic Nanoparticle Gates. ACS Nano. 5 (2), 1259-1266 (2011).
- Murdock, R. C., Braydich-stolle, L., Schrand, A. M., Schlager, J. J., Hussain, S. M. Characterization of Nanomaterial Dispersion in Solution Prior to In Vitro Exposure Using Dynamic Light Scattering Technique. Toxicol Sci. 101 (2), 239-253 (2008).
- Hupfield, S., Holsaeter, A. M., Skar, M., Frantzen, C. B., Brandl, M. Liposome size analysis by dynamic/static light scattering upon size exclusion-/field flow fractionation. J Nanosci Nanotechnol. 6 (7), 3025-3031 (2006).
- Roberts, G. S., et al. Tunable pores for measuring concentrations of synthetic and biological nanoparticle dispersions. Biosens Bioelectron. 31 (1), 17-25 (2012).
- Roberts, G. S., Kozak, D., Anderson, W., Broom, M. F., Vogel, R., Trau, M. Tunable nano/micropores for particle detection and discrimination: scanning ion occlusion spectroscopy. Small. 6 (23), 2653-2658 (2010).
- Vogel, R., et al. Quantitative sizing of nano/microparticles with a tunable elastomeric pore sensor. Anal Chem. 83 (9), 3499-3506 (2011).
- Booth, M. A., Vogel, R., Curran, J. M., Harbison, S., Travas-Sejdic, J. Detection of target-probe oligonucleotide hybridization using synthetic nanopore resistive pulse sensing. Biosens Bioelectron. 45, 136-140 (2013).
- Kozak, D., Anderson, W., Vogel, R., Chen, S. Simultaneous size and ζ-potential measurements of individual nanoparticles in dispersion using size-tunable pore sensors. ACS Nano. 6 (8), 6990-6997 (2012).
- Kozak, D., Anderson, W., Vogel, R., Trau, M. Advances in Resistive Pulse Sensors: Devices bridging the void between molecular and microscopic detection. Nano Today. 6 (5), 531-545 (2011).
- Weatherall, E., Willmott, G. R. Applications of tunable resistive pulse sensing. Analyst. 140, 3318-3334 (2015).
- Willmott, G. R., et al. Use of tunable nanopore blockade rates to investigate colloidal dispersions. J Phys Condens Matter. 22 (45), 454116 (2010).
- Blundell, E. L. C. J., Mayne, L. J., Billinge, E. R., Platt, M. Emergence of tunable resistive pulse sensing as a biosensor. Anal Methods. 7, 7055-7066 (2015).
- Platt, M., Willmott, G. R., Lee, G. U. Resistive Pulse Sensing of Analyte-Induced Multicomponent Rod Aggregation Using Tunable Pores. Small. 8 (15), 2436-2444 (2012).
- Vogel, R., Anderson, W., Eldridge, J., Glossop, B., Willmott, G. A variable pressure method for characterizing nanoparticle surface charge using pore sensors. Anal Chem. 84 (7), 3125-3131 (2012).
- Blundell, E. L. C. J., Vogel, R., Platt, M. Particle-by-Particle Charge Analysis of DNA-Modified Nanoparticles Using Tunable Resistive Pulse Sensing. Langmuir. 32 (4), (2016).
- Hunter, R. J. . Zeta Potential in Colloid Science: Principles and Applications. , (1981).
- Arjmandi, N., Van Roy, W., Lagae, L., Borghs, G. Measuring the electric charge and zeta potential of nanometer-sized objects using pyramidal-shaped nanopores. Anal Chem. 84 (20), 8490-8496 (2012).
- Bacri, L., et al. Dynamics of colloids in single solid-state nanopores. J Phys Chem B. 115 (12), 2890-2898 (2011).
- Cabello-Aguilar, S., et al. Dynamics of polymer nanoparticles through a single artificial nanopore with a high-aspect-ratio. Soft Matter. 10 (42), 8413-8419 (2014).
- Billinge, E. R., Muzard, J., Platt, M. Tunable resistive pulse sensing as a tool to monitor analyte induced particle aggregation. Nanomater Nanosci. 1 (1), 11 (2013).
- Li, J., Fan, C., Pei, H., Shi, J., Huang, Q. Smart Drug Delivery Nanocarriers with Self-Assembled DNA Nanostructures. Adv Mater. 25 (32), 4386-4396 (2013).
- Billinge, E. R., Broom, M., Platt, M. Monitoring aptamer-protein interactions using tunable resistive pulse sensing. Anal Chem. 86 (2), 1030-1037 (2014).
- Gold, L., et al. Aptamer-Based Multiplexed Proteomic Technology for Biomarker Discovery. PLoS One. 5 (12), e15004 (2010).
- Park, S. -. J., Taton, T. A., Mirkin, C. A. Array-Based Electrical Detection of DNA with Nanoparticle Probes. Science. 295 (5559), 1503-1506 (2002).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten