Method Article
Identifizierung von Protein-Interaktionspartner in Säugetierzellen mit SILAC-Immunpräzipitation Quantitative Proteomics
In diesem Artikel
Zusammenfassung
SILAC Immunpräzipitationsexperimenten stellt ein wirksames Mittel zur Entdeckung neuer Protein-Protein-Interaktionen. Dadurch, dass die genaue relative Quantifizierung von Protein Hülle und Fülle in den Kontroll-und Testproben, true Wechselwirkungen können leicht von experimentellen Verunreinigungen unterschieden werden, und niedriger Affinität Wechselwirkungen durch die Verwendung von weniger strengen Pufferbedingungen erhalten.
Zusammenfassung
Quantitative Proteomik in Kombination mit Immun-Affinitätsreinigung, SILAC Immunpräzipitation, stellt ein wirksames Mittel zur Entdeckung von neuen Protein-Protein-Interaktionen. Dadurch, dass die genaue relative Quantifizierung der Proteinmenge in den Kontroll-und Testproben, mag wahr Wechselwirkungen leicht von experimentellen Verunreinigungen unterschieden werden. Geringe Affinität Wechselwirkungen können durch die Verwendung von weniger strengen Pufferbedingungen beibehalten werden und bleiben leicht identifizierbar. Dieses Protokoll beschreibt die Markierung von Gewebekulturzellen, die mit stabilen Isotopen markierten Aminosäuren, Transfektion und Immunpräzipitation eines affinitätsmarkierten Proteins von Interesse, gefolgt von der Herstellung bis zur Vorlage einer Massenspektrometrie Möglichkeiten. Dieses Protokoll beschreibt, wie dann zu analysieren und zu interpretieren, die aus dem Massenspektrometer, um die zellulären Partnern interagiert mit einem Protein von Interesse zu identifizieren zurückgegebenen Daten. Als ein Beispiel dieser Technik wird angewendet, um Identifizieren Proteine die Bindung an die eukaryontischen Translationsinitiationsfaktoren: eIF4AI und eIF4AII.
Einleitung
Ein wesentlicher Schritt zum Verständnis der Proteinfunktion ist die Identifizierung von interagierenden Proteinen relevant. Wo solche Proteine sind bekannt gibt es eine Reihe von Techniken zur Verfügung, die jeweils ihre eigenen Vorzüge und Nachteile. Dazu gehören das Hefe-Zwei-Hybrid-System, Pulldown-Assays mit rekombinanten Proteins sowie Tandemaffinitätsreinigung oder TAP-Tagging 1, 2.
Eine neuere Zusätzlich zu diesen Techniken ist die Kombination von Affinitätsreinigung des Proteins von Interesse aus einer entsprechenden Säugerzelllinie, gefolgt von der quantitativen Massenspektrometrie stabile Isotopenmarkierung von Aminosäuren in Zellkultur (SILAC) 3. Dies hat Vorteile gegenüber dem Hefe-Zwei-Hybrid-Ansatz in dieser Zelle Lokalisierung und post-translationale Modifikationen werden nicht gestört, als auch Vorteile gegenüber traditionellen TAP-Tagging, dass es eine quantitative statt qualitative Ansatz ermöglicht dem Benutzer, Grund istdily unterscheiden nicht-spezifisch interagierenden Proteinen und Verunreinigungen von Wirtsfaktoren, die spezifisch binden. Ferner kann als eine Probe analysiert typischerweise aus und nicht als einzelne Proteinbanden, interessierende Proteine nicht durch Migration in ähnlicher Proteine auf einem Gel, noch müssen sie in der Regel vorhanden sein in ausreichenden Mengen nach der Färbung sichtbar zu sein, was zu maskiert erhöhte Anzahl von vertrauens 4 identifizierten Proteine.
Um diese Technik zu demonstrieren, GFP-Fusionen der nahe verwandten eukaryontischen Translationsinitiationsfaktor eIF4AI und eIF4AII, dass der Anteil über 90% Aminosäureidentität wurden von SILAC-Immunpräzipitation quantitative Proteomik untersucht. Menschliche eIF4AI und II wurden in pEGFP-C1 kloniert, um ein Fusionsprotein, wo GFP an den N-Terminus von eIF4A fusioniert bilden. Um zu vermeiden, die Notwendigkeit zur Erzeugung von stabilen Zelllinien transiente Transfektion wurde verwendet, um diese Konstrukte zu stabilen Isotop markiert 293T-Zellen zu liefern.
Zellen wurden zunächst für zwei Wochen in SILAC Zellkulturmedium, gefolgt von der Transfektion von Plasmid-DNA, die ein Protein von Interesse kodiert, bezeichnet. Zellen wurden dann lysiert, die Proteinkonzentration normalisiert, und gleiche Mengen von Lysat Affinität an Anti-GFP-Agarose gereinigt. Gleiche Mengen Eluat wurden dann kombiniert und für LC-MS/MS Analyse unterzogen. Die Ergebnisse dieser Analyse werden dann verarbeitet, um ein hohes Vertrauen Protein zu identifizieren: Protein-Wechselwirkungen (Fig. 1).
SILAC Immunpräzipitation ermöglicht die Identifizierung nicht nur direkte Interaktionen, sondern auch geringe Affinität oder indirekten Wechselwirkungen mit Proteinkomplexen 4. Mit diesem System erlaubt eIF4AI und II Immunopräzipitationen reproduzierbare und zuversichtlich Identifizierung der primären Bindungspartner eIF4G (Isoformen I / II und III) 5, als auch indirekte Interaktionen mit eIF4E und zahlreiche Komponenten des eIF3 komplex.
Protokoll
1. Generierung und Passagierung von SILAC-markierten Zelllinien
Hinweis: die Verwendung von Trypsin-EDTA in allen Phasen der Passagierung und Herstellung von Versuchsproben für eine Analyse zu vermeiden, da das Trypsin kann unmarkierten Aminosäuren, die zu einer unvollständigen Markierung von Proben führen würde, enthalten kann.
- Um eine Flasche SILAC Medien vorzubereiten fügen Sie ein 0,5-ml-Aliquot einer entsprechend SILAC-markiertem Arginin (84 mg / ml in PBS) und Lysin (146 mg / ml in PBS) in einen 500-ml-Flasche von Arg / Lys DMEM (mit L-Glutamin).
- Als nächstes werden 50 ml dialysiert FBS und 5 ml Penicillin / Streptomycin in den 500-ml-Flasche.
Hinweis: HEK 293T-Zellen (ATCC) sollten in DMEM-Medium gehalten werden fehlen Arginin und Lysin und mit Licht (R0K0) ergänzt, mittel (R6K4) oder schwere (R10K8) Aminosäuren, dialysiert fetales Rinderserum (10 kDa Cutoff) und Penicillin / Streptomycin. Zellen müssen in Medien für mindestens 5 Zellteilungen gehalten werden, um sicherzustellen, vollständigeKennzeichnung. In den meisten Fällen werden Zellen leicht in ≥ 2 Wochen markiert. - So teilen Zellen für die Passage sollten die Zellen von der Monoschicht durch Schlagen der Flasche verdrängt werden. Alternativen umfassen die Verwendung von Zellschaber oder durch Verwendung von enzymfreien PBS-basierten Zell-Dissoziationspuffer.
Hinweis: Um Medien zu bewahren, sollten die Zellen in T25-Flaschen passagiert werden und größere Zellzahlen nur unmittelbar vor einem Experiment erzeugt. - 24 Stunden vor der Transfektion von Zellen, Saatgut 3,5 x 10 6 SILAC-markierten Zellen in einem einzigen 10 cm 2 Teller für jeden experimentellen Zustand untersucht.
2. Transfektion von Zellen markiert mit pEGFP-Fusionskonstrukte
- Entfernen Sie die Medien aus den Zellen und ersetzen Sie es mit 9 ml Antibiotika-freien SILAC DMEM (Leicht, Mittel oder Schwer-) Medien.
Hinweis: Antibiotika sollten nicht zu den Medien, da sie mit der Leistungsfähigkeit auf Liposomen basierende Transfektion Reagenzien behindern. - Prepare einer Mischung aus 10 ug der entsprechenden Plasmid (epEGFP-C1, pEGFP-eIF4A-I, pEGFP-eIF4A-II) in 500 ul Antibiotika-freien DMEM SILAC und mischen Sie es mit 500 ul Antibiotika-freien SILAC DMEM mit 10 ul Transfektionsreagenz (z. B. Lipofectamin 2000). Mischen Sie die Reaktion gründlich durch Pipettieren von oben und unten mehrmals.
- Inkubieren der Reaktionsmischung bei Raumtemperatur für 20 Minuten und füge tropfenweise zu dem Zellmonolayer dann. Rock the Platte sanft von Seite zu Seite.
- Die transfizierten Zellen bei 37 ° C und 10% CO 2 für 24 Stunden.
Hinweis: Wenn die Expression eines Proteins von Interesse führt jede offensichtliche Toxizität, dann kann es notwendig sein, die Menge an Plasmid transfiziert und / oder die Dauer des anschließenden Expressionszeit zu reduzieren.
3. Ernten Zelllysaten
- Ernten Sie die Zellen aus der Schüssel in eiskaltes PBS mit einem Zellschaber. Sammle die Zellen durch Zentrifugation bei 220 × g, 476, C für 5 min. In 10 ml eiskaltem PBS Waschen der Zellen eine weitere 3x.
- Zellpellet in 200 ul der Zell-Lyse-Puffer (10 mM TrisCl / pH 7,5, 150 mM NaCl, 0,5 mM EDTA, 0,5% NP40), die frisch zugesetzte Protease-Inhibitor-Cocktail III zu 1x Konzentration und RNase-Cocktail (optional) in 5 ul pro ml.
Anmerkung: Für Proteine mit bekannten Nukleinsäure-Bindungsaktivität, kann es notwendig sein, Nukleasen zu dem Lysat vor der Ausfällung hinzu. In Fällen, in denen Nuklease zugegeben, die Proben auf Eis für 30 min auf Eis inkubiert, mit dem Pipettieren alle 10 min. Extrahieren von Gesamtnukleinsäure aus einer kleinen Fraktion der Probe und Analyse durch Agarose-Gelelektrophorese kann die Wirksamkeit der Nuklease testen. - Zentrifuge Proben bei 13.000 × g, 4 ° C für 10 min unter Beibehaltung der Überstand als lösliche Zelllysat.
- Die Konzentration des Zell-Lysat sollte durch BCA-Assay gemäß den Anweisungen des Herstellers beurteilt.
- Verwenden Lysepuffer Proteaseinhibitorcocktail III Proteinkonzentration in einem Endvolumen von 500 ul zu normalisieren.
- Die Lautstärke auf 1 ml durch Zugabe von 500 &mgr; l Verdünnungspuffer (10 mM TrisCl / pH 7,5, 150 mM NaCl, 0,5 mM EDTA), enthaltend Proteaseinhibitor-Cocktail III bei Schluss 1x Konzentration. Ein 50 ul Aliquot der Probe als Probeneingangs beibehalten werden, und das Lysat auf Eis gehalten, während die Erstellung von Anti-GFP-Perlen (z. B. GFP-Trap).
Anmerkung: In der Regel Ausbeuten variieren zwischen 1-3,5 mg Protein in 1 ml der endgültigen Probe. Während die obigen Puffer eignen sich für viele Proteine von Interesse, für andere kann es nötig sein, Pufferkomponenten zu modifizieren, um sicherzustellen, dass das Köderprotein solubilisiert und Protein-Protein-Wechselwirkungen zu erhalten. Mögliche Änderungen sind die Puffer (Phosphat, HEPES), Salzkonzentration (150-500 mm), die Wahl des Waschmittels oder andere Zusatzstoffe.
4. Bindung an Anti-GFP Perlen
- Kurz vortexen Kügelchenaufschlämmung, um die Perlen zu suspendieren. Mit einer 200 ul Pipettenspitze mit dem Ende abgeschnitten, Transfer 25 ul Perlen pro Probe in ein frisches Röhrchen.
Hinweis: Der Anwender sollte die Perlen für ein einzelnes Experiment SILAC als Mastermix zur Probe zu Probe Variation zu minimieren vorzubereiten. - Für jeweils 25 ul Kügelchenaufschlämmung, mit 20 Bänden (1500 ul pro 75 ul Slurry) Verdünnungspuffer und Zentrifuge die Perlen auf 2.700 xg für 5 min. Weiter, waschen Sie die Perlen eine weitere 2x in 20 Volumina Verdünnungspuffer.
- 100 l Verdünnungspuffer pro 25 ul Kügelchenaufschlämmung. Mit einer 200 ul Spitze mit dem Ende abgeschnitten, Transfer 85 ul dieser resuspendiert Schlamm zu jedem der SILAC-markierten Proben aus Schritt 3.6.
- Die Proben mit Perlen auf einem Rotator Inkubieren bei 4 ° C für 2 Stunden.
5. Waschen, Elution und Vorbereitung der Proben für die MS-Analyse
- Centrifuge Proben bei 2.700 × g, 46, C für 5 min. 50 ul des Überstands als der Rest der Probe zurückgehalten werden, wobei der Rest des Überstandes verworfen.
- 1 ml Verdünnungspuffer zu jedem Röhrchen zugegeben, um die Perlen zu resuspendieren, und die Probe bei 2700 × g zentrifugiert, 4 ° C für 5 min. Der Überstand sollte verworfen werden. Dies sollte zweimal durchgeführt werden.
- Eluieren Protein von den Kügelchen durch Zugabe von 50 ul 2x SDS-Ladepuffer, und Erhitzen bei 95 ° C für 10 min. Pellet die Kügelchen durch Zentrifugation bei 2.700 × g für 2 min bei 4 ° C
- Behalten den Überstand in Rohren vorgeschmiert, wo es dann bei -80 ° C bis zur Verwendung gelagert werden. Zur Vorlage bei einer Massenspektrometrie-Anlage, mischen 01.01.01 markierten Proben (z. B. 10 ul von jedem) und senden Sie die Mischprobe.
Hinweis: An dieser Stelle Proben können durch Western-Blot-, Silber-Färbung oder andere Mittel, um für die Interaktion mit bekannten / unbekannten Bindungspartner testen getestet werden. Ein Beispiel ist in Abb.Abbildung 2 zeigt die spezifische Bindung eines bekannten Interaktionspartner - eIF4G durch Western-Blot, und das Auftreten von Silberfärbung im Pulldown-Proben aus einem Protein von Interesse vorhanden Bänder, nicht aber eine Kontrollprobe. - Probe Abschicken LC-MS/MS Analyse.
Hinweis: einmal zufrieden, dass ein markiertes Protein von Interesse erfolgreich verbindliche Interaktionspartner, gleiche Volumina jeder markierten Probe (leicht, mittel und schwer) werden kombiniert und für LC-MS/MS Analyse unterzogen. Es ist üblich, insgesamt 30 ul einer IP-Probe zur Analyse einreichen. Dies würde voraussetzen, indem 10 ul der Licht-markierten Probe mit 10 ul Medium-markierte und 10 ul schwer markierten Proben, um eine 30 &mgr; l Gesamt geben.
6 Datenanalyse I:. Verständnis der Ergebnisse und Entfernen von Low-Vertrauen Identifikationen
Hinweis: Eine Liste der Spaltenüberschriften von der Proteome Discoverer Software zurückgegeben wird in Tabelle 1 angegeben Dif.schiedenen Software (zB MSQuant, MaxQuant) wird verschiedenen Rubriken jedoch zurückkehren, nur eine Teilmenge davon sind für die Analyse benötigt, die bei den verschiedenen Software-Pakete sind. Daten sollten immer auch eine Zugangsnummer für jedes Protein identifiziert, Quote Vergleichen jeder Probe Verhältnis (Licht vs Medium Licht vs schwere, mittelschwere vs schweren etc.), die Anzahl der eindeutigen Peptiden identifiziert, und eine Form von falsch-positiven Rate oder Vertrauensanzeige.
- Bevor die Daten zuerst die Roh-Daten in eine neue Tabelle kopieren. Aus dieser Tabelle entfernen Sie alle Spalten außer für solche, die Zugangsnummer, die Anzahl der einzigartigen Peptiden, Verhältnisse Vergleich Proben Verhältnis Variabilität und das Protein Beschreibung. Wenn Replik Experimente wurden durchgeführt, sollten diese in einer einzigen Excel-Datei kombiniert werden, wobei jedes Experiment auf einer separaten Registerkarte erscheint.
Hinweis: geringes Vertrauen Daten Proteine durch nur eine einzige einzigartige Peptid identifiziert, und die whier Quantifizierung war nicht möglich. - Verwenden Zeichnet '"sort"-Funktion, um die Daten durch die Anzahl der Peptide bestellen und entfernen Sie die Einträge für Proteine fehlen mehr als ein Peptid. Dann sortieren Verhältnis und Proteine, die SILAC Verhältnisse (nicht quantifizierte Proteine) fehlt entfernen.
- Konvertieren SILAC Verhältnisse, um ein Protokoll 2-Werte nach der Formel: '= log (SILAC Verhältnis, 2)', wobei 'SILAC Verhältnis "wird für die Zellkennung ersetzt.
Hinweis: Als SILAC Verhältnis Ergebnissen in den Daten für Proteine, die eine Abnahme in Hülle und Fülle auf Werte zwischen 0 und 1 beschränkt ist, ist es üblich, eine SILAC Verhältnis zu einem log 2 SILAC Verhältnis zu konvertieren, da dies bedeutet, beide Proteine erhöht oder verringert eine Probe auf einer logarithmischen Skala, wobei 2 oder -2 eine 4-fache Zunahme oder Abnahme der Häufigkeit und 3 oder -3 eine 9-fache Zunahme oder Abnahme der Häufigkeit bzw. dargestellt. Nach dieser Transformation, sollten die Daten einen Ga passenussian Verteilung um ein log 2 SILAC Verhältnis von 0 zentriert. Ein Beispiel hierfür ist in Fig. 3 angegeben. - Erstellen Sie neue Spalten in der Excel-Datei und berechnen log 2 SILAC Verhältnisse für alle Proben / Mock Spalten. Für die Konvertierung von Layout / Probe Verhältnis zu einer Probe / Mock-Verhältnis, verwenden Sie die Formel "= 1/ratio"
7 Datenanalyse II:. Auswahl Hohe Vertrauen Wechselwirkungen für weiteres Studium
- Offene Graphpad Prism, wählen Sie Neu> Datentabelle und Grafik ... Select 'Spalte' aus der Liste auf der linken Seite des Fensters, und wählen Sie die Enter / Import von Daten> Geben Sie Replik Werte in Spalten Optionen gestapelt. Drücken Sie auf "Erstellen".
- Markieren und kopieren Sie eine bestimmte log 2 Sample / Mock SILAC Verhältnis Spalte aus der Excel-Tabelle in die neue Prism-Datei.
- Klicken Sie auf den Dropdown-Menü 'Einfügen' und wählen Sie "Neue Analyse". Unter Spalte Analyse auswählen "Häufigkeitsverteilung. Halten Sie die Standardoptionen klicken Sie auf 'OK'.
Anmerkung: Dieser Schritt erzeugt ein Histogramm, das die Anzahl von Proteinen in einem gegebenen Verhältnis gekennzeichnet. Dies sollte eine Gauß-Verteilung bilden. - Im Ordner "Ergebnisse" ein neuer Abschnitt "Histogramm" wird erzeugt wurden. Wählen Sie die Häufigkeitsverteilung Abschnitt.
- Klicken Sie auf den Dropdown-Menü 'Einfügen' und wählen Sie Neue Analyse> XY> Nichtlineare Regression (Kurvenanpassung). Klicken Sie auf "Gauß-Verteilung" und klicken Sie auf OK.
Anmerkung: Dieser Schritt entspricht einer Kurve an die Frequenzverteilungsdaten in Schritt 7.3 erzeugt, wodurch Werte, die anschließend verwendet werden können, einen Schwellenwert für Signifikanz zu berechnen. - Im Ergebnisfenster, das erscheint, der Mittelwert und Standardabweichung angegeben. Generieren Sie einen Schwellenwert, indem 1,96 Standardabweichungen zum Mittelwert.
Hinweis: Die Werte repräsentieren den Mittelwert und die Standardabweichungen der Gaußschen distribution, nicht die Gesamtdatenmenge. 1,96 Standardabweichungen würde eine Schwelle, bei der 95%-Vertrauensbereich (p ≤ 0,05) zu platzieren. 2,58 SD würde 99%, und 3,3 SD 99,9% zu ergeben. Der Schwellenwert für jede Replikat-Versuch bestimmt, wie es variieren kann.
8 Datenanalyse III:. Zusammenführen von Datasets Replica
Hinweis: Um sicher bei der Identifizierung der Interaktionspartner ein typisches SILAC Pulldown Experiment sollte idealerweise drei Mal durchgeführt werden, mit der mittelschweren und schweren markierten Proben eingeschaltet für eine der Wiederholungen für jede Wirkung der Medien auf die Ergebnisse zu kontrollieren. Ein Protein, das zeigt, wie die Interaktion in mindestens zwei der drei Experimente mit "switched' medialen Probe idealer entsprechenden von diesen ist ein hohes Vertrauen Interaktion.
- Zurück zu der Excel-Datei, erstellen Sie eine neue Registerkarte "kombiniert". Spalte Label A 'Beitritt' und kopieren Sie alle Zugangsnummern von jedem of die einzelnen Experimente in diesem einzigen Spalte.
- Wählen Sie die "Daten" Registerkarte und dann die Option "Duplikate entfernen".
- Erstellen Sie Spalten für die SILAC Verhältnissen, und Verhältnis Variabilität von jedem Experiment als auch für das Protein Name / Beschreibung Spalte.
- In der Beschreibung Registerkarte, verwenden Sie die vlookup Formel, um die Beschreibung der einzelnen Zugangsnummer zu sammeln. Ziehen Sie die Formel nach unten in der Spalte auf der Proteinbeschreibung zu füllen. Die vlookup Formel lautet: "= SVERWEIS (AccessionNo, WheretoLook, ColumnsAccross, False)", wobei: AccessionNo ist $ aX, wobei X die Zeilennummer WheretoLook ist das 'Experiment Register Name' $ a $ 2:.! $ Y $ Z, wo Y und Z die untere rechte Teil der Daten auf die Tabelle verwiesen wird. ColumnsAccross ist die Anzahl der Spalten gegenüber dem Beitritt number in Spalte A ein Sollwert liegt.
Hinweis: Zum Beispiel, wenn die Zugangsnummer in Spalte A, und die Daten von Interesse in Spalte E. Der Wert liegt hier würde 5 sein (Spalte A zählt als 1). Zugangsnummern in Schritt b vereinigt, ist es sehr wahrscheinlich, dass vlookup werden alle erforderlichen Namen von einem Experiment nicht erhalten. Wo gibt es N / A, ändern Sie die Formel, die Beschreibung von jedem Experiment wiederum zu erhalten, bis alle Beschreibungen erworben werden. - Zur Hervorhebung der interagierenden Proteinen in der kombinierten Datensatzes, wählen Sie die einzelnen Spaltenverhältnis individuell und klicken Sie auf der Registerkarte Start, gefolgt von Bedingte Formatierung> Highlight Zellen Regeln> Weitere Regeln.
- Wählen Sie Stil 'Classic'. Nur Zellen formatieren, die "Zellwert" enthalten, "Größer als oder gleich '. In dem Feld in der 1,96 Standardabweichungswert, und klicken Sie auf 'OK'.
- Bewerten Sie die Verhältnis Variabilität für jede positive Interaktion. Wenn Subtraktion der var%AFTUNG fallen würde einen "Hit" unter den Schwellenwert, sollte dies mit Vorsicht behandelt werden.
Hinweis: Diese stellt, wie konsequent die individuellen Verhältnisse für jedes Peptid, die zusammen ein Verhältnis SILAC Proteine "sind, und wird in Prozent angegeben. Wenn das Verhältnis Variabilität berücksichtigt wird und ein Protein ergibt eine SILAC Verhältnis über dem Schwellenwert liegt, dieses Protein einen Treffer. Wenn das Verhältnis Variabilität gibt einen Bereich, der unter den Schwellenwert fällt, kann die "Treffer" eine Verunreinigung darstellen. - Wiederholen Sie diesen Vorgang für jede der Spalten und vergleichen Sie die Ergebnisse in Experimenten. In zwei oder mehreren Experimenten identifiziert Hervorgehoben Proteine stellen ein hohes Vertrauen Interaktion.
Hinweis: Je nach der zur Protein-Zugangsnummern zuordnen, in verschiedenen Experimenten Protein kann unter einem anderen oder sogar mehrere Zugangsnummern identifizierten Datenbank. Es ist wichtig, dies zu überprüfen, um sicherzustellen, dass die Proteine nicht versehentlich weggelassen.
Ergebnisse
In einem typischen SILAC Pulldown Experiment wurde die überwiegende Mehrheit der identifizierten Proteine (> 90%) stellen Verunreinigungen sowie Proteine bindenden unspezifisch an die Affinitätsmatrix und dieser ist in Fig. 2B dargestellt ist, auch dann, wenn Waschprotokolle Entfernen einer Mehrheit von cytoplasmatischen Verunreinigungen wie GAPDH (2A). Allerdings ist die Clusterbildung von nicht-spezifisch bindenden Proteinen in einer Normalverteilung können Proteine, die spezifisch an ein Protein von Interesse binden, um so diese zu unterscheiden höhere Proben / Mock-Verhältnisse als der Hintergrund. Während Verunreinigungen theoretisch um einen log 2 SILAC Verhältnis von 0 Cluster, ist dies nicht notwendigerweise der Fall ist, wird ein Beispiel dafür in Fig. 3 angegeben. Mögliche Gründe hierfür sind unvollkommen SILAC Markierung von Zellen geladen ungleichen Mengen oder Konzentrationen des Lysats auf die Anti-GFP-Perlen, den zufälligen Verlust von Perlen während der Reinigung oder ungleichen Mixten von Samples am Ende des Reinigungsverfahrens 3. Jedoch werden unter der Annahme, Daten basierend auf einem Schwellen-Standardabweichung vom Mittelwert der normal verteilten Verunreinigungen analysiert sollten geringfügige Verschiebungen in der Zentrierung der Daten keine Auswirkungen auf die Qualität der Ergebnisse.
Beim Vergleich Unterschiede in Protein-Wechselwirkungen zwischen zwei verwandten Proteinen kann eine ähnliche Situation auftreten, wo eine interessierende Protein zu höheren Ebenen in Zellen als ein zweiter (entweder aufgrund von Variationen in der Transfektionseffizienz oder eine intrinsische Eigenschaft des Proteins oder mRNA) hergestellt . Einige Unterschiede in der Expression (z. B. 2A) kann durch die Analyse der SILAC-Verhältnis für diese beiden Proben korrigiert werden. In diesem Beispiel würden diese die GFP-und GFP-eIF4AI eIF4AII Proben sein. Durch die Analyse der 4AI/4AII SILAC Verhältnis wie in Kapitel 7 beschrieben, ist es möglich, Proteine, deren Bindungs schwankt erheblich von Isoformen zu identifizieren.
In Abbildung 4 ist eine Darstellung der eIF4A-bindende Proteine in einem der Replik Experimente durchgeführt, identifiziert, die die Abdeckung der Initiationsfaktor-Komplex dieses Protokoll erreicht gezeigt. Die höchsten Werte wurden typischerweise mit eIF4G, die direkt an eIF4A bindet beobachtet, wobei niedrigere Verhältnisse für eIF4E, die eIF4G an einer Stelle entfernt von der eIF4A-Bindungsstelle bindet. Niedrigere Verhältnisse wurden für die Mitglieder der eIF3 Komplex beobachtet. Dies jedoch deutlich, dass das Experiment zufriedenstellend sowohl direkte als auch indirekte Bindungspartner eIF4AI und II identifiziert. Wie nicht anders von ihren hohen Sequenzidentität 6, erwartet werden Protein-Protein-Wechselwirkungen erschien weitgehend zwischen den beiden Isoformen in diesem experimentellen System 7, 8 erhalten. Eine Auswahl einiger der interagierenden Proteine sind in Tabelle 2 angegeben, veranschaulicht das Datenformat.| Spaltenüberschrift | Beschreibung |
| Beitritt | Zeigt die Zugangsnummer für die Sequenz |
| Berichterstattung | Anteil der Proteinsequenz der identifizierten Peptide abgedeckt |
| ♯ PSM | Peptide spektrale Übereinstimmung |
| ♯ Peptide | Gesamtanzahl von einzigartigen Peptide für ein Protein identifiziert |
| ♯ AAs | Die Länge eines Proteins in Aminosäuren |
| MW (Da) | Das Molekulargewicht eines Proteins in Dalton. Ohne Änderungen |
| gef. PI | Der theoretische isoelektrische Punkt des Proteins |
| Partitur | Die Gesamtpunktzahl eines Proteins (die die Summe der Einzelverfahren stelltidual Peptid-Scores). Die genaue Punktzahl für Bedeutung erforderlich sind, zwischen den Experimenten variieren. Ein MS-Anlage wird in der Regel gelten 5% falsch-Entdeckungsrate Cutoff. |
| Folge | Die Sequenz der Aminosäuren, die das Protein bilden, |
| Verhältnis | Die relative Intensität der Peptide in einer benannten markierten Probe, verglichen mit einem zweiten markierten Probe |
| Quote Anzahl | Die Anzahl der Peptidverhältnisse, die verwendet wurden, um die ein gegebenes Protein-Verhältnis zu berechnen |
| Verhältnis Variabilität (%) | Die Variabilität der einzelnen Peptidverhältnisse verwendet werden, um einen gegebenen Proteinverhältnis zu berechnen |
| Beschreibung | Der Name des Proteins |
Tabelle 1. Standard-Spaltenüberschriften aus einer Proteome Discoverer Bericht.Während nützliche Informationen aus all diesen Spalten gewonnen werden, sind diejenigen, die kritisch für diese Analyse fett dargestellt.
| Beitritt | Peptide | 4AI/Mock | 4AII/Mock | Name | SILAC Analyse |
| A8K7F6 | 21 | 100 | 1 | eIF4AI | "Köder"-Protein |
| Q14240 | 22 | 0,01 | 90,855 | eIF4AII | |
| G5E9S1 | 25 | 47,575 | 30.53 | eIF4GI | Interagierenden Proteinen |
| Q59GJ0 | 5 | 11,778 | 10,619 | eIF4GII60; | |
| P06730 | 3 | 7,22 | 7,57 | eIF4E | |
| Q5T6W5 | 4 | 0,685 | 0.646 | hnRNPK | Nicht spezifisch bindenden Verunreinigungen |
| P62805 | 1 | 0.531 | 0.498 | Histon H4 | |
| H6VRG2 | 18 | - | - | Keratin-1 | Umweltkontaminanten |
| P35527 | 11 | 0,01 | 0,01 | Keratin-Zytoskelett 1 9 |
Tabelle 2. Typische Daten aus einem Experiment SILAC Immunpräzipitation. Geben Beispieldaten für ein Protein von Interesse / Köder (hohe Peptide, hohes Verhältnis), Proteine mit einem ein Protein von Interesse (High / Low-Peptide, High-Verhältnis), nicht-spezifischen Bindung Proteine (high / low PeptIden, fällt Verhältnis unter Abschaltung - in diesem Experiment 0,96), und Umweltschadstoffen (oft hohen Peptide, negative Verhältnis / unterhalb der Schwelle).
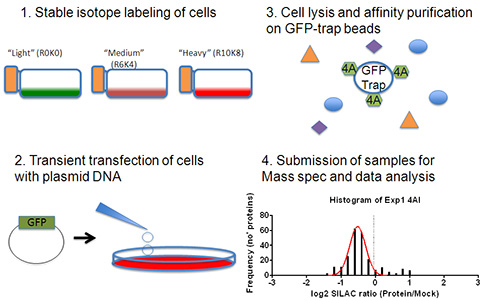
Fig. 1 ist. Experimentelle Plan. Erstens Zellen werden in Medien für 2 Wochen gezüchtet Arginin und Lysin fehlt und substituiert mit stabilen Isotopen markierten Arginin und Lysin (1). (2) Die Zellen werden in 10 cm 2 Schalen ausgesät und transient mit Plasmiden GFP codiert (Mock) oder GFP-Fusionsproteinen (Proben) transfiziert. (3) Die Zellen werden lysiert und GFP oder GFP-Fusionsproteine aus Zelllysaten immunpräzipitiert. (4) Die Proben werden in einem Verhältnis von 1:1 kombiniert und LC-MS/MS-Analyse vorgelegt. Die Daten werden dann analysiert, um ein geringes Vertrauen Protein ident entfernenkationen und auf ein Niveau von Proteinanreicherung entsprechend echten interagierende Proteine zu wählen.

Abbildung 2. Bestätigen geeignet Immunpräzipitation Bedingungen. A) Western-Blot-Analyse von Zell-Lysaten, wie auch die nicht-gebundenen und gebundenen Fraktionen der Immunpräzipitation bestätigt Expression und Immunpräzipitation des Proteins von Interesse. Ein Western-Blot gegen GAPDH bestätigt Verbrauch nicht interagierenden Proteinen und einem weiteren Western-Blot eine bekannte Interaktionspartner von eIF4A bestätigt die erfolgreiche Immunpräzipitation von Proteinen, die an das Protein von Interesse. B) wenn Interaktionspartner eines Proteins von Interesse nicht bekannt sind, eine Silber-gefärbten Gel kann Immunpräzipitation von interagierenden Proteinen bestätigen. Auf dieser Silber-Flecken ed Gel Bänder für GFP und GFP-eIF4AI/II sind klar, und eine Bande, die in der richtigen Größe für eIF4G ist nur in den GFP-4AI/II-bound Gassen und nicht in der GFP-Kontrollspur.
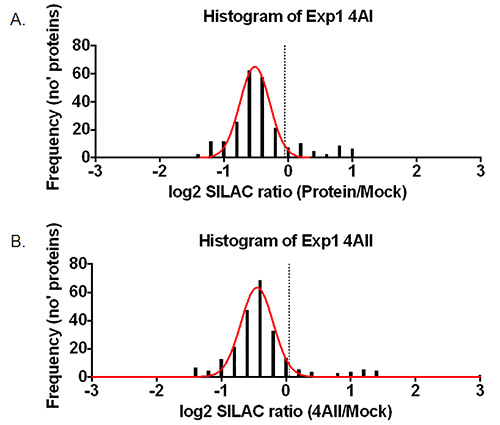
3. Repräsentative Ergebnisse. Histogramm, das die Verteilung von Protein-Verhältnissen von einer Wiederholung von (A) GFP-eIF4AI oder (B) GFP-eIF4AII Pulldown. Die Standardabweichung 1,96 Cutoff ist mit einer gestrichelten Linie markiert. Interagierende Proteine, die außerhalb der normalverteilten Verunreinigungen sind aus ~ 0,25 und 1 in (A) und ~ 0,3-1,5 in (B).
les/ftp_upload/51656/51656fig4.jpg "/>
Abbildung 4. Identifizierung von eIF komplexe Elemente aus einer einzigen Kopie eines SILAC IP-Experiment. Proteine in grün waren das Protein von Interesse für die Pulldown-interagierende Proteine verwendet werden, von rot nach weiß schattiert, um nach 2 SILAC-Verhältnis in der SILAC IP mit roten einloggen wobei das häufigste Protein in der Analyse, wobei die weißen und 1,96 SD Cutoff. Proteine grau schattiert wurden in dieser Analyse nicht identifiziert.
Diskussion
Die hier beschriebene SILAC Pulldown-Strategie stellt eine sehr empfindliche und leistungsfähige Methode zur Entdeckung von neuartigen Protein-Protein-Interaktionen, und darüber hinaus ermöglicht die schnelle und einfache Diskriminierung von veränderten Bindungsmuster zwischen eng verwandten Proben von Interesse. In diesem Beispiel wurde die Technik verwendet, um das Protein zu untersuchen: Protein-Wechselwirkungen der Proteine eIF4AI und eIF4AII 6. Nach Kenntnis des Autors, ist dies die erste Studie, in der Literatur die Nutzung der Nutzen der SILAC Proteomik, um die zelluläre Interaktom dieser beiden Isoformen von eIF4A zu untersuchen.
Der Ansatz, wie oben beschrieben, verwendet eine GFP-Tag-und Anti-GFP-Perlen 9, 10 und können daher Änderungen erforderlich, diesen Ansatz zu ermöglichen, für ein bestimmtes Protein von Interesse, zum Beispiel verwendet werden, ob das Tag am N-oder platziert werden C-Terminus eines Proteins. Wenn möglich, Western Blots oder funktionelle Assays solltendurchgeführt werden, um die Bindung eines bekannten Protein-Interaktionspartner. Sollte ein Protein nicht tolerieren Fusion mit einem GFP-Tag, Tagging oder anderen Pull-Down-Strategien mit den beiden anderen Tags zu SILAC pulldowns angewandt worden (FLAG 11, Biotin 12, STREP (eigene Daten, unveröffentlicht)) oder durch Verwendung primärer Antikörper gegen ein Protein von Interesse, wo siRNA Knockdown des Zielproteins bietet eine Kontrollprobe 13. Solche Experimente wurden an anderer Stelle in der Literatur beschrieben, aber kurz gesagt, Schritt 1, und die Schritte 5.4-8 würde wie oben angewendet werden, mit den Schritten 2 bis 5,3 als für die Expression / Pulldown-System der Wahl mit gleichen Protein Eingängen wie in geeignete modifizierte Schritte 2.4-2.5. Da die Quantifizierung Stufen ermöglichen nicht-spezifische Bindungsproteine auf der Analyseebene entfernt werden, empfiehlt es sich, vor der Inkubation mit Steuer Perlen oder hohe Salz wäscht, um mit geringer Affinität zu bewahren weglassen Protein-Protein-Interaktionen mit einem Protein von Interesse . Ein Nuklease may eingeschlossen oder von diesem Protokoll nach den Besonderheiten einer speziellen Experiment weggelassen. Zum Beispiel: wie die in diesem Verfahren verwendeten Proteine sind RNA-Helikasen, RNase-Cocktail wurde in das Protokoll aufgenommen, um über die indirekten Wechselwirkungen RNA (Schritt 3.2) vermittelt zu entfernen. In einigen Fällen konnte jedoch dort von parallel Versuche mit und ohne Nuklease Nukleinsäure-abhängigen Wechselwirkungen zu identifizieren profitieren werden.
In diesem Protokoll wird das Schalten von "mittel" und "schweren" Proben in wiederholten Experimenten empfohlen, um eine Schwankung von Differenzen in den SILAC Medien oder Zellwachstums eingeführt steuern. Eine alternative Steuerung beinhaltet die sequentielle Schalt aller drei ("leicht", "mittel" und "schwer") Medien in Replik Experimenten. Während dieser Ansatz möglicherweise strenger, erhöht es die Komplexität der Analyse, wie in mindestens einem Replikat, ein Protein von Interesse wkrank im "Licht" markierten Zellen hergestellt werden und so ist es notwendig, zwischen Proteinen konsequent in "Licht"-Proben (Umweltschadstoffe wie Keratine), und solche, die nur in der "light"-Proben angereichert sind, wenn an ein Protein gebunden identifiziert zu unterscheiden von Interesse.
Während die Verwendung von Quantifizierungsdaten erlaubt die Unterscheidung von spezifischen unspezifischer Wechselwirkungen durch Verwendung eines Schwellen zwangsläufig einige echte Wechselwirkungen können verworfen werden. Der obige Ansatz ist eine einfache und schnelle Ansatz zur Identifizierung von Protein-Protein-Wechselwirkungen, die leicht von den Forschern ohne vorherige Erfahrung mit der Massenspektrometrie, oder die Analyse von großen Protein versucht werden kann: Protein-Interaktionsdatensätzen. Für die meisten Anwendungen ist dies mehr als ausreichend für die Identifizierung neuer Proteine von Interesse. Weitere Modifikationen dieser Ansatz zur Verringerung dieses Datenverlust an anderer Stelle in der Literatur beschrieben und umfassen die Verwendung einer protein Frequenz Bibliothek, in der bekannten Schadstoff Proteine, die für eine bestimmte Gruppe von experimentellen Parameter (Zelllinie, Perlen Matrix, Pufferbedingungen) kann 10, 14 ausgeschlossen werden. In Abhängigkeit von den speziellen experimentellen Parameter kann es erforderlich sein, eine Anzahl von Kontrollexperimenten durchgeführt, um einen Wulst Proteom zu erzeugen, und dies kann daher sowohl die Kosten und die Komplexität des Experiments zu erhöhen. Weitere Informationen zu dieser Technik ist, von der Website www.peptracker.co.uk 14 zur Verfügung.
Es sollte auch angemerkt, dass die oben beschriebenen Protokoll beinhaltet das Mischen unterschiedlich markierten Proben am Ende der Immunpräzipitation Verfahren (bezeichnet eine Misch nach Reinigung - MAP SILAC Versuch) werden. Dies wird als Protein geführt: Protein-Wechselwirkungen bei einer gegebenen Gleichgewichts 15 auftreten. Es sei darauf hingewiesen, dass auch andere Gruppen haben diese in diesem Protokoll mit Inkubation der Proben beschrieben MAP SILAC Ansatz kombiniert werdenvor Pulldown (Reinigung nach dem Mischen - PAM SILAC) für verschiedene Zeitdauern (20 min bis 2 h wurden in der Literatur verwendet wurde) 15, 16. Je nachdem, wie schnell ein Protein-Verhältnis fällt in Richtung 1:1 ist, ist es möglich, qualitativ untersuchen Bindungsaffinitäten und Proteine als stabil oder dynamisch interagierende Proteine 15 definieren.
Zusammenfassend SILAC pulldowns stellen ein sehr leistungsfähiges Mittel zur Identifizierung von Proteinen, die Interaktion mit einem bestimmten Protein von Interesse, in einem physiologisch relevanten Einstellung. Die Technik kann sehr leicht zu einer Reihe von verschiedenen Reinigungsstrategien angepasst werden, so dass ihre Anwendung auf einem bestimmten Protein von Interesse. Quantifizierung der Ergebnisse erheblich vereinfacht Identifizierung von echten Interaktionen und erlaubt Entspannung strengen Pufferbedingungen verwendet werden, um nicht-spezifische Bindemittel zu entfernen, und bewahrt so geringe Affinität Wechselwirkungen. Bis zu drei Proben in der oben verglichen werdenStrategie, die Technik hat klare Stärken im Vergleich Unterschiede in der Proteinbindung zwischen verschiedenen Protein-Isoformen, mutierten Proteine, oder die Wirkung von pharmakologischen Inhibitoren. Als ganze Gelscheiben statt einzelner Bands, die durch Coomassie-Färbung analysiert, sind die Zahlen von Proteinen bei hohen Vertrauen identifiziert in der Regel höher als in einer Standard-GST / TAP-Pulldown identifiziert und Experimentator Bias bei der Auswahl der Proteine von Interesse wird entfernt. Die Technik vergleicht daher sehr gut mit anderen häufig verwendeten Techniken in der Identifizierung neuer Protein-Wechselwirkungen (Hefe-2-Hybrid, GST / Sein oder Pulldowns TAP) verwendet.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen konkurrieren.
Danksagungen
Diese Arbeit wurde durch Zuschüsse aus dem Wellcome Trust und BBSRC IG unterstützt. IG Wellcome ist ein Senior Fellow.
Materialien
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium (DMEM) | Dundee Cell Products | LM010 | DMEM lacking Arginine and Lysine. (and containing L-glutamine) |
| Dialysed FBS (10 kDa cutoff) 500 ml | Dundee Cell Products | DS1003 | |
| Arginine (R0) 25 g | Sigma-Aldrich | A8094 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R6) 0.5 g | Cambridge Isotope Labs | CLM-2265 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R10) 0.5 g | Cambridge Isotope Labs | CNLM-539 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Lysine (K0) 25 g | Sigma-Aldrich | L8662 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K4) 0.5 g | Cambridge Isotope Labs | DLM-2640 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K8) 0.5 g | Cambridge Isotope Labs | CNLM-291 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Penicillin/streptomycin, Liquid. 100 ml | Gibco | 15140-122 | |
| Lipofectamine 2000 transfection reagent | Invitrogen | 11668-027 | |
| GFP-trap Agarose (500 μl resin) | Chromotek | gta-20 | |
| 5x SDS Sample loading buffer | Fisher Scientific | PN39000 | The use of purchased rather than homemade sample buffer is recommended to minimize keratin contamination. |
| Protease inhibitor cocktail set III | Calbiochem | 539134 | |
| 1.7 ml prelubricated tubes | Costar | 3207 | |
| BCA protein assay kit (1 L) | Pierce | 23225 | |
| Tris (Trizma) | Sigma-Aldrich | T1503 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E6758 | |
| Sodium chloride | Sigma-Aldrich | S3014 | |
| Rnase cocktail | Ambion | AM2286 | |
| NP-40 alternative | Millipore | 492016 |
Referenzen
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem Soc Trans. 38, 875-878 (2010).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), (2012).
- Trinkle-Mulcahy, L. Resolving protein interactions and complexes by affinity purification followed by label-based quantitative mass spectrometry. Proteomics. 12, 1623-1638 (2012).
- Emmott, E., et al. The cellular interactome of the coronavirus infectious bronchitis virus nucleocapsid protein and functional implications for virus biology. J Virol. 87, 9486-9500 (2013).
- Li, W., Belsham, G. J., Proud, C. G. Eukaryotic initiation factors 4A (eIF4A) and 4G (eIF4G) mutually interact in a 1:1 ratio in vivo. J Biol Chem. 276, 29111-29115 (2001).
- Nielsen, P. J., Trachsel, H. The mouse protein synthesis initiation factor 4A gene family includes two related functional genes which are differentially expressed. EMBO J. 7, 2097-2105 (1988).
- Galicia-Vazquez, G., Cencic, R., Robert, F., Agenor, A. Q., Pelletier, J. A cellular response linking eIF4AI activity to eIF4AII transcription. RNA. 18, 1373-1384 (2012).
- Zakowicz, H., et al. Mutational analysis of the DEAD-box RNA helicase eIF4AII characterizes its interaction with transformation suppressor Pdcd4 and eIF4GI. RNA. 11, 261-274 (2005).
- Rothbauer, U., et al. A versatile nanotrap for biochemical and functional studies with fluorescent fusion proteins. Mol Cell Proteomics. 7, 282-289 (2008).
- Trinkle-Mulcahy, L., et al. Identifying specific protein interaction partners using quantitative mass spectrometry and bead proteomes. J Cell Biol. 183, 223-239 (2008).
- Dobreva, I., Fielding, A., Foster, L. J., Dedhar, S. Mapping the integrin-linked kinase interactome using SILAC. J Proteome Res. 7, 1740-1749 (2008).
- Mittler, G., Butter, F., Mann, M. A SILAC-based DNA protein interaction screen that identifies candidate binding proteins to functional DNA elements. Genome research. 19, 284-293 (2009).
- Selbach, M., Mann, M. Protein interaction screening by quantitative immunoprecipitation combined with knockdown QUICK. Nat Methods. 3, 981-983 (2006).
- Boulon, S., et al. Establishment of a protein frequency library and its application in the reliable identification of specific protein interaction partners. Mol Cell Proteomics. 9, 861-879 (2010).
- Wang, X., Huang, L. Identifying dynamic interactors of protein complexes by quantitative mass spectrometry. Mol Cell Proteomics. 7, 46-57 (2008).
- Fang, L., et al. Characterization of the human COP9 signalosome complex using affinity purification and mass spectrometry. J Proteome Res. 7, 4914-4925 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten