Method Article
Falten und Charakterisierung eines Bio-responsive Robot von DNA-Origami
In diesem Artikel
Zusammenfassung
DNA origami is a powerful method for fabricating precise nanoscale objects by programming the self-assembly of DNA molecules. Here we describe a protocol for the folding of a bio-responsive robot from DNA origami, its purification and negative staining for transmission electron microscopic imaging (TEM).
Zusammenfassung
Die DNA-Nanoroboter ist eine hohle hexagonale Nanometereinrichtung, entworfen, um als Reaktion auf bestimmte Reize und Gegenwart Fracht innerhalb abgesondert zu öffnen. Beide Reize und Ladung kann nach spezifischen Bedürfnisse zugeschnitten werden. Hier beschreiben wir die DNA Nanoroboters Herstellungsprotokoll, wobei die Verwendung des DNA-Origamitechnik. Das Verfahren startet, indem man kurze einsträngige DNA von Klammern in einer Stammischung, der dann in Gegenwart eines Faltungspuffer zu einem langen, kreisförmigen, einsträngigen DNA-Gerüst aufgenommen. Ein Standard-Thermocycler so programmiert ist, allmählich gesenkt werden das Mischen der Reaktionstemperatur, um die Klammern zu Gerüstglühen, die die treibende Kraft hinter der Faltung des Nanoroboters ist erleichtern. Sobald die 60 h Faltungsreaktion vollständig ist, werden die überzähligen Klammern unter Verwendung einer Filterzentrifuge, gefolgt von Visualisierung über Agarose-Gelelektrophorese (AGE) verworfen. Schließlich wird erfolgreiche Herstellung des Nanoroboters durch Transmissionselektronenmikroskopie (TEM) überprüft wird,mit der Verwendung von Uranyl-Formiat als negative Färbung.
Einleitung
Die Einsatzmöglichkeiten für Nukleinsäuren Nanotechnologie sind erstaunlich. Die Lenkbarkeit des Watson-Crick-Basenpaarung sowie die einfache und relativ kostengünstige großtechnische Synthese von maßgeschneiderten Oligos 2 hat eine Explosion von Anwendungen 3 und Forschung auf dem Gebiet der DNA-Nanotechnologie erzeugt. Strukturelle DNA Nanotechnologie, bezogen auf das immobile Seeman Kreuzung 4,5 als grundlegender Baustein macht Verwendung von DNA als eine selbstorganisierende Elementareinheit für die Konstruktion von beliebigen Formen 6-8.
Die jüngste Entwicklung der rüstet DNA-Origami-9-Technik ermöglicht die Konstruktion von komplexen 2D- / 3D-Nanoarchitekturen 10-12 mit Sub-Nanometer Präzision und ist ein effizienter Weg für den Bau neuer funktionale Objekte mit zunehmender Komplexität und erstaunliche Vielfalt. Der Bauprozess wird auf eine lange Gerüsteinzelstrang-DNA basiert, in der Regel von einem viralen Genom abgeleitete, der durch die Hybridisierung von Hunderten von kurzen Einzelstrang-DNA-Oligos gefaltet werden kann bezeichnet Heftklammern. Die hohe Strukturauflösung durch diese Technik erhalten ist das direkte Ergebnis der natürlichen Dimensionen der DNA-Doppelhelix, während die Reproduzierbarkeit der Herstellung ist das Ergebnis der Anpassung der kurzen Einzelstrang-Stapelsequenzen, um die maximale Wasserstoffbrücken Komplementarität erreichbar zu erleichtern. Mit dem Einsatz von einem langsamen Temperaturglühen Rampe die entworfen niedrigsten Energie, in hohen Ausbeuten und Treue thermodynamisch bevorzugte Nanostruktur erreicht wird. Die einfache Umsetzung der Kreuzung Design-Regeln in einem Computercode aktiviert die Entwicklung von CAD-Tools, wie caDNAno 13, die extrem vereinfachen die Aufgabe der Gestaltung großer, komplexer Strukturen, die Hunderte von Übergängen verbunden.
Zuvor beschrieben wir die Konstruktion eines DNA-Nanoroboter mit Hilfe des caDNAno Werkzeug 14,15. Die Herstellung Hier zeigen wir, undVisualisierung, über Transmissionselektronenmikroskopie (TEM), der Nanoroboter, einem 3D hohlen sechseckigen Nanovorrichtung, mit den Abmessungen von 35 x 35 x 50 nm 3, entworfen, um eine große Konformationsänderung in Reaktion auf eine vorbestimmte Stimuli und Gegenwart spezifische Ladung, unterzogen werden wie wie Proteine oder Nukleinsäure-Oligos, abgesondert innen. Während 12 Verladestationen stehen innerhalb der hohlen Gehäuse, die tatsächliche Anzahl der gebundenen Ladung unterscheidet mit Ladung groß. Cargomoleküle reichen von kleinen DNA-Moleküle, Enzyme, Antikörper und 5-10 nm Goldnanopartikel. Cargocan entweder einheitlich oder heterogen, so dass jedes Nanoroboters enthält ein Gemisch aus verschiedenen Molekülen. Sensor ist über zwei Doppelschräg Sperriegel Design entweder aptasensor 16,17 oder DNA-Strang-Verdrängungs-18-Technologien erreicht werden, um Proteine, Nukleinsäuren oder andere Chemikalien zu erfassen, beruht. Jüngste Entwicklungen in der Aptamer Selektionsprotokolle 19-21 ermöglichen die Gestaltung von Nanoroboter reagiertzu einer ständig wachsenden Palette von Molekülen und Zelltypen.
Frühere Arbeiten zeigten eine Nanoroboter, der einen spezifischen Antikörper, der nach der Bindung an sein Antigen kann entweder eine hemmende oder ein produktiver Signal an der Innenseite von spezifischen Zelltypen in einer gemischten Zellpopulation 15 weiterzuleiten. Eine spannende Merkmal dieser Nanomaschinen ist ihre Fähigkeit, auch komplexere Aufgaben und Logic-Control mit der Einführung von verschiedenen Nanorobotik-Subtypen in einer einzigen Population durchzuführen. Kurzem zeigten wir bestimmte Subtypen von Nanoroboter Führen entweder positive oder negative Regulatoren, Steuern eines Effektorpopulation enthaltend einen aktiven Frachtmoleküls 22.
Die hier vorgestellte Protokoll beschreibt die Herstellung, Reinigung und Bildgebung eines Nanoroboter mit Aptamer-Sensor-Sequenzen, die selektiv an PDGF binden, um die Öffnung der Nanorobotik 15,22 erleichtern gated. Das beschriebene Herstellungsverfahren ist ähnlich dem nanorobot Herstellungsverfahren zunächst von Douglas et al. 15 mit Änderungen zur Verringerung der Gesamtprozessdauer ausgerichtet dargestellt, während die Erhöhung der Ausbeute und Reinigungsraten.
Protokoll
1. Herstellung von Staples Pool Mixture
- Sortieren lyophilisiert DNA Nanorobotik Heftklammern auf 96-Well-Platten, wie in Tabelle 1 aufgeführt (siehe Materialien) und zu normalisieren, um 10 nmol. Für eine detaillierte Beschreibung der Konstruktion und Architektur des DNA Nanoroboters siehe Ben-Ishay et al. 14 und Douglas et al. 15).
- Rekonstituieren Stapel gut mit DNase / RNase-freies Reinstwasser auf eine Konzentration von 100 uM. Für Heftklammern bis 10 nmol normalisiert, auflösen mit 100 ul von ultrareinem Wasser.
- Pool zusammen 20 ul jeder Klammer mit einer Mehrkanalpipette und ein steriles 55 ml-Lösung Becken.
Hinweis: Da der Nanorobotik Form von 254 Heftstränge (einschließlich Kern, Kanten, Griffe und Guides Sequenzen, Tabelle 1), die Konzentration von jedem der Heftklammern in der Heftklammern Pool umfasste ist 394 nM. Entfernen von Heftklammern aus der Heftklammer-Pool oder das Hinzufügen einige an verschiedenen volUMEs, kann die Ausbeute deutlich zu reduzieren, oder auf andere Weise in ungefalteten Aggregate führen.
2. Herstellung von Fabrication Reaktionsgemisch
- Für ein Standard-Reaktionsgemisch zu verwenden 40 ul M13mp18 viralen Genoms kreisförmigen Einzelstrang-DNA, entsprechend 4 pmol Gerüst DNA (100 nM Stammlösung, siehe Materialien). Endkonzentration von Gerüst bei der Herstellung Gemisch 20 nM, Endvolumen 200 ul.
- Stellen Sie die Höhe der Gerüst DNA nach deren spezifischen Bedürfnissen; Jedoch halten die Endkonzentration von Gerüst-DNA in der Reaktionsmischung auf einem konstanten 20 nM.
- Hinzufügen Klammern an ein Gerüst, Klammern Verhältnis von 1 zu 10 zu erreichen sind. Für ein 20 nM Gerüst-DNA-Konzentration ist jede Endkonzentration 254 Staples 200 nM. Für eine 200 ul Endreaktionsvolumen fügen 102 ul der Heftklammern Pool Mischung (Abschnitt 1.2).
- In bestimmten Gate-Sequenzen getrennt zu the Klappreaktionsgemisch zu diesem Zeitpunkt. Diese Oligos sind separat zu bestellen und erfordern HPLC-Reinigung. Stellen Sie sicher, dass Tor Oligos in einer 1.10 Gerüst Tor Sequenz Verhältnis vorliegen, dh 200 nM jedes Oligos für eine 20 nM Gerüst Konzentration. Für ein 200 & mgr; l Reaktionsvolumen Falten, je 0,4 & mgr; l für jede der vier Gatter Oligo bei 100 uM Stammkonzentration.
- Hinzufügen 10x TAE Lager Puffer zu einer endgültigen Konzentration von 1x TAE (40 mM Tris-Acetat, 1 mM EDTA) zu erreichen. Für eine 200 ul Klappreaktionsvolumen, fügen Sie 20 ul 10x TAE.
- 1 M MgCl 2 zu einer Endkonzentration von 10 mM. Für eine 200 ul Klappreaktionsvolumen, fügen Sie 2 ul 1 M MgCl 2.
- Hinzuzufügen 36 ul DNase / RNase frei Reinstwasser ein Erreichbarkeits ein Endvolumen von 200 ul.
- Vortex und aliquoten 100 ul Proben in PCR-Röhrchen.
Hinweis: Wenden Thermocycler-Spezifikation in Bezug auf die zu verwendenden maximalen Reaktionsvolumina.Verringerung des Volumens, um diese Grenzen erfüllen nicht die erzielten Ausbeuten reduzieren. Reaktionsvolumina über dem angegebenen werden die Erträge beeinträchtigen Maximum.
3. Temperaturglühen Ramp von Fabrication Reaction
- Programm Thermocycler wie folgt:
- Rampe 85 ° C bis 60 ° C mit einer Geschwindigkeit von 5 min / ºC.
- Rampe 60 ° C bis 4 ° C bei einer Geschwindigkeit von 75 min / ºC.
- Halten bei 4 ° C auf unbestimmte Zeit.
- Nach der Herstellung hat Proben bei -20 ° C beendet.
4. Entfernung von überschüssigem Staples
- 100 ul Klappreaktionsgemisch auf ein 0,5 ml Zentrifugalfilter mit einem MWCO von 100 kDa. Speichern Sie eine 10 ul Probe der Nanoroboter Vorreinigung für eine spätere Analyse.
- Zentrifuge für 10 min bei 9.600 x g.
- Fügen Sie 400 ul Faltungspuffer (1x TAE, 10 mM MgCl 2).
- Wiederholen Sie die Schritte 4.2 und 4.3 zwei weitere Male.
- Zentrifuge für 5 min bei 9.600 x g.
- Wiederherstellen des Konzentrats, indem Sie den Filter auf den Kopf in einem sauberen Mikrozentrifugenröhrchen und Spin für 1 min bei 9600 x g. Endvolumina je nach Ausgangsvolumen der Fertigung Reaktionsmischung, die in den Filter gesetzt wurde, variieren. Typischerweise ein 25-60 ul Endvolumen konzentrierte Nanoroboters Probe erhalten.
- Messung der DNA-Konzentration in den Proben mittels Spektrophotometer bei 260 nm. Verwenden Molekulargewicht von 5,3 g / pmol bei der Berechnung der molaren Konzentrationen von Nanorobotik Proben.
Anmerkung: Der molare Extinktionskoeffizient für dsDNA, 50 ug / OD 260, reicht für die meisten Anwendungen. 48 & mgr; g / OD 260 berücksichtigt die Poly Thymin ssDNA erstreckt sich an den Rändern Klammern.
5. Agarose-Gel-Elektrophorese Die Analyse der Folded Nanoroboter
- Herstellung von 0,5x TBE (45 mM Tris-Borat, 1 mM EDTA), 2% Agarosegel mit 10 mM MgCl 2 supplementiert (von Ernesto Castro et al angepasst. 21):
- Bereiten 0,5x TBE-Puffer durch Verdünnen von 6,25 ml 10x TBE stock Puffer in 118,75 ml ddH 2 O.
- Aufzulösen 2,5 g Agarose in 125 ml 0,5 x TBE-Puffer.
- Sieden Mikrowelle, bis die Agarose vollständig gelöst ist.
- Hinzufügen 1,25 ml 1 M MgCl 2 zu einer Endkonzentration von 10 mM.
- Hinzufügen 7 ul 10 mg / ml Ethidiumbromid.
- Warten Sie, bis Lösung etwas abkühlen lassen und füllen Sie den Gelträger, bevor die Agarose-Gel verfestigt. Installieren Sie die gewünschten Kamm sofort.
- Vorbereitung 1 L 0,5x TBE Laufpuffer durch Zugabe von 50 ml 10x TBE Stammpuffer und 10 ml 1 M MgCl 2 bis 940 ml ddH 2 O.
- Sobald Gel ist solide Add Laufpuffer in die Elektrophoresevorrichtung und legte Gerät in einem Eisbad.
- Last 1 & mgr; g Gesamt-DNA des Nanoroboter Vorreinigung (Schritt 3.2), und nach der Reinigung (Schritt 4.7), neben einem GerüstDNA-Probe und eine 1 kb DNA-Marker, auf das Gel. Ein Beispiel ist in Abbildung 2 wiedergegeben. Die tatsächliche Probenvolumina hängen von den nach der Reinigung erhalten Konzentrationen. 6 Endvolumen Verhältnis: jeweils Proben mit Beladungspuffer bei einer 1 geladen.
- Set Stromquelle auf 80 V und führen Gel für 3 Stunden. Führen Sie das Gel in einem Bad mit Eis und Wasser gefüllt. Nanoroboter wird sich entfalten und erscheinen als ein Abstrich, wenn das Agarose-Gel erwärmt sich während der Elektrophorese. Während der Elektrophorese fügen Sie zusätzliche Eis zum Gerät von Heizung zu halten.
- Anzeigen Gel auf einem UV-Tabelle (Figur 2).
6. Negative Stain von Nanoroboter mit Uranyl Formiat-
- Herstellung von 2% Uranyl Formiat-Stammlösung (Castro et al angepasst. 23):
- Wiegen Sie 100 mg Uranyl Formiat-Pulver in einen 15-ml-Tube.
- Boil DNase / RNase frei von ultrareinem Wasser für 3 min zu de-Sauerstoffverbindung.
- 5 ml desoxygeniertem heißem Wasser (~ 60 ° C)in die 15 ml Röhrchen mit dem Uranyl Formiat-Pulver (aus dem vorherigen Schritt). Dicht schließende Deckel, in Alufolie wickeln und Wirbel konsequent für 10 min (fasten Rohr zu einem Vortex). Lösung sollte trüb mit gelber Farbe angezeigt.
- Filter-Lösung durch einen 0,2 um Spritzenfilter. Lösung sollte klar sein.
- 200 ul Aliquot der Lösung in Mikrozentrifugenröhrchen.
- Zentrifuge bei Höchstgeschwindigkeit für 5 min in einer Tischzentrifuge zur Poolproben an der Unterseite des Rohres.
- Laden der Probe auf Raster und negative Färbung:
- Tauen Sie eine 200-ul-Aliquot von 2% Uranylacetat-Formiatlösung (in Abschnitt 6.1) hergestellt. In 10 ul 0,5 M NaOH-Lösung und Wirbel konsequent für 3 min. Zentrifuge bei Höchstgeschwindigkeit für 5 Minuten unter Verwendung einer Tischzentrifuge.
- Inzwischen Glimmentladung Gitter mit Raumluft bei 0,2 mbar und 25 mA für 30 Sekunden.
- In 15 ul 2 nM Nanorobotik Probe nach der Reinigung (Abschnitauf 4.7) auf Oberseite des Gitters (mit einer Pinzette gehalten) und lassen Sie absorbieren für 1 min. Verwenden Filterpapier, um die überschüssige Flüssigkeit, indem das Gitter über dem Filterpapier abtropfen. Die Gitteroberfläche nicht berühren.
- Zugabe von 10 ul 2% Uranylacetat-Formiat (aus Schritt 6.2.1) auf der Oberseite des Gitters (mit einer Pinzette gehalten werden).
- Benutzen schnell Filterpapier, um die überschüssige Flüssigkeit, indem das Gitter auf der Oberseite des Filterpapier abtropfen lassen. Die Gitteroberfläche nicht berühren.
- Wiederholen Sie die Schritte 6.2.4 und 6.2.5.
- Zugabe von 10 ul 2% Uranylacetat-Formiat (aus Schritt 6.2.1) auf der Oberseite des Gitters (mit einer Pinzette gehalten werden). Lassen Färbelösung absorbieren 30 Sek.
- Verwenden Filterpapier, um die überschüssige Flüssigkeit, indem das Gitter über dem Filterpapier abtropfen. Die Gitteroberfläche nicht berühren.
- Ließ das Gitter vollständig trocknen für mindestens 30 min, nach oben auf ein Filterpapier, vor dem Einspritzen in das TEM.
Ergebnisse
Repräsentative Ergebnisse sind in 2A gezeigt. Alle Spuren enthalten 1 & mgr; g Gesamt-DNA über Spektrophotometer (OD 260) gemessen. Im Vergleich mit dem kreisförmigen Einzelstrang-DNA-Gerüst (Spur 2), sind Nanoroboter im Gel aufgrund ihrer höheren Molekulargewicht behindert, ist das Ergebnis von Klammern Hybridisierung an das Gerüst DNA (Spur 3 roter Pfeil). Das Gewicht Band mit niedrigem Molekular in Spur 3 stellt überschüssige Heftklammern, die nicht auf das Schafott DNA (grüner Pfeil) binden wollte. Nach Reinigung über Zentrifugalfiltration meisten der überschüssigen Klammern werden entfernt (Spuren 4-6) und die Nanoroboter sind bereit, mit Frachtmoleküle an die Griffe komplementären Strang 14,15,22 konjugiert geladen werden. Spuren 4-6 enthalten Gewichtsbänder mit hohem Molekular darstellt Nanorobotik-Dimere (blauer Pfeil), eine Bevölkerung von Nanoroboter, die aneinander gebunden sind. Reduzierung der Konzentration des Gerüsts in dem Herstellungsverfahren (entweder 5 oder 10 nM) potentiell reduCE Das relative Menge dieser hochmolekulare Strukturen. Spuren 4-6 zeigen Nanoroboter Nachreinigung mit leichten Variationen auf das beschriebene Reinigungsprotokoll, nämlich das Ausgangsvolumen der hergestellten Nanoroboter zum Spin-Filter (Schritt 4.1 hinzugefügt Lane. 4: 50 & mgr; l; Spur 5: 100 & mgr; l; Spur 6: 100 ul), und die Anzahl der Male der Puffer zu dem Filter addiert (Schritt 4.4 Spur 4: 2-fache, Spur 5: 2. Zeiten; Spur 6: 5-mal).
Vergleich der Ausbeute und Reinigungsergebnisse zwischen den verschiedenen Varianten des Protokolls (2C) zeigen, dass die Verminderung des Ausgangsvolumens der hergestellten Roboter zu dem Zentrifugalfilter geladen (Schritt 4.1) hat kaum Auswirkungen auf den Ertrag und die Reinigungsraten. Überdies die Erhöhung der Anzahl der Pufferaustausch und zentrifugalen Drehungen (Schritte 4.4) hat geringen Einfluss auf die Reinheit auf den Nanoroboter, während erheblich reduziert die Ausbeute. Daher der beschriebenen Protokoll zu 100 ul entsprichtAnfangsvolumen und 2 Puffer Zusätze (Spur 5), wird als optimal angesehen.
Ausbeute Schätzungen auf Spektrophotometer (OD 260) Messungen von jeder Probe Nachreinigung auf und bezogen auf die Ausgangsmenge in jedem Zentrifugalfilter geladen berechnet. Aufreinigung basiert auf der Gewichtsprozentanteil der Nanoroboter aus der gesamten DNA in der Probe, die den Nanoroboter und überschüssiges Heftklammern, die während der Reinigung nicht ausgewaschen war. Reinigung Schätzungen werden von der relativen Intensität der Nanoroboter (rote und blaue Pfeile in 2A) an, dass der überschüssige Klammern (grüner Pfeil in 2B) berechnet. Die relativen Nanoroboter / Klammern Intensitäten wurden mit dem vorgereinigten Probe (Bahn 3), in der die gesamte DNA Gewicht der Klammern (grüner Pfeil) normiert ist ~ 4,5-mal derjenige des gereinigten Nanoroboter (roter Pfeil), entsprechend der Anfangs 10: 1 GerüstMolverhältnis Klammern in dem Herstellungsprotokoll.
Endgültige Validierung von strukturellen Integrität über Transmissionselektronenmikroskopie unter Verwendung von 2% Uranylacetat-Formiat als Negativfärbung erzielt. Bilder werden in Abbildung 3 dargestellt.
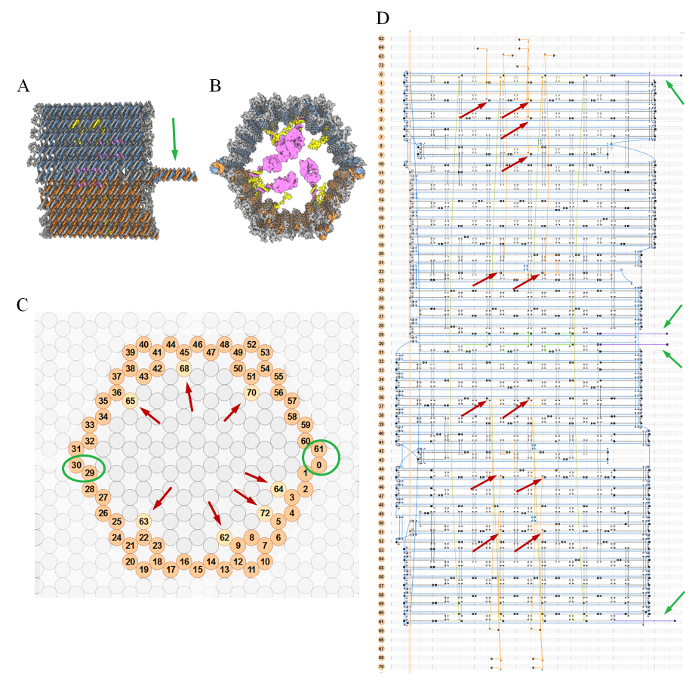
Abbildung 1: Entwerfen schematische Darstellung des Nanorobotik (A) Seitenansicht eines geschlossenen Nanorobotik.. Gate-Doppelhelix, von einem Aptamer-Sequenz und einem komplementären Strang erzeugt wird, wird durch einen grünen Pfeil angezeigt. (B) eine Vorderansicht eines geschlossenen Nanoroboter, die Protein-Ladung im Inneren gebunden. (C) Querschnitt schematische Darstellungen der geschlossenen Nanoroboter hergestellt. Verarbeitet Klammern werden mit einem roten Pfeil markiert. Grüne Kreise zeigen Wendeln, die ein Scharnier an einer Seite und ein Gate-Sequenz an der gegenüberliegenden Seite, enthalten. (D) von Blueprintsein Nanorobotik in der Seitenansicht. Griffe sind durch rote Pfeile gekennzeichnet. Gate-Sequenzen werden durch grüne Pfeile angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
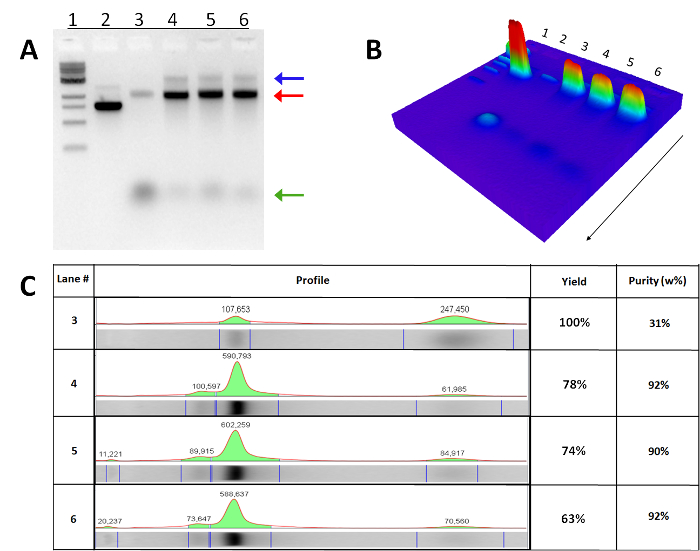
Abb. 2: Agarosegelelektrophorese Ergebnisse (A) Spur 1: 1kb Marker. Spur 2: M13mp18 kreisförmigen Einzelstrang-Gerüst. Spur 3: Nanoroboter Vorreinigung. Spuren 4-6: Nanoroboter zu posten Reinigungen. Spur 4: 50 & mgr; Anfangsvolumen; 2 Puffer Ergänzungen. Spur 5: 100 & mgr; Anfangsvolumen; 2 Puffer Ergänzungen. Spur 6: 100 & mgr; Anfangsvolumen; 5 Puffer Ergänzungen. Überschüssiges Klammern werden durch einen grünen Pfeil Fabricated Nanoroboter sind mit einem roten Pfeil angezeigt ist. Blauer Pfeil angezeigt Nanorobotik-Dimere. (B) in 3D-Ansicht der relativen Intensität der Banden in der AgaroseGel. (C) Ausbeute und Reinigung Schätzungen.
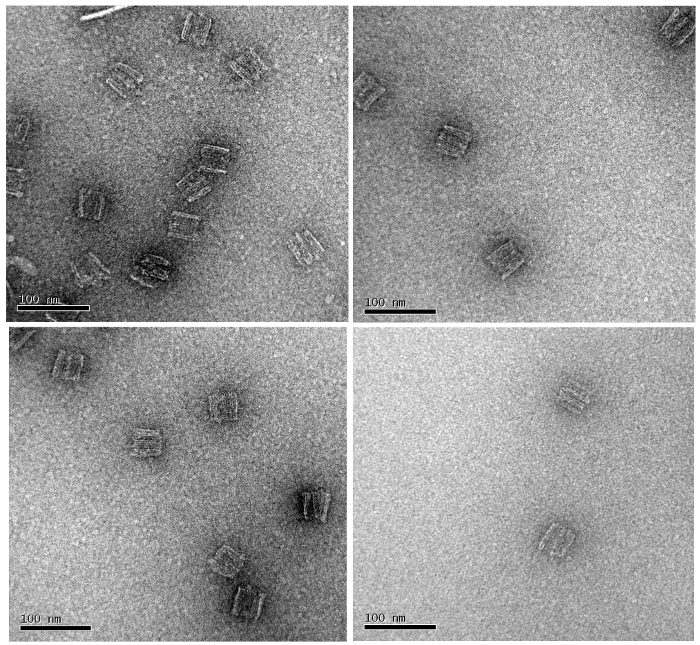
Abbildung 3: TEM Foto von DNA-Nanoroboter aus mehreren Erfindungen / Färbeverfahren gemacht.
| Anzahl | Bezeichnung | Sequenz | ||||||||||
| 1 | Ader | AAAAACCAAACCCTCGTTGTGAATATGGTTTGGTC | ||||||||||
| 2 | Ader | GGAAGAAGTGTAGCGGTCACGTTATAATCAGCAGACTGATAG | ||||||||||
| 3 | Ader | TACGATATAGATAATCGAACAACA | ||||||||||
| 4 | Ader | CTTTTGCTTAAGCAATAAAGCGAGTAGA | ||||||||||
| 5 | Ader | GTCTGAAATAACATCGGTACGGCCGCGCACGG | ||||||||||
| 6 | Ader | GGAAGAGCCAAACAGCTTGCAGGGAACCTAA | ||||||||||
| 7 | Ader | AAAATCACCGGAAGCAAACTCTGTAGCT | ||||||||||
| 8 | Ader | CCTACATGAAGAACTAAAGGGCAGGGCGGAGCCCCGGGC | ||||||||||
| 9 | Ader | CATGTAAAAAGGTAAAGTAATAAGAACG | ||||||||||
| 10 | Ader | ATTAAATCAGGTCATTGCCTGTCTAGCTGATAAATTGTAATA | ||||||||||
| 11 | Ader | ATAGTCGTCTTTTGCGGTAATGCC | ||||||||||
| 12 | Ader | AGTCATGGTCATAGCTGAACTCACTGCCAGT | ||||||||||
| 13 | Ader | AACTATTGACGGAAATTTGAGGGAATATAAA | ||||||||||
| 14 | Ader | ATCGCGTCTGGAAGTTTCATTCCATATAGAAAGACCATC | ||||||||||
| 15 | Ader | AAATATTGAACGGTAATCGTAGCCGGAGACAGTCATAAAAAT | ||||||||||
| 16 | Ader | GTCTTTACAGGATTAGTATTCTAACGAGCATAGAACGC | ||||||||||
| 17 | Ader | GCACCGCGACGACGCTAATGAACAGCTG | ||||||||||
| 18 | Ader | AACTTCATTTTAGAATCGCAAATC | ||||||||||
| 19 | Ader | CGTAGAGTCTTTGTTAAGGCCTTCGTTTTCCTACCGAG | ||||||||||
| 20 | Ader | CCAATCAAAGGCTTATCCGGTTGCTATT | ||||||||||
| 21 | Ader | AGAGGCGATATAATCCTGATTCATCATA | ||||||||||
| 22 | Ader | CCGTAATCCCTGAATAATAACGGAATACTACG | ||||||||||
| 23 | Ader | AAATGGTATACAGGGCAAGGAAATC | ||||||||||
| 24 | Ader | TCCTCATCGTAACCAAGACCGACA | ||||||||||
| 25 | Ader | CATTATCTGGCTTTAGGGAATTATGTTTGGATTAC | ||||||||||
| 26 | Ader | ACCCGCCCAATCATTCCTCTGTCC | ||||||||||
| 27 | Ader | CGACCAGTCACGCAGCCACCGCTGGCAAAGCGAAAGAAC | ||||||||||
| 28 | Ader | CTAAAGGCGTACTATGGTTGCAACAGGAGAGA | ||||||||||
| 29 | Ader | TTGGCAGGCAATACAGTGTTTCTGCGCGGGCG | ||||||||||
| 30 | Ader | |||||||||||
| 31 | Ader | AAGTATAGTATAAACAGTTAACTGAATTTACCGTTGAGCCAC | ||||||||||
| 32 | Ader | ACATTCAGATAGCGTCCAATATTCAGAA | ||||||||||
| 33 | Ader | AAACATCTTTACCCTCACCAGTAAAGTGCCCGCCC | ||||||||||
| 34 | Ader | GAGATGACCCTAATGCCAGGCTATTTTT | ||||||||||
| 35 | Ader | TCCTGAATTTTTTGTTTAACGATCAGAGCGGA | ||||||||||
| 36 | Ader | GCCGAAAAATCTAAAGCCAATCAAGGAAATA | ||||||||||
| 37 | Ader | AGCGTAGCGCGTTTTCACAAAATCTATGTTAGCAAACGAACGCAACAAA | ||||||||||
| 38 | Ader | ACCAATCGATTAAATTGCGCCATTATTA d> | ||||||||||
| 39 | Ader | ATCTTACTTATTTTCAGCGCCGACAGGATTCA | ||||||||||
| 40 | Ader | CCCTAAAAGAACCCAGTCACA | ||||||||||
| 41 | Ader | GGAAGGGCGAAAATCGGGTTTTTCGCGTTGCTCGT | ||||||||||
| 42 | Ader | CAGACCGGAAGCCGCCATTTTGATGGGGTCAGTAC | ||||||||||
| 43 | Ader | TAATATTGGAGCAAACAAGAGATCAATATGATATTGCCTTTA | ||||||||||
| 44 | Ader | TTCCTTATAGCAAGCAAATCAAATTTTA | ||||||||||
| 45 | Ader | ACTACGAGGAGATTTTTTCACGTTGAAACTTGCTTT | ||||||||||
| 46 | Ader | AAACAGGCATGTCAATCATATAGATTCAAAAGGGTTATATTT | ||||||||||
| 47 | AACAGGCACCAGTTAAAGGCCGCTTTGTGAATTTCTTA | |||||||||||
| 48 | Ader | TTCCTGAGTTATCTAAAATATTCAGTTGTTCAAATAGCAG | ||||||||||
| 49 | Ader | AAAGAAACAAGAGAAGATCCGGCT | ||||||||||
| 50 | Ader | TTGAGGGTTCTGGTCAGGCTGTATAAGC | ||||||||||
| 51 | Ader | TTTAACCGTCAATAGTGAATTCAAAAGAAGATGATATCGCGC | ||||||||||
| 52 | Ader | ACGAGCGCCCAATCCAAATAAAATTGAGCACC | ||||||||||
| 53 | Ader | AATAAGTCGAAGCCCAATAATTATTTATTCTT | ||||||||||
| 54 | Ader | ACGAAATATCATAGATTAAGAAACAATGGAACTGA | ||||||||||
| 55 | Ader | TTTCATAGTTGTACCGTAACACTGGGGTTTT | ||||||||||
| 56 | Ader | AGGAGCGAGCACTAACAACTAAAACCCTATCACCTAACAGTG | ||||||||||
| 57 | Ader | CAAAGTATTAATTAGCGAGTTTCGCCACAGAACGA | ||||||||||
| 58 | Ader | TGGGGAGCTATTTGACGACTAAATACCATCAGTTT | ||||||||||
| 59 | Ader | ATAACGCAATAGTAAAATGTTTAAATCA | ||||||||||
| 60 | Ader | ACGAATCAACCTTCATCTTATACCGAGG | ||||||||||
| 61 | Ader | TAATGGTTTGAAATACGCCAA | ||||||||||
| 62 | Ader | CGGAACAAGAGCCGTCAATAGGCACAGACAATATCCTCAATC | ||||||||||
| 63 | Ader | ATTAAAGGTGAATTATCAAAGGGCACCACGG | ||||||||||
| 64 | GGCAACCCATAGCGTAAGCAGCGACCATTAA | |||||||||||
| 65 | Ader | AGAAACGTAAGCAGCCACAAGGAAACGATCTT | ||||||||||
| 66 | Ader | AGAGGTCTTTAGGGGGTCAAAAGGCAGT | ||||||||||
| 67 | Ader | GGGGACTTTTTCATGAGGACCTGCGAGAATAGAAAGGAGGAT | ||||||||||
| 68 | Ader | TTTTAGAACATCCAATAAATCCAATAAC | ||||||||||
| 69 | Ader | AAATGTGGTAGATGGCCCGCTTGGGCGC | ||||||||||
| 70 | Ader | ACGGATCGTCACCCTCACGATCTAGAATTTT | ||||||||||
| 71 | Ader | CGCCATAAGACGACGACAATAGCTGTCT | ||||||||||
| 72 | Ader | GCGTATTAGTCTTTAATCGTAAGAATTTACEIN | ||||||||||
| 73 | Ader | AGAGAACGTGAATCAAATGCGTATTTCCAGTCCCC | ||||||||||
| 74 | Ader | AACGAAAAAGCGCGAAAAAAAGGCTCCAAAAGG | ||||||||||
| 75 | Ader | TAATTTAGAACGCGAGGCGTTAAGCCTT | ||||||||||
| 76 | Ader | ACCAGGCGTGCATCATTAATTTTTTCAC | ||||||||||
| 77 | Ader | CAGCCTGACGACAGATGTCGCCTGAAAT | ||||||||||
| 78 | Ader | ATTAGTCAGATTGCAAAGTAAGAGTTAAGAAGAGT | ||||||||||
| 79 | Ader | CTCGAATGCTCACTGGCGCAT | ||||||||||
| 80 | Ader | GGGCAGTCACGACGTTGAATAATTAACAACC | ||||||||||
| 81 | Ader | TAAAAACAGGGGTTTTGTTAGCGAATAATATAATAGAT | ||||||||||
| 82 | Ader | TCAACCCTCAGCGCCGAATATATTAAGAATA | ||||||||||
| 83 | Ader | ATTATACGTGATAATACACATTATCATATCAGAGA | ||||||||||
| 84 | Ader | GCAAATCTGCAACAGGAAAAATTGC | ||||||||||
| 85 | Ader | ATAATTACTAGAAATTCTTAC | ||||||||||
| 86 | Ader | TATCACCGTGCCTTGAGTAACGCGTCATACATGGCCCCTCAG | ||||||||||
| 87 | Ader | AAGTAGGGTTAACGCGCTGCCAGCTGCA | ||||||||||
| 88 | Ader | CCAGTAGTTAAGCCCTTTTTAAGAAAAGCAAA | ||||||||||
| 89 | Ader | TGGCGAAGTTGGGACTTTCCG | ||||||||||
| 90 | CAGTGAGTGATGGTGGTTCCGAAAACCGTCTATCACGATTTA | |||||||||||
| 91 | Ader | AAATCAAAGAGAATAACATAACTGAACACAGT | ||||||||||
| 92 | Ader | CTGTATGACAACTAGTGTCGA | ||||||||||
| 93 | Ader | ATCATAAATAGCGAGAGGCTTAGCAAAGCGGATTGTTCAAAT | ||||||||||
| 94 | Ader | TTGAGTAATTTGAGGATTTAGCTGAAAGGCGCGAAAGATAAA | ||||||||||
| 95 | Ader | ATAAGAATAAACACCGCTCAA | ||||||||||
| 96 | Ader | CGTTGTAATTCACCTTCTGACAAGTATTTTAA | ||||||||||
| 97 | Ader | AACCGCCTCATAATTCGGCATAGCAGCA | ||||||||||
| 98 | Ader | AAATAGGTCACGTTGGTAGCGAGTCGCGTCTAATTCGC | ||||||||||
| 99 | Ader | CAGTATAGCCTGTTTATCAACCCCATCC | ||||||||||
| 100 | Ader | TTGCACCTGAAAATAGCAGCCAGAGGGTCATCGATTTTCGGT | ||||||||||
| 101 | Ader | CGTCGGAAATGGGACCTGTCGGGGGAGA | ||||||||||
| 102 | Ader | AAGAAACTAGAAGATTGCGCAACTAGGG | ||||||||||
| 103 | Ader | CCAGAACCTGGCTCATTATACAATTACG | ||||||||||
| 104 | Ader | ACGGGTAATAAATTAAGGAATTGCGAATAGTA | ||||||||||
| 105 | Ader | CCACGCTGGCCGATTCAAACTATCGGCCCGCT | ||||||||||
| 106 | Ader | GCCTTCACCGAAAGCCTCCGCTCACGCCAGC | ||||||||||
| 107 | CAGCATTAAAGACAACCGTCAAAAATCA | |||||||||||
| 108 | Ader | ACATCGGAAATTATTTGCACGTAAAAGT | ||||||||||
| 109 | Ader | CAACGGTCGCTGAGGCTTGATACCTATCGGTTTATCAGATCT | ||||||||||
| 110 | Ader | AAATCGTACAGTACATAAATCAGATGAA | ||||||||||
| 111 | Ader | TTAACACACAGGAACACTTGCCTGAGTATTTG | ||||||||||
| 112 | Ader | AGGCATAAGAAGTTTTGCCAGACCCTGA | ||||||||||
| 113 | Ader | GACGACATTCACCAGAGATTAAAGCCTATTAACCA | ||||||||||
| 114 | Ader | AGCTGCTCGTTAATAAAACGAGAATACC | ||||||||||
| 115 | Ader | CTTAGAGTACCTTTTAAACAGCTGCGGAGATTTAGACTA | ||||||||||
| 116 | Ader | CACCCTCTAATTAGCGTTTGCTACATAC | ||||||||||
| 117 | Ader | GAACCGAAAATTGGGCTTGAGTACCTTATGCGATTCAACACT | ||||||||||
| 118 | Ader | GCAAGGCAGATAACATAGCCGAACAAAGTGGCAACGGGA | ||||||||||
| 119 | Ader | ATGAAACAATTGAGAAGGAAACCGAGGATAGA | ||||||||||
| 120 | Ader | GGATGTGAAATTGTTATGGGGTGCACAGTAT | ||||||||||
| 121 | Ader | GGCTTGCGACGTTGGGAAGAACAGATAC | ||||||||||
| 122 | Ader | TAAATGCCTACTAATAGTAGTTTTCATT | ||||||||||
| 123 | Ader | TGCCGTCTGCCTATTTCGGAACCAGAATGGAAAGCCCACCAGAAC | ||||||||||
| 124 | Ader | TGACCATAGCAAAAGGGAGAACAAC | ||||||||||
| 125 | Ader | CGAGCCAGACGTTAATAATTTGTATCA | ||||||||||
| 126 | Ader | GCTCAGTTTCTGAAACATGAAACAAATAAATCCTCCCGCCGC | ||||||||||
| 127 | Ader | AGACGCTACATCAAGAAAACACTTTGAA | ||||||||||
| 128 | Ader | AGTACTGACCAATCCGCGAAGTTTAAGACAG | ||||||||||
| 129 | Ader | GATTCCTGTTACGGGCAGTGAGCTTTTCCTGTGTGCTG | ||||||||||
| 130 | Ader | GGTATTAAGGAATCATTACCGAACGCTA | ||||||||||
| 131 | Ader | GTTCATCAAATAAAACGCGACTCTAGAGGATCGGG | ||||||||||
| 132 | Ader | AGCCTTTAATTGGATAGTTGAACCGCCACCCTCATAGGTG | ||||||||||
| 133 | Ader | ACAGAGGCCTGAGATTCTTTGATTAGTAATGG | ||||||||||
| 134 | Ader | AACGAGATCAGGATTAGAGAGCTTAATT | ||||||||||
| 135 | Ader | TACCAAGTTATACTTCTGAATCACCAGA | ||||||||||
| 136 | Ader | CAGTAGGTGTTCAGCTAATGCGTAGAAA | ||||||||||
| 137 | Ader | AGGATGACCATAGACTGACTAATGAAATCTACATTCAGCAGGCGCGTAC | ||||||||||
| 138 | Ader | TTTCAACCAAGGCAAAGAATTTAGATAC | ||||||||||
| 139 | Ader | TTGAAATTAAGATAGCTTAACTAT | ||||||||||
| 140 | Ader | CTATTATCGAGCTTCAAAGCGTATGCAA | ||||||||||
| 141 | Ader | CAGGGTGCAAAATCCCTTATAGACTCCAACGTCAAAAGCCGG | ||||||||||
| 142 | Ader | GAGCTTGTTAATGCGCCGCTAATTTTAGCGCCTGCTGCTGAA | ||||||||||
| 143 | Ader | CGAACGTTAACCACCACACCCCCAGAATTGAG | ||||||||||
| 144 | Ader | GTGTGATAAATAAGTGAGAAT | ||||||||||
| 145 | Ader | GCTATATAGCATTAACCCTCAGAGA | ||||||||||
| 146 | Ader | AGGAGAGCCGGCAGTCTTGCCCCCGAGAGGGAGGG | ||||||||||
| 147 | Ader | CGGCCTCCAGCCAGAGGGCGAGCCCCAA | ||||||||||
| 148 | Ader | CCAAAACAAAATAGGCTGGCTGACGTAACAA | ||||||||||
| 149 | Ader | GGCGGTTAGAATAGCCCGAGAAGTCCACTATTAAAAAGGAAG | ||||||||||
| 150 | Ader | ATAAAGGTTACCAGCGCTAATTCAAAAACAGC | ||||||||||
| 151 | Ader | ATTGCCCCCAGCAGGCGAAAAGGCCCACTACGTGACGGAACC | ||||||||||
| 152 | Ader | TTTTAAAACATAACAGTAATGGAACGCTATTAGAACGC | ||||||||||
| 153 | Ader | AATTGGGTAACGCCAGGCTGTAGCCAGCTAGTAAACGT | ||||||||||
| 154 | Rand | TTACCCAGAACAACATTATTACAGGTTTTTTTTTTTTTTTT | ||||||||||
| 155 | Rand | TTTTTTTTTTTTTTTTAATAAGAGAATA | ||||||||||
| 156 | Rand | TTTTTTTTTTTTTTTTCCAGTTTGGGAGCGGGCTTTTTTTTTTTTTTT | ||||||||||
| 157 | Rand | GGTTGAGGCAGGTCAGTTTTTTTTTTTTTTT | ||||||||||
| 158 | Rand | TTTTTTTTTTTTTTTGATTAAGACTCCTTATCCAAAAGGAAT | ||||||||||
| 159 | Rand | TTTTTTTTTTTTTTTTCTTCGCTATTACAATT | ||||||||||
| 160 | Rand | TTTTTTTTTTTTTTTCTTGCGGGAGAAGCGCATTTTTTTTTTTTTTTT | ||||||||||
| 161 | Rand | TTTTTTTTTTTTTTGGGAATTAGAGAAACAATGAATTTTTTTTTTTTTTT | ||||||||||
| 162 | Rand | TCAGACTGACAGAATCAAGTTTGTTTTTTTTTTTTTTT | ||||||||||
| 163 | Rand | TTTTTTTTTTTTTTTGGTCGAGGTGCCGTAAAGCAGCACGT | ||||||||||
| 164 | Rand | TTTTTTTTTTTTTTTTTTAATCATTTACCAGACTTTTTTTTTTTTTTT | ||||||||||
| 165 | Rand | TTTTTTTTTTTTTCATTCTGGCCAAATTCGACAACTCTTTTTTTTTTTTT | ||||||||||
| 166 | Rand | TTTTTTTTTTTTTTTACCGGATATTCA | ||||||||||
| 167 | Rand | TTTTTTTTTTTTTTTTAGACGGGAAACTGGCATTTTTTTTTTTTTTTT | ||||||||||
| 168 | Rand | TTTTTTTTTTTTTTTCAGCAAGCGGTCCACGCTGCCCAAAT | ||||||||||
| 169 | Rand | CTGAGAGAGTTGTTTTTTTTTTTTTTT | ||||||||||
| 170 | Rand | CAATGACAACAACCATTTTTTTTTTTTTTTT | ||||||||||
| 171 | Rand | TTTTTTTTTTTTTTTTGAGAGATCTACAAGGAGAGG | ||||||||||
| 172 | Rand | TCACCAGTACAAACTATTTTTTTTTTTTTTT | ||||||||||
| 173 | Rand | TTTTTTTTTTTTTGGCAATTCATCAAATTATTCATTTTTTTTTTTTTTTT | ||||||||||
| 174 | Rand | TAAAGTTACCGCACTCATCGAGAACTTTTTTTTTTTTTTT | ||||||||||
| 175 | Rand | TTTTTTTTTTTTTTTCACCCTCAGAACCGCC | ||||||||||
| 176 | Rand | TTTTTTTTTTTTTAGGTTTAACGTCAATATATGTGAGTTTTTTTTTTTTT | ||||||||||
| 177 | Rand | CCACACAACATACGTTTTTTTTTTTTT | ||||||||||
| 178 | Rand | TTTTTTTTTTTTTTTGCTAGGGCGAGTAAAAGATTTTTTTTTTTTTTT | ||||||||||
| 179 | Rand | TTTTTTTTTTTTTTTAGTTGATTCCCAATTCTGCGAACCTCA | ||||||||||
| 180 | Rand | TTATTTAGAGCCTAATTTGCCAGTTTTTTTTTTTTTTTTT | ||||||||||
| 181 | Rand | TTTTTTTTTTTTTTTACGGCGGAT | ||||||||||
| 182 | Rand | TTTTTTTTTTTTTTTATATGCGTTAAGTCCTGATTTTTTTTTTTTTTT | ||||||||||
| 183 | Rand | TTTTTTTTTTTTTTTACGATTGGCCTTGATA | ||||||||||
| 184 | Rand | TTTTTTTTTTTTTTTCAACGCCTGTAGCATT | ||||||||||
| 185 | Rand | TTTTTTTTTTTTTTTGGCTTTGAGCCGGAACGATTTTTTTTTTTTTTT | ||||||||||
| 186 | Rand | TTTTTTTTTTTTTTTAAGCAAGCCGTTT | ||||||||||
| 187 | Rand | TTTTTTTTTTTTTATGTGTAGGTAAGTACCCCGGTTGTTTTTTTTTTTTT | ||||||||||
| 188 | Rand | ATCGTCATAAATATTCATTTTTTTTTTTTTTTTT | ||||||||||
| 189 | Rand | |||||||||||
| 190 | Rand | TTTTTTTTTTTTTGTATTAAATCCTGCGTAGATTTTCTTTTTTTTTTTTT | ||||||||||
| 191 | Rand | GCCATATAAGAGCAAGCCAGCCCGACTTGAGCCATGGTT | ||||||||||
| 192 | Rand | GTAGCTAGTACCAAAAACATTCATAAAGCTAAATCGGTTTTTTTTTTTTT | ||||||||||
| 193 | Rand | ATAACGTGCTTTTTTTTTTTTTTTTTT | ||||||||||
| 194 | Rand | TTTTTTTTTTTTTTTAAAATACCGAACGAACCACCAGTGAGAATTAAC | ||||||||||
| 195 | Rand | TTTTTTTTTTTTTTTACAAAATAAACA | ||||||||||
| 196 | Rand | TTTTTTTTTTTTTTTACAAGAAAAACCTCCCGATTTTTTTTTTTTTTT | ||||||||||
| 197 | Rand | TTTTTTTTTTTTTTTGACGATAAAAAGATTAAGTTTTTTTTTTTTTTT | ||||||||||
| 198 | Rand | TTTTTTTTTTTTTCAATTACCTGAGTATCAAAATCATTTTTTTTTTTTTT | ||||||||||
| 199 | Rand | GGTACGGCCAGTGCCAAGCTTTTTTTTTTTTTTT | ||||||||||
| 200 | Rand | TTTTTTTTTTTTTTGAATAACCTTGAAATATATTTTATTTTTTTTTTTTT | ||||||||||
| 201 | Rand | CACTAAAACACTTTTTTTTTTTTTTTT | ||||||||||
| 202 | Rand | TTTTTTTTTTTTTTTTAACCAATATGGGAACAATTTTTTTTTTTTTTT | ||||||||||
| 203 | Rand | TACGTCACAATCAATAGAATTTTTTTTTTTTTTT | ||||||||||
| 204 | Rand | TTTTTTTTTTTTTTTAGAAAGATTCATCAGTTGA | ||||||||||
| 205 | Rand | TTTTTTTTTTTTTGTGGCATCAATTAATGCCTGAGTATTTTTTTTTTTTT | ||||||||||
| 206 | Rand | TTTTTTTTTTTTTTTTTGCATGCCTGCATTAATTTTTTTTTTTTTTTT | ||||||||||
| 207 | Rand | CCAGCGAAAGAGTAATCTTGACAAGATTTTTTTTTTTTTTT | ||||||||||
| 208 | Rand | TTTTTTTTTTTTTTTGAATCCCCCTCAAATGCTT | ||||||||||
| 209 | Rand | AGAGGCTGAGACTCCTTTTTTTTTTTTTTTT | ||||||||||
| 210 | Rand | ACAAACACAGAGATACATCGCCATTATTTTTTTTTTTTTTT | ||||||||||
| 211 | Rand | TTTTTTTTTTTTTTTCAAGAGAAGGATTAGG | ||||||||||
| 212 | Rand | TTTTTTTTTTTTTGAATTGAGGAAGTTATCAGATGATTTTTTTTTTTTTT | ||||||||||
| 213 | Kanten | CAGAACAATATTTTTTTTTTTTTTTTT | ||||||||||
| 214 | Rand | TTTTTTTTTTTTTAGCCGGAAGCATAAAGTGTCCTGGCC | ||||||||||
| 215 | Rand | TGACCGTTTCTCCGGGAACGCAAATCAGCTCATTTTTTTTTTTTTTTTTT | ||||||||||
| 216 | Rand | TTTTTTTTTTTTTTTGGTAATAAGTTTTAAC | ||||||||||
| 217 | Rand | TTTTTTTTTTTTTTGTCTGTCCATAATAAAAGGGATTTTTTTTTTTTTTT | ||||||||||
| 218 | Rand | TTTTTTTTTTTTTTTCCTCGTTAGAATCAGAGCGTAATATC | ||||||||||
| 219 | Rand | AATTGCTCCTTTTGATAAGTTTTTTTTTTTTTTT | ||||||||||
| 220 | Rand | CATCGGACAGCCCTGCTAAACAACTTTCAACAGTTTTTTTTTTTTTTT | ||||||||||
| 221 | TTTTTTTTTTTTTTTAACCGCCTCCCTCAGACCAGAGC | |||||||||||
| 222 | Rand | TCTGACAGAGGCATTTTCGAGCCAGTTTTTTTTTTTTTTT | ||||||||||
| 223 | Rand | TTTTTTTTTTTTTTTTTTCAGCGGAGTTCCATGTCATAAGG | ||||||||||
| 224 | Rand | TTTTTTTTTTTTTTTCGCCCACGCATAACCG | ||||||||||
| 225 | Rand | AATTACTTAGGACTAAATAGCAACGGCTACAGATTTTTTTTTTTTTTT | ||||||||||
| 226 | Rand | CAAGTTTTTTGGTTTTTTTTTTTTTTT | ||||||||||
| 227 | Rand | TTTTTTTTTTTTTTTCCTTTAGCGCACCACCGGTTTTTTTTTTTTTTT | ||||||||||
| 228 | Rand | TTTTTTTTTTTTTTTGAATCGGCCGAGTGTTGTTTTTTTTTTTTTTTT | ||||||||||
| 229 | Rand | TTTTTTTTTTTTTCATCTTTGACCC | ||||||||||
| 230 | Rand | TTTTTTTTTTTTTATAATCAGAAAATCGGTGCGGGCCTTTTTTTTTTTTT | ||||||||||
| 231 | Rand | GATACAGGAGTGTACTTTTTTTTTTTTTTTT | ||||||||||
| 232 | Rand | TTTTTTTTTTTTTTTGGCGCAGACAATTTCAACTTTTTTTTTTTTTTT | ||||||||||
| 233 | Rand | GGAGGTTTAGTACCGCTTTTTTTTTTTTTTT | ||||||||||
| 234 | Rand | TTTTTTTTTTTTTACCGCCAGCCATAACAGTTGAAAGTTTTTTTTTTTTT | ||||||||||
| 235 | Rand | TTTTTTTTTTTTTTTATAGCAATAGCT | ||||||||||
| 236 | Griffe | AATAAGTTTTGCAAGCCCAATAGGGGATAAGTTGTGCTACTCCAGTTC | ||||||||||
| 237 | Griffe | ACATAGCTTACATTTAACAATAATAACGTTGTGCTACTCCAGTTC | ||||||||||
| 238 | Griffe | CCTTTTTGAATGGCGTCAGTATTGTGCTACTCCAGTTC | ||||||||||
| 239 | Griffe | CGTAACCAATTCATCAACATTTTGTGCTACTCCAGTTC | ||||||||||
| 240 | Griffe | CACCAACCGATATTCATTACCATTATTGTGCTACTCCAGTTC | ||||||||||
| 241 | Griffe | CCACCCTCATTTTCTTGATATTTGTGCTACTCCAGTTC | ||||||||||
| 242 | Griffe | AACTTTGAAAGAGGAGAAACATTGTGCTACTCCAGTTC | ||||||||||
| 243 | Griffe | CAAGGCGCGCCATTGCCGGAATTGTGCTACTCCAGTTC | ||||||||||
| 244 | Griffe | CATAGCCCCCTTAAGTCACCATTGTGCTACTCCAGTTC | ||||||||||
| 245 | Griffe | TTTCCCTGAATTACCTTTTTTACCTTTTTTGTGCTACTCCAGTTC | ||||||||||
| 246 | Griffe | AACGGTGTACAGACTGAATAATTGTGCTACTCCAGTTC | ||||||||||
| 247 | Griffe | GATTCGCGGGTTAGAACCTACCATTTTGTTGTGCTACTCCAGTTC | ||||||||||
| 248 | Guides | AGAGTAGGATTTCGCCAACATGTTTTAAAAACC | ||||||||||
| 249 | Guides | ACGGTGACCTGTTTAGCTGAATATAATGCCAAC | ||||||||||
| 250 | Guides | CGTAGCAATTTAGTTCTAAAGTACGGTGTTTTA | ||||||||||
| 251 | Guides | GCTTAATGCGTTAAATGTAAATGCTGATCTTGAAATGAGCGTT | ||||||||||
| 252 | Guides | AAGCCAACGGAATCTAGGTTGGGTTATATAGATTAAGCAACTG | ||||||||||
| 253 | Guides | TTTAACAACCGACCCAATCGCAAGACAAAATTAATCTCACTGC | ||||||||||
| 254 | Guides | TTTAGGCCTAAATTGAGAAAACTTTTTCCTTCTGTTCCTAGAT | ||||||||||
| 255 | Guides Entfernen | GGTTTTTAAAACATGTTGGCGAAATCCTACTCT | ||||||||||
| 256 | Guides Entfernen | GTTGGCATTATATTCAGCTAAACAGGTCACCGT | ||||||||||
| 257 | Guides Entfernen | TAAAACACCGTACTTTAGAACTAAATTGCTACG | ||||||||||
| 258 | Guides Entfernen | AACGCTCATTTCAAGATCAGCATTTACATTTAACGCATTAAGC | ||||||||||
| 259 | Guides Entfernen | CAGTTGCTTAATCTATATAACCCAACCTAGATTCCGTTGGCTT | ||||||||||
| 260 | Guides Removal | GCAGTGAGATTAATTTTGTCTTGCGATTGGGTCGGTTGTTAAA | ||||||||||
| 261 | Guides Entfernen | ATCTAGGAACAGAAGGAAAAAGTTTTCTCAATTTAGGCCTAAA | ||||||||||
| 262 | Gates | Gate29 | TGGGGCGCGAGCTGAAAAGTACTCAGGGCACTGCAAGCAATTGTGGTCCCAATGGGCTGAGTA | |||||||||
| 263 | Gates | Gate30 | TACTCAGCCCATTGGGTTTTTTTTTTTTTTTTTTTTTTTTTTTTAGGTCTGAGAGACTACCTT | |||||||||
| 264 | Gates | Gate0 | TGATGAGCGTGGATGATACTCAGCCCATTGGGTTTTTTTTTTTTTTTTTTTTTTTTTTTTAGGTCATTTTTGCGGATGG | |||||||||
| 265 | Gates | Gate61 | ATACAAAAAGCCTGTTTAGTATCTACTCAGGGCACTGCAAGCAATTGTGGTCCCAATGGGCTGAGTA | |||||||||
Tabelle 1: Liste der Stapelsequenzenverwendet, um die Nanoroboter zu konstruieren. Staples 1-153 bezeichnet sind Kern und machen den Großteil der Struktur. Klammern 154-235 werden Kanten bezeichnet und werden an jedem der Enden der Wendeln 61 der Struktur entfernt. Rand Klammern enthalten eine Poly Thymin Schwanz zur Aggregation von Nanoroboter zu vermeiden. Staples 236-247 sind Griffe bezeichnet und bilden die Fracht Andockstellen. Griffe Klammern haben einen einzigartigen Sequenzbereich, verbindet sie den Aufstellungsort der Struktur und einer Konsensussequenzbereich, der als Andockstelle für den Cargomoleküle verwendet wird. Staples 248-254 bezeichnet sind Guides und befestigen Sie die beiden Hälften der Vorrichtung während des Wärmebehandlungsprozesses. Nach der Herstellung sind die Führungen mit dem Zusatz von Handbuch Removal Klammern 255-261 (Schritt 6) entfernt, so dass das Gerät von den beiden Sensoren des Nanorobotik gesperrt. Sensor-Sequenzen werden Tore bezeichnet. Jeder der zwei Sensoren ist aus einer PDGF-Aptamer und einem komplementären Strang hergestellt. Für eine detailliertere Sälen mit KaminNation sehen, Ben-Ishay et al. 14 und Douglas et al. 15. Staples werden kommerziell auf drei 96-Loch-Platten (tiefe Rund) bestellt. Jede Klammer Menge auf 10 nmol normalisiert. Mit Ausnahme der Gate-Sequenzen, die HPLC-Reinigung erfordern, müssen Klammern nicht ein spezielles Reinigungsverfahren erforderlich.
Diskussion
Wir beschrieben die Herstellung, Reinigung und Visualisierung der DNA-Nanoroboter. Nach der Herstellung des hexagonalen Chassis der Vorrichtung wird die Funktion des Nanoroboters mit der einfachen Einführung spezifischer Ladung und Erfassen Stränge an den Roboter, die leicht zu finden ihre bestimmten Position aufgrund von Wasserstoffbrücken Komplementarität vorhanden einsträngigen Andockstellen 14 programmiert , 15,22.
Die beschriebenen Herstellungsprotokoll verwendet eine langsame Glühen Rampe, die in der Regel in unserem Labor verwendet wird, um eine breite Palette von Origami-Formen zu falten. Wenn die Produktionszeit wird zu einem Schlüsselfaktor für andere Protokolle, wie beispielsweise die durch Sobczak et al schnelle Faltung Protokoll. 24 verwendet werden. Dieses Protokoll wird berichtet, dass Origami Falte in hohen Ausbeuten zu erreichen, aber es ist die Kalibrierung für jede Origami Form erfordert.
Spin Filtration wird verwendet, um die Roboter aus gestaute Klammern reinigen. Beim Laden der spin-Säule mit Proben oder Puffer, sollte darauf geachtet werden, dass die Membran mit der Pipettenspitze beschädigt wird. Die Membran kann möglicherweise platzen, was zu drastisch reduziert Erträge. Es ist ratsam, den Durchlauf nicht zu verwerfen, bis die Nanoroboter sind nach Alter visualisiert.
Für bestimmte Anwendungen eine höhere Reinigungsrate gewünscht wird; Dies kann durch Wiederholung der Filtration unter Verwendung des Original-Spin-Säule erreicht werden. Für eine noch höhere Reinheit kann ein neues Spin-Säule verwendet werden, jedoch wird dies eine dramatische negative Auswirkungen auf die Renditen haben. Andere Verfahren zur Reinigung von DNA origami Strukturen wurden getestet, wie Ausschneiden aus Agarosegel-Elektrophorese und anschließend an die Dialyse von überschüssigem Heftklammern. Diese Verfahren führten entweder schlechte Ausbeute oder schlechte Reinigungsraten gegenüber dem beschriebenen Protokoll. Andere Verfahren, wie beispielsweise PEG-basierte Reinigungs 25 und geschwindigkeits zonale Ultrazentrifugation 26 wurden nicht getestet. Diese Verfahren sind berichtet ACHIeve hohe Reinigungsraten, aber sie entweder in schlechten Ausbeuten (Rate-zonale Ultrazentrifugation) bzw. es muss Niederschlag (PEG-Basis), die den hohlen Nanorobotik Form potenziell schaden können.
Nanoroboter sind mit Führungsklammern, die die Form in der geschlossenen Position, um Herstellungs yeilds 15 erhöhen verriegeln hergestellt. Es ist wichtig, diese Klammern durch Zugabe Leitfaden Removal Heftklammern für Nanoroboter in Reaktion auf die Reize ausgelegt tatsächlich offen (PDGF in dem Protokoll beschrieben) zu entfernen. 1 Molverhältnis am Ende der Reinigung der Überschuß: Führungsklammern sind mit einem Brückenkopf-Einzelstrangbereich für das Andocken von Führungs Removal Heftklammern, die die Führungsklammern durch einen Prozess der Strangverdrängung 18 .Diese Tackerklammern bei 10 zugegeben werden frei entworfen Staples Stufe (Stufe 4.7) und für 2 h bei Raumtemperatur inkubiert auf einem über Kopf geschüttelt.
Visualisierung mit TEM wurde beschrieben, including-Uranyl Formiat 2% negative Färbung. Verglichen mit Uranyl-Acetat, Uranyl-Formiat erzeugt feinere Kornstrukturen, die für eine bessere Auflösung der DNA-Origami-Designs zu ermöglichen. Man sollte bei Uranylacetat-Formiat genommen wird verfestigt, wenn Aliquots werden für längere Zeit eingefroren werden. Es ist ratsam, eine frisch hergestellte 2% Uranylacetat-Formiatlösung zum Färben verwendet werden. Bessere Auflösungen erzielbar sind mit der Verwendung von Kryo-TEM 27.
Die grundlegende architektonische Gestaltung und Herstellung Protokoll der DNA-Nanoroboter sind letztlich gleichmäßige und unkompliziert. Jedoch wird eine große Flexibilität in Form von spezifischen Fracht Mischungen und Sensorstränge angeboten. Weiterhin werden in einer einzigen Population vieler Subtypen eingeführt werden kann und deren relative Stöchiometrie programmiert und optimiert werden, um spezifische Anforderungen. Noch mehr Feinabstimmung der spezifischen Kinetik an jedem geschwindigkeitsbestimmende Schritt ist erreichbar mit zunehmender / Verringerung der Länge der Doppelstrang-Hybridisierung oderEinführung der Nichtübereinstimmungen zu bestimmten Schlüsselpositionen. Diese hohe Flexibilität bei einfacher Konstruktion ermöglicht das Engineering von Nanometer-Bauteilen der Lage ist, sehr unterschiedlich komplexe Aufgaben in einem biologischen Milieu relevant.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Die Autoren danken S. Douglas für äußerst wertvolle Diskussionen und Beratung, und alle Mitglieder der Bachelet Labor für hilfreiche Diskussionen und Arbeit danken. Diese Arbeit wird durch Zuschüsse von der Fakultät für Lebenswissenschaften und Institut für Nanotechnologie und Neue Materialien an der Bar-Ilan-Universität unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| DNase/RNase free distilled water | Gibco | 10977 | |
| M13mp18 ssDNA scaffold | NEB | N4040S | |
| 10x TAE | Gibco | 15558-042 | |
| 1 M MgCl2 | Ambion | AM9530G | |
| Amicon Ultra 0.5 ml centrifugal filter 100K MWCO | Amicon | UFC510024 | |
| Agarose | Promega | V3125 | |
| TBE buffer | Promega | V4251 | |
| Ethidium bromide 10 mg/ml solution | Sigma Aldrich | E1510 | |
| 1 kb DNA marker | NEB | N3232S | |
| Loading Dye | NEB | B7021S | |
| uranyl formate | polysciences | 24762 | |
| carbon-coated TEM grids | Science services | EFCF400-Cu-50 | |
| Thermal Cycler c1000 Touch | Bio-Rad | ||
| Glow Discharge K100X | Emitech | ||
| UV table Gel Doc EZ Imager | Bio-Rad | ||
| NanoDrop 2000c | Thermo Scientific | ||
| TEM FEI-G12 | Tecnai |
Referenzen
- Watson, J. D., Crick, F. H. Genetical implications of the structure of deoxyribonucleic acid. Nature. 171, 964-967 (1953).
- Kosuri, S., Church, G. M. Large-Scale de novo. DNA synthesis: technologies and applications. Nature Meth. 11 (5), 499-507 (2014).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature Nanotech. 6 (12), 763-772 (2011).
- Seeman, N. C. Nucleic acid junctions and lattices. J Theor Biol. 99 (2), 237-247 (1982).
- Chen, J. H., Seeman, N. C. Synthesis from DNA of a molecule with the connectivity of a cube. Nature. 350 (6319), 631-633 (1991).
- Wei, B., Dai, M., Yin, P. Complex shapes self-assembled from single-stranded DNA tiles. Nature. 485 (7400), 623-626 (2012).
- He, Y., et al. Hierarchical self-assembly of DNA into symmetric supramolecular polyhedra. Nature. 452 (7184), 198-201 (2008).
- Yin, P., Hariadi, R. F., Sahu, S., Choi, H. M. T., Park, S. H., Labean, T. H., Reif, J. H. Programming DNA tube circumferences. Science. 321 (5890), 824-826 (2008).
- Rothemund, P. W. Folding DNA to create nanoscale shapes and patterns. Nature. 440 (7082), 297-302 (2006).
- Dietz, H., Douglas, S. M., Shih, W. M. Folding DNA into twisted and curved nanoscale shapes. Science. 325 (5941), 725-730 (2009).
- Douglas, S. M., et al. Self-assembly of DNA into nanoscale three-dimensional shapes. Nature. 459 (7245), 414-418 (2009).
- Zhang, F., Nangreave, J., Liu, Y., Yan, H. Structural DNA nanotechnology: state of the art and future perspective. J Am Chem Soc. 136 (32), 11198-11211 (2014).
- Douglas, S. M., et al. Prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Res. 37 (15), 5001-5006 (2009).
- Ben-Ishay, E., Abu-Horowitz, A., Bachelet, I. Designing a bio-responsive robot from DNA origami. J Vis Exp. (77), e50268 (2013).
- Douglas, S. M., Bachelet, I., Church, G. M. A logic-gated nanorobot for targeted transport of molecular payloads. Science. 335 (6070), 831-834 (2012).
- Tan, W., Donovan, M. J., Jiang, J. Aptamers from cell-based selection for bioanalytical applications. Chem Rev. 113 (4), 2842-2862 (2013).
- Xiang, D., et al. Nucleic Acid Aptamer-Guided Cancer Therapeutics and Diagnostics: The Next Generation of Cancer Medicine. Theranostics. 5 (1), 23-42 (2015).
- Zhang, D. Y., Seelig, G. Dynamic DNA nanotechnology using strand-displacement reactions. Nat Chem. 3 (2), 103-113 (2011).
- Tang, Z., Parekh, P., Turner, P., Moyer, R. W., Tan, W. Generating aptamers for recognition of virus-infected cells. Clin Chem. 55 (4), 813-822 (2009).
- Sefah, K., Shangguan, D., Xiong, X., O'Donoghue, M. B., Tan, W. Development of DNA aptamers using Cell-SELEX. Nature Prot. 5 (6), 1169-1185 (2010).
- McKeague, M., DeRosa, M. C. Challenges and Opportunities for Small Molecule Aptamer Development. J Nucleic Acids. 2012, (2012).
- Amir, Y., et al. Universal computing by DNA origami robots in a living animal. Nature Nanotech. 9 (5), 353-357 (2014).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature Meth. 8 (3), 221-229 (2011).
- Sobczak, J. P., Martin, T. G., Gerling, T., Dietz, H. Rapid folding of DNA into nanoscale shapes at constant temperature. Science. 338 (6113), 1458-1461 (2012).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and scalable preparation of pure and dense DNA origami solutions. Angew Chem Int Ed Engl. 53 (47), 12735-12740 (2014).
- Lin, C., Perrault, S. D., Kwak, M., Graf, F., Shih, W. M. Purification of DNA-origami nanostructures by rate-zonal centrifugation. Nucleic Acids Res. 41 (2), (2012).
- Bai, X. C., Martin, T. G., Scheres, S. H., Dietz, H. Cryo-EM structure of a 3D DNA-origami object. Proc Natl Acad Sci U S A. 109 (49), 20012-20017 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten