Method Article
Chromatin Immunopräzipitation (ChIP) mit Drosophila Gewebe
In diesem Artikel
Zusammenfassung
Kürzlich Hochdurchsatz-Sequenzierung Technologie hat stark Empfindlichkeit von Chromatin Immunopräzipitation (ChIP) Experiment erhöht und aufgefordert ihre Anwendung unter Verwendung von gereinigten Zellen oder Gewebe präpariert. Hier haben wir beschreiben eine Methode zur Chip-Technik mit nutzen Drosophila Gewebe, das den endogenen Chromatin Zustand in einem gut charakterisierten biologischen System adressieren kann.
Zusammenfassung
Epigenetik bleibt ein sich schnell entwickelndes Feld, das untersucht, wie das Chromatin Zustand zu differentiellen Genexpression in verschiedenen Zelltypen in verschiedenen Entwicklungsstadien beiträgt. Epigenetische Regulation trägt zu einem breiten Spektrum an biologischen Prozessen wie Zelldifferenzierung während der Embryonalentwicklung und Homöostase im Erwachsenenalter. Eine kritische Strategie in der epigenetischen Studien ist zu untersuchen, wie verschiedene Histon-Modifikationen und Chromatin Faktoren Genexpression zu regulieren. Um dieses Problem anzugehen, wird Chromatin Immunopräzipitation (ChIP) weit verbreitet, um einen Schnappschuss des Vereins von besonderer Faktoren, die mit DNA in den Zellen von Interesse zu erhalten verwendet.
Chip-Technik häufig verwendet kultivierten Zellen als Ausgangsmaterial, das in Hülle und Fülle und Homogenität erhalten werden können, um reproduzierbare Daten zu generieren. Es gibt jedoch mehrere Einschränkungen: Erstens ist die Umgebung, um Zellen in Petri-Schale wachsen anders als in vivo, kann somitspiegeln nicht die endogenen Chromatin Zustand der Zellen in einem lebenden Organismus. Zweitens kann nicht alle Arten von Zellen ex vivo kultiviert werden. Es gibt nur eine begrenzte Anzahl von Zelllinien, von denen die Menschen genügend Material für die Chip-Assay erhalten können.
Hier beschreiben wir eine Methode, um ChIP Experiment mit Drosophila Gewebe zu tun. Das Ausgangsmaterial wird Gewebe aus dem lebenden Tier seziert, so kann genau die endogenen Chromatin Zustand. Die Anpassungsfähigkeit dieser Methode mit vielen verschiedenen Arten von Gewebe ermöglicht es Forschern, viel mehr biologisch relevanten Fragen rund um epigenetische Regulation in vivo 1, 2 anzugehen. Die Kombination dieser Methode mit Hochdurchsatz-Sequenzierung (ChIP-seq) wird weiter Forschern erlauben, eine epigenomischen Landschaft zu erhalten.
Protokoll
(Der gesamte Chip Prozedur dauert etwa zwei Tage. Vorbereitung der ChIP-Bibliotheken für Hochdurchsatz-Sequenzierung dauert weitere 2-3 Tage.)
1. Bereiten Sie sezieren und Tissue für Chip-Technologie (~ 1 Mio. Zellen)
- Sezieren Gewebe von Interesse (zB 200 Paare von Drosophila Hoden) in kaltem PBS + 1x Protease-Inhibitor (auflösen 1 Kugel von Protease-Inhibitor-Cocktail in 1,5 ml 1x PBS zu 7x Stammlösung zu erhalten) + PMSF (Endkonzentration 100 pg / mL). Das Gewebe spülen zweimal und resuspendieren Gewebe in 200 ul der gleichen PBS-Lösung mit Inhibitoren.
- Um das Beispiel zu beheben, fügen 5,5 ul 37% Formaldehyd (Formaldehyd in warmen 37 ° C Wasserbad für 1 min vor der Verwendung). Bei 37 ° C für 15 Minuten, alle 5 Minuten Wirbel dazwischen.
- Legen Sie die Probe auf Eis für das Gewebe, um für 2 Minuten absitzen. Spülen Sie 2x mit 450 ul PBS (mit Protease-Inhibitoren und PMSF). Probe kann nun sein Geschäftd bei -20 ° C Wiederholen Sie Dissektion mehrmals um genügend Material für ChIP zu erhalten.
2. Bereiten Überstand mit Protein-DNA-Konjugation für Chip-Assay
- Kombinieren Sie genug Probe aus Schritt 1.3 in einer 1,75 ml Eppendorf-Röhrchen.
- Entfernen von PBS Gewebeprobe. Zugabe von 200 ul Lysepuffer (50 mM Tris-HCl, pH 7,6, 1 mM CaCl 2, 0,2% Triton X-100 oder NP-40, 5 mM Butyrat und 1x Proteinase-Inhibitor-Cocktail). In frischen PMSF Stammlösung zu Lysepuffer (PMSF Endkonzentration von 0,5 mM). Homogenisieren mit blauen Homogenisator (Fisher Cat # 749521-1590) bis zum Gewebe vollständig distanzieren, ohne Aggregation, bei RT (Raumtemperatur) für 10 Minuten inkubieren.
- Beschallung: Verwendung Mikrospitze Sonicator (Misonix HS-XL200) in Kraft 20. Mit Ultraschall für 10 Sekunden und liegen auf Eis für 50 Sekunden. Wiederholen Sie 4-5 mal (optimale Beschallung Zeit sollte empirisch ermittelt werden, da längere Beschallung kleinere Fragmente erhalten werden. Für CHIP Verwendung von Antikörpern gegen Histone Modifikationen, die wir normalerweise beschallen Chromatin auf ca. 200-300 bp; für Transkriptionsfaktor oder Chromatin-Regler (z. B. Polycomb Proteine), die wir normalerweise zu beschallen Chromatin 200-1000 bp. Allerdings sollte die optimale Fragmentgröße empirisch getestet werden. Hinweis: Halten Sie das Röhrchen auf Eis AUCH WÄHREND Beschallung für die Stichprobenziehung aus erwärmt wird!
- Verdünnen der Probe mit indem 1,8 ml RIPA-Puffer (10 mM Tris-HCl, pH 7,6, 1 mM EDTA, 0,1% SDS, 0,1% Na-Desoxycholat, 1% Triton X-100, mit Protease-Inhibitoren PMSF und bei der gleichen Konzentration wie beschrieben vorher). Sparen Sie 40 ul als Input-Steuerung durch Zugabe von 2 ul 5 M NaCl und Inkubation bei 65 ° C über Nacht (O / N) zur Vernetzung umzukehren.
- Um Antikörper zu konjugieren Perlen, dann werden 40 ul Protein A-Kügelchen in ein 1,5 ml Eppendorf-Röhrchen, fügen Sie dann 600 ul PBS und Rock bei 4 ° C für 2 Minuten, gelten Perlen Magnet und entfernen Überstand. Geben Sie 100 ul PBS und den Antikörper von Interesse (Menge des Antikörpers sollte bestimmt werden, empitisch). Inkubieren bei Raumtemperatur für 1 Stunde oder 4 ° C für 4 Stunden.
- Entfernen Sie aus Perlen von Magneten und Überstand 1ml Chromatin Auszug aus Schritt 2.4 an den Beads. Drehen bei 4 ° CO / N
- Bewerben Chipprobe zu Magnet und entfernen Überstand. Waschen der Kügelchen mit der folgenden Puffer bei 4 ° C für 10 Minuten:
2x mit 1 ml RIPA-Puffer [1,89 ml 'RIPA Puffer' + 315 ul 7x Proteaseinhibitoren PMSF + 20 ul];
2x mit 1 ml RIPA-Puffer + 0,3 M NaCl [1,89 ml RIPA-Puffer + 220 ul 3 M NaCl];
2x mit 1 ml LiCl-Puffer (0,25 M LiCl, 0,5% NP40, 0,5% NaDOC);
1x mit 1 ml 1x TE + 0,2% Triton X-100;
1x mit 1 ml 1x TE. - Zu vernetzen rückgängig zu machen, die Perlen zu resuspendieren in 100 ul TE-Puffer + 3 ul 10% SDS + 5 ul Proteinase K (20 mg / mL). Inkubation bei 65 ° CO / N.
- Bewerben Perlen an Magneten und den Überstand in ein neues Röhrchen. Der Überstand enthält Ihre DNA-Probe. Waschen Sie Perlen mit 100 ul TE + 0,5 MNaCl und kombinieren die beiden Überstand.
- Phenol-Chloroform extrahieren die DNA-Probe. Fügen Sie 200 ul Phenol: Chlorofrom: IAA (25:24:1)-Mix und Wirbel. Zentrifugieren bei 14k für 5 Minuten bei RT. Alternativ können DNA extrahiert unter Verwendung von Qiagen PCR Purification Kit werden.
- Die überstehende / wässrige Schicht in ein neues Röhrchen, und geben linearen Acrylamid bis zu einer Endkonzentration von 20 ug / ml (Alternativ 1 ul Glykogen bei 20 mg / ml pro 1 ml des Überstandes können ebenfalls verwendet werden. Glycogen für verwendet wird anschließende qPCR oder PCR-Analysen während lineare Acrylamid-Assays für die Sequenzierung verwendet wird.) Geben Sie 20 ul 3 M NaOAc und 500 ul 100% EtOH. Gut mischen und inkubieren bei 80 ° C für 10 Minuten. Spin bei maximaler Geschwindigkeit für 20 Minuten bei 4 ° C
- Überstand abnehmen und waschen mit 300 ul 70% EtOH. Der Luft trocknen und resuspendieren Probe in 50 ul TE-Puffer. Probe kann nun bei -20 ° C gelagert werden Probe für die quantitative PCR (qPCR)-Tests (1) verwendet werden.
3. Analysieren ChIP-ED-DNA
3a. Analysieren ChIP-ED-DNA mittels qPCR
- Verdünnen Sie genomischer DNA unter der folgenden Konzentration, um eine Standardkurve zu machen: unverdünnt, 1/10, 1/100, 1/1000, 1/5000.
- Für jedes Primerpaar einrichten qPCR in zweifacher Ausfertigung in einer 96-Well-Platte mit genomischer DNA aus Schritt 3a.1, Input-Steuerung und ChIP-ED-DNA:
10 ul 2x SYBR PCR-Mix (Fermentas, K0222);
1 ul 10 uM Vorwärts-Primer;
1 ul 10 uM Reverse-Primer;
x ul ChIP-ED-DNA (Input-Steuerung, oder genomische DNA);
y ul Nuclease-freiem H 2 O um das Reaktionsvolumen auf 20 ul einzustellen. - Drehen Sie den 96-Well Platte mit Mini-Plate Spinner (Labnet).
- Führen Sie mit qPCR ABI 7300 real time PCR-System (oder andere Echtzeit-PCR-Systeme) mit der folgenden Bedingung:
Stufe 1: 50 ° C für 2 min, 1 Zyklus;
Stufe 2: 95 ° C für 10 min, 1 Zyklus;
Stufe 3: 95 ° C für 15 s, 60 ° C für 1 min, 40 cycles;
Stufe 4 (Dissoziationsstufe): 95 ° C für 15 s, 60 ° C für 1 min, 95 ° C für 15 s. - Überprüfen Sie die Dissoziationskurve: Ein Peak bei der thermischen Spaltung Grundstück deutet auf eine einzelne Fragment aus der PCR-Reaktion. Mehr als ein Peak auf nicht-spezifische Produkt mit dem Primerpaar, sollten die qPCR-Daten nicht verwendet werden.
- Bestimmen der linearen Phase des exponentiellen Amplifikation der PCR-Reaktion für jeden Primer, durch Berechnung der Formel, die DNA-Menge löst gemäß der Ct-Wert (Schwellenwert-Zyklus)-Wert, der auf der Standardkurve unter Verwendung der Reihe von verdünnten genomischer DNA in 3a basiert. 1.
- Wenn der Ct-Wert von CHIP-ED-DNA und Input-Steuerung im linearen Bereich der Ct-Wert sind, berechnen die Anreicherung von ChIP-ED-DNA-Technologie bzw.-Eingang, nach dem Verdünnen Faktor von CHIP-ED-DNA und Eingabekontrolle in 2.4.
3b. Amplify ChIP-ED-DNA für die Hochdurchsatz-Sequenzierung.
- End-Reparatur von Chip-ED-DNA-Mix (Verwenden SieEpicentre DNA-END-Reparaturset), bis auf Eis gesetzt:
1-34 ul genomische DNA aus Schritt 2.12 (0,1 bis 5 ug)
5 ul 10x Puffer End Reparatur
5 ul 2,5 mM dNTP-Mix
5 ul 10 mM ATP
x ul H 2 O um das Reaktionsvolumen auf 49 ul anzupassen
1 ul End-Reparatur-Enzym-Mix (T4 DNA-Polymerase + T4 Polynukleotid-Kinase)
Inkubieren für 45 Minuten bei RT. Entschlacken Reaktion unter Verwendung von Qiagen MinElute Reaction
Cleanup Kit. Man eluiert in 30 ul Elutionspuffer (EB). - In A-Überhänge an das 3'-Ende:
30 ul eluierte DNA aus Schritt 3b.1
5 ul 10x NEB Buffer # 2
10 ul 1 mM dATP
2 ul dH 2 O
3 ul 5 U / ul Klenow-Fragment (3 '→ 5' Exo-)
Inkubieren für 30 Minuten bei 37 ° C Entschlacken Reaktion unter Verwendung von MinElute ReactionCleanup Kit. Man eluiert in 10 ul EB. - Solexa Linkerligation:
10 ul eluierte DNA aus Schritt 3b.2
10 pl ddH 2 O
2.5 μ l 10x T4 DNA-Ligase-Puffer
1 ul Adapter Oligo-Mix von Illumina (1/10-diluted ab Lager lieferbar)
2,5 ul T4 DNA-Ligase (400 Einheiten / ul)
Inkubation für 1 Stunde bei RT. Entschlacken Reaktion unter Verwendung von MinElute ReactionCleanup Kit. Man eluiert in 20 ul EB. Probe kann nun bei -20 ° C gelagert werden - Führen Sie den eluierten DNA-Probe von Schritt 3b.3 mit dem E-Gel-Apparatur. Isolieren von 300 bis 500 bp-Bande im Gel und Reinigung unter Verwendung des Qiagen Gel Extraction Kit (dieser Schritt entfällt die ~ 125 bp selbst ligierten Linker aus dem Adapter Oligo-Ligation). Man eluiert in 12 ul EB.
- Amplify Bibliothek unter Verwendung von gepaarten-End-(PE)-Primer von Illumina:
10,5 ul der eluierten DNA aus Schritt 3b.4
12,5 ul Mastermix (2X Phusion HF, Finnzymes)
1 ul PE1 PCR-Primer 1
1 ul PE2 PCR-Primer 2
PCR-Bedingungen:
98 ° C für 30 Sek.
(98 ° C 10 sec, 65 ° C 30 sec, 72 ° C 30 sec), 20 Zyklen wiederholen
72 ° C für 5 Minuten. - Proben aus Schritt 3b.5 für ChIP-seq nach der Auswahl der richtigen Größe DNA (300-500 bp) unter Verwendung des 2% igen Agarosegel verwendet werden. Probe kann nun bei -20 ° C gelagert werden Am besten ist es jeden Chip Probe auf einem einzigen Gel zur Vermeidung von Kontaminationen zu isolieren. Die Proben können für Hochdurchsatz-Sequenzierung eingereicht werden.
3c. Solexa Pipeline-Analyse
- Der 25-bp-Sequenzierung liest wurden aus dem Illumina Genome Analyzer (GA)-Pipeline erhalten.
- Alle gelesen wurden zu dem Drosophila-Genoms (dm3) unter Verwendung des ELAND (Efficient Local Alignment der Nukleotid-Daten) Software ausgerichtet sind, so dass bis zu zwei Fehlpaarungen mit der Referenzsequenz.
- Einzigartig abgebildet mal gelesen wurden für Downstream-Analyse erhalten. Für mehrere identische mal gelesen wurden bis zu drei Exemplare erhalten, um die Möglichkeit von Verzerrungen von PCR-Amplifikation zu reduzieren.
- Der Ausgang des GA-Pipeline wurde in Browser erweiterbare Daten (BED) konvertiert.
- So generierendie Perücke Dateien zum Upload auf die UCSC Browser zur Visualisierung verwendeten wir eine Python-Tasche, die in einer früheren Veröffentlichung 3 wurde mit 4 bp als der Bildschirmgröße und der 160 bp DNA-Fragment wie die Größe beschrieben.
- Um Drosophila-Gene in stille und äußerte Gene klassifizieren, verwendeten wir eine digitale Zahl genannt RPKM (liest / pro Kilobasen zusammengeführt exonische Region / pro Million mapped liest). Gene mit RPKM = 0 wurden als stille Gruppe und Gene mit RPKM ≥ 1 wurden in die Gruppen zum Ausdruck, die in drei Untergruppen, wie Gene, die mit niedrigen, mittleren und hohen Expressionsniveaus klassifiziert werden können eingestuft, und jede Gruppe hat etwa die gleiche Anzahl von Gene.
- Um die Histonmodifikationen Ebene und Genexpression zu vergleichen, haben wir ein Python-Skript, das in einer früheren Veröffentlichung 3 beschrieben wurde.
4. Repräsentative Ergebnisse
Beispiele für ChIP-qPCR-Ergebnisse mit BAM (Beutel Murmeln)-Mutante Hoden sind in Abbildung 1 4 gezeigt. In Bam Hoden, es zu einem Ausfall beim Übergang von der proliferativen Spermatogonien zu differenzieren Spermatozyten 5, 6. Wir verwenden bam Hoden als Quelle für undifferenzierte Keimzellen, die in diesem Gewebetyp angereichert sind. Differenzierung Gene für Spermien Differenzierung erforderlich, wie männlich-spezifischen Transkriptionsfaktors 87 (mst87F), Don Juan (dj) und Fuzzy-Zwiebel (FZO) sind nicht in bam Hoden exprimiert. Diese Gene sind mit der repressiven H3K27me3 Histonmodifikationen 7 (Abbildung 1A) angereichert, sind aber ohne die aktive H3K4me3 Histonmodifikationen 8 (Abbildung 1B), ein Chromatin-Signatur wir als "monovalenten" 7. Die Anreicherung von entweder H3K27me3 oder H3K4me3 wird durch Normalisierung zu einem konstitutiv exprimierten Cyclin A (CycA)-Gen 4 bestimmt.
Inhalt "> Chip-seq-Analyse unter Verwendung der gleichen Menge von Antikörpern (dh repressive H3K27me3 und aktive H3K4me3) mit BAM-Mutante Hoden (Abbildung 2) die qPCR Ergebnisse in Abbildung 1 dargestellt validiert. Für die drei untersuchten Gene mst87F terminalen Differenzierung, DJ und FZO , ihre genomische Regionen stark mit H3K27me3 aber nicht H3K4me3 (2A-2C) angereichert. Im Gegensatz dazu weist das konstitutiv exprimierte Gen signifikante CycA H3K4me3 wenig H3K27me3 Bindung nahe der Transkriptionsstartstelle (TSS) (2D).Darüber hinaus sind die Profile der ChIP H3K27me3 und H3K4me3 in der Nähe der TSS von vier Klassen von differentiell exprimierten Genen, die der Funktion der einzelnen Histonmodifikationen. Wie in 3A gezeigt ist, ist die Anreicherung von H3K27me3 stromabwärts des TSS invers mit Genexpression korreliert. Die stillen Gene haben die höchste H3K27me3 während diehoch exprimierte Gene haben keine H3K27me3 bindend. Diese Daten sind konsistent mit der repressiven Rolle der H3K27me3 auf die Genexpression. Im Gegensatz dazu zeigten die Anreicherung von H3K4me3 um die TSS den umgekehrten Korrelation mit Genexpressions-Ebene (3B), im Einklang mit der aktiven Rolle der H3K4me3 auf die Genexpression.
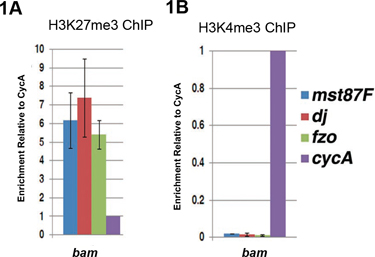
Abbildung 1. QPCR Analyse der ChIP-ED-DNA unter Verwendung von entweder Antikörper gegen das repressive H3K27me3 Histonmodifikationen (A) oder aktiv H3K4me3 Histon-Modifikation (B) in undifferenzierten Zellen angereicherte bam mutierten Hoden. (A) in der BAM Hoden, Differenzierung Gene (Mst87F, dj, FZO) werden mit dem H3K27me3 repressive Histon-Modifikation bereichert. (B) Differentiation Gene sind mit H3K4me3 aktive Marke erschöpft. Das Niveau der Chip-DNA (ChIP DNA / Eingang) auf das Ziel-Gen (Mst87F, DJ oder FZO) wurde zum ersten Mal an den lev normalisiert el der Chip-DNA an der Steuerung CycA Gen. Fehlerbalken zeigen die Standardabweichung aus drei unabhängigen biologischen Replikate.

Abbildung 2. UCSC Genom-Browser Momentaufnahmen H3K27me3 und H3K4me3 Anreicherung über die gesamte genomische Regionen (A) Mst87F (B) dj und (C) FZO, (D) CycA Gene. Die eingekreisten H3K27me3 angereicherte Region (D) spiegelt die Chromatin Status ein überlappender Gen CG7264, die in der BAM niedrig exprimiert wird Hoden (RPKM = 1), aber hoch in Wildtyp-Hoden exprimiert (RPKM = 130) 8 (ChIP-seq Daten aus 7). Sonden in der quantitativen PCR-Analyse von ChIP Ergebnisse in Abbildung 1 verwendet werden, am unteren Rand jeder Parzelle beschriftet. Klicken Sie hier für eine größere Abbildung anzuzeigen .
tp_upload/3745/3745fig3.jpg "/>
Abbildung 3. ChIP-seq Profile mit H3K27me3 und H3K4me3 in bam Hoden 7. Die vier Gruppen Gens (Gene 7509) wurden entsprechend ihrer Genexpression auf RNA-seq Ergebnisse 8 klassifiziert. Die repräsentativen Klassen von Genen sind für die Anreicherung eines bestimmten Histon-Modifikation aufgetragen, mit Sequenzen aus 3kb stromaufwärts bis 3kb hinter ihren Transkriptionsstartstellen (TSS). Dies erzeugt ein Profil der Anreicherung von (A) H3K27me3 (K27) und (B) H3K4me3 (K4) ChIP-seq Analysen in jeder Gruppe. Klicken Sie hier für eine größere Abbildung anzuzeigen .
Diskussion
Die Vielseitigkeit der Chip-Analysen in diesem Protokoll behandelt werden, können auf verschiedenen Geweben, die eine Gelegenheit, die Chromatin-Zustand in einer biologisch relevanten System zu studieren bietet eingesetzt werden. Chip-Experimenten unter Verwendung von Zellen aus kultivierten Systeme sind bequem durchgeführt werden, da große Menge von Zellen leicht erhalten werden kann. Jedoch nicht kultivierten Zellen nicht notwendigerweise Zellen in einem mehrzelligen Umgebung. Durch die Entwicklung dieser Technik mit sezierten Gewebe von lebenden Tieren, können wir auf viele Fragen, die kultivierten Zellen kann es nicht.
Trotz der Nützlichkeit dieses Protokoll, gibt es einige Fallstricke. Zum Beispiel ist die seziert Gewebe noch heterogenen mit verschiedenen Zelltypen. Während die relativ homogene Zellen können in Drosophila Gewebe wie Imaginalscheiben, andere Gewebe wie Darm, Hoden, Eierstöcke und die alle enthalten begrenzte adulten Stammzellen sind heterogen bezogen werden. Diese Komplikation kann teilweiseadressiert mit leistungsfähigen Drosophila-Genetik. Obwohl zum Beispiel adulte Stammzellen in einer kleinen Population im Gewebe vorhanden sind und oben besprochen sind extrem schwierig, in einer ausreichenden Anzahl für Chip-Analyse isoliert werden, können Stammzellen Bevölkerung bereichert die Verwendung von genetisch mutierten Hintergrund werden. Die BAM-Gen ist notwendig für Stammzell-Differenzierung in Drosophila Keimbahn-Linie 5,6. Deshalb bam mutierten Gonaden sind eine gute Quelle für angereichertes undifferenzierten Keimzellen. Mit der Durchführung Chip mit BAM-Mutante, erhält man eine epigenomischen Landschaft in undifferenzierten Keimzellen.
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Die Autoren danken Herrn Dr. Keji Zhaos Labor (NIH / NHLBI) für ihre Hilfe bei der Bereitstellung von Sequenzierung Ergebnisse danken. Wir möchten auch, um die UCSC Genom-Projekt für die Nutzung der Genom-Browser zu visualisieren abgebildet Sequenzierung liest danken.
Diese Arbeit wurde durch die NIH R00HD055052 Pathway worden zu Independence Award und R01HD065816 aus NICHD, der Lucile Parkard-Stiftung, und der Johns Hopkins University Anschubfinanzierung zu XC unterstützt
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| Kompletter Mini Protease Inhibitor Cocktail | Roche | 11836153001 | |
| Formaldehyd (37%) | Supelco | 47.083 U- | |
| PMSF | Sigma | 78830 | |
| Kontes Pellet Pistill | Fischer Scientific | K749521-1590 | |
| PCR Purification Kit | Qiagen | 28104 | |
| Linearem Polyacrylamid | Sigma | 56575-1ML | |
| Glykogen | Qiagen | 158930 | |
| SYBR Green / ROX qPCR Master Mix | Fermentas | K0223 | |
| Mini Plate Spinner | Labnet | Z723533 | |
| Real-time PCR-System | Applied Biosystem | 4351101 | |
| Small Volume Ultraschall-Prozessor | Misonix | HS-XL2000 | Modell eingestellt |
| Dynabeads, Protein A | Invitrogen | 100-01D | |
| Dynamag Magnet | Invitrogen | 123-21D | |
| Phenol: Chlorofrom: IAA | Invitrogen | 15593-049 | |
| Epicentre DNA-END-Reparatur-Kit | Epicentre Biotechnologies | ER0720 | |
| MinElute Reaction Reinigungs Kit | Qiagen | 28204 | |
| Klenow-Fragment (3 '→ 5' Exo-) | New England Biolabs | M0212S | |
| T4 DNA-Ligase | Promega Corporation | M1794 | |
| Adaptor-Oligonukleotiden | Illumina | PE-400-1001 | |
| Gepaart-Ende-Primer 1,0 und 2,0 | Illumina | 1001783 1001 784 | |
| E-Gel Elektrophorese-System | Invitrogen | G6512ST | |
| 2X Phusion HF Mastermix | Finnzymes | F-531 |
Referenzen
- Kharchenko, P. V., Alekseyenko, A. A., Schwartz, Y. B., Minoda, A., Riddle, N. C., Ernst, J., Sabo, P. J., Larschan, E., Gorchakov, A. A., Gu, T. Comprehensive analysis of the chromatin landscape in Drosophila melanogaster. Nature. 471, 480-485 (2011).
- Filion, G. J., van Bemmel, J. G., Braunschweig, U., Talhout, W., Kind, J., Ward, L. D., Brugman, W., de Castro, I. J., Kerkhoven, R. M., Bussemaker, H. J., van Steensel, B. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Barski, A., Cuddapah, S., Cui, K., Roh, T. Y., Schones, D. E., Wang, Z., Wei, G., Chepelev, I., Zhao, K. High-resolution profiling of histone methylations in the human genome. Cell. 129, 823-837 (2007).
- Chen, X., Lu, C., Prado, J. R., Eun, S. H., Fuller, M. T. Sequential changes at differentiation gene promoters as they become active in a stem cell lineage. Development. 138, 2441-2450 (2011).
- Gonczy, P., Matunis, E., DiNardo, S. bag-of-marbles and benign gonial cell neoplasm act in the germline to restrict proliferation during Drosophila spermatogenesis. Development. 124, 4361-4371 (1997).
- McKearin, D. M., Spradling, A. C. bag-of-marbles: a Drosophila gene required to initiate both male and female gametogenesis. Genes Dev. 4, 2242-2251 (1990).
- Gan, Q., Schones, D. E., Eun, S. H., Wei, G., Cui, K., Zhao, K., Chen, X. Monovalent and unpoised status of most genes in undifferentiated cell-enriched Drosophila testis. Genome Biol. 11, 42-42 (2010).
- Gan, Q., Chepelev, I., Wei, G., Tarayrah, L., Cui, K., Zhao, K., Chen, X. Dynamic regulation of alternative splicing and chromatin structure in Drosophila gonads revealed by RNA-seq. Cell Res. 7, 763-783 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten