Method Article
非核苷酸逆转录酶抑制剂的定量构效关系、活性预测和分子动力学
摘要
本研究使用 计算机模拟 策略来确定列举的 Etravirine 是一种有前途的 HIV 治疗剂。我们对分子相互作用和动力学的研究结果支持合理设计新型 NNRTI 作为可能的 HIV 治疗选择。
摘要
HIV-1 耐药性发病率的增加对联合抗逆转录病毒治疗的有效性构成了挑战,尤其是在南部非洲。对非核苷酸逆转录酶抑制剂 (NNRTI) 的耐药性的发展威胁到抗逆转录病毒治疗的长期成功。2019 年,抗微生物药物耐药性直接导致全球约 127 万人死亡。本研究采用 计算机模拟 方法来研究 NNTRI 药物及其衍生物。使用的技术包括密度泛函理论计算、分子对接、计数、定量构效关系 (QSAR) 分析、分子动力学模拟 (MDS) 以及具有广义 Born 和表面积方法的分子力学。分析侧重于各种嘧啶衍生物和六种 NNRTI 药物,检查它们与 HIV-1 蛋白(PDB 代码 1HQU)的相互作用。
开发了一个 QSAR 模型来预测所研究的 6 种 NNRTIs 的生物活性。使用 94 种嘧啶衍生物,QSAR 模型实现了 R2 为 0.822,Q2 为 0.815,表明预测准确性很高。
进行 MDS 以评估各种配体及其新开发的替代品的稳定性,确保它们在 200 纳秒的模拟时间范围内保持与蛋白质活性位点的结合。依曲韦林的均方根偏差 (RMSD) 波动约为 4.5 Å,而其列举的衍生物的 RMSD 波动为 3.5 Å。通过分子对接、MDS 和自由能计算,列举的 Etravirine 表现出最佳性能,活性值为 7.373,对接评分为 -10.517 kcal/mol。此外,计算出的 Etravirine 的结合自由能为 -89.684 kcal/mol,优于正在研究的其他配体。显着的改进表明,改性的 Etravirine 作为抗逆转录病毒治疗的新型药物具有广阔的潜力。
获得的较低 RMSD 值、增强的氨基酸相互作用和最高的结合自由能表明,列举的 Etravirine 可以作为 HIV/AIDS 治疗的可行替代方案。
引言
尽管在治疗方面取得了长足的进步,但由人类免疫缺陷病毒 1 (HIV-1) 引起的获得性免疫缺陷综合症 (AIDS) 对全球健康的严重威胁仍然是一个持续的威胁1。被称为逆转录酶抑制剂 (RTI) 的抗逆转录病毒药物用于治疗 HIV 感染。逆转录酶是一种逆转录病毒复制所必需的病毒脱氧核糖核酸 (DNA) 聚合酶,可被逆转录酶抑制剂 (RTI) 抑制。核苷类逆转录酶抑制剂 (NRTIs) 和非核苷酸逆转录酶抑制剂 (NNRTIs) 是主要的RTIs2。
高效抗逆转录病毒疗法 (HAART) 是各种抗病毒药物的组合,已成为 HIV 的标准疗法,可有效控制艾滋病的传播,并将这种曾经致命的疾病转变为可控制的慢性病3。HIV-1 的 NNRTI 目前是 HAART 方案的重要组成部分4。抗微生物药物耐药性 (AMR) 是最严重的全球健康威胁之一,估计有 127 万人死亡5。因此,迫切需要对 AMR 进行故障排除并确定新型抗菌药物。根据世界卫生组织 (WHO) 的数据,世界许多地方的 AMR 已达到令人震惊的水平。
这对实现可持续发展目标构成了严重风险,因为它破坏了粮食安全、经济增长和健康安全,同时还加剧了社会和经济不平等6。耐药病毒的流行率正在上升,即使在旨在对抗 HIV 的抗逆转录病毒药物中也是如此。根据最近的统计数据,截至 2022 年底,全球有近 3000 万人正在接受抗逆转录病毒治疗7。
WHO 进行了 30 项调查,发现在其 21 项调查中,超过 10% 的开始一线抗逆转录病毒治疗的个体对奈韦拉平或依非韦伦8 产生耐药性。此外,既往接触过抗逆转录病毒药物的个体对 NNRTI 产生耐药性的可能性是未接触过的个体的三倍9。研究表明,相当多的 18 个月以下新诊断患有 HIV 的婴儿表现出很高的耐药菌株率10,11。令人震惊的是,他们中几乎有一半甚至在开始治疗之前就患有 - (NNRTI) 耐药菌株。这些发现强调了加快研究以设计创新 HIV 疗法的必要性。
耐药菌株的发展和长期使用的不良副作用不可避免地对 NNRTI 的临床使用构成挑战12。尽管可能存在不良副作用,但开始联合抗逆转录病毒疗法 (cART) 可延长 HIV 感染者的预期寿命。这些副作用包括患非传染性疾病的风险,包括脂肪代谢障碍、高脂血症、骨密度降低、血糖水平升高导致 2 型糖尿病、高血压、中风风险增加以及与肥胖相关的问题13。从 HIV 感染的初始阶段早期诊断和及时获得适当的医疗服务,从临床和公众的角度来看,都提供了相当大的优势。及时开始 ART 和预防机会性感染可显著降低 HIV 相关疾病和死亡率。
使用 ART 还可以通过降低循环 HIV 核糖核酸的水平来降低 HIV 进一步传播的可能性。此外,解决其他性传播疾病和合并感染的治疗同样可以降低进一步感染 HIV 的可能性14。NNRTIs 是抑制剂的可选治疗类别之一,通过与 HIV 的变构区域或部位结合与 RT 相互作用。这种类型的抑制通常被称为非竞争性抑制剂,因为 NNRTI 不在底物的活性位点结合,而是在外部结合。该效应改变了底物结合位点的构象,阻碍了标准底物的结合并导致链过早终止。由于 NNRTI 与 NRTI 相比毒性更低、结构简单、生物利用度优于蛋白酶抑制剂以及出色的选择性,因此已成为最具吸引力的 HIV 抑制剂15。因此,从药代动力学的角度来看,新型 NNRTI 的合成和设计至关重要16,17。
NNRTIs 因其疗效强、毒性低而在 HIV/AIDS 治疗中至关重要。然而,奈韦拉平、地拉韦定和依非韦伦等早期 NNRTI 在 NNRTI 结合位点遇到病毒突变的耐药性 18。这些药物是二芳基嘧啶 (DAPY) 家族的一部分,对各种 NNRTI 菌株表现出有效的活性,包括对早期 NNRTI 耐药的菌株19,这可能是由于它们的分子灵活性和对 HIV-1 的更高耐药屏障20,21。尽管他们取得了成功,但 HIV-1 RT 的高突变率和缺乏内在的校对活性导致使用 Etravirine 和 Rilpivirine 的患者出现新的耐药谱22,23。这些病毒突变彼此不同,与早期 NNRTI 药物相关的病毒突变也不同24。超过 50 类结构不同的化合物已被鉴定为 NNRTIs。值得注意的是,6 个 NNRTI 已获得治疗 HIV-1 的批准。这些获批的药物包括奈韦拉平 (NVP)、地拉韦定 (DLV)、依非韦伦 (EFV)、依曲韦林 (ETR)、利匹韦林 (RPV) 和多拉韦林 (DOR)。图 1 显示了这六种已获批的 NNRTI 药物的化学结构25。
在最近的一项研究中26,DFT 计算和分子对接表明,洛伐他汀和辛伐他汀显示出作为抗冠状病毒药物的潜力。虚拟筛选确定了 5 种经 FDA 批准的类似于依非韦伦支架的新型候选分子,在 COVID-19 主蛋白酶的活性口袋中具有优异的结合亲和力。
Soltan 等人27 进行了一项类似的研究,通过采用基于 FDA 批准的药物的基于片段的策略来设计化学衍生物,鉴定提高与 HIV RT 结合能力的新分子。他们专门利用地拉韦定、依非韦伦、依曲韦林和利匹韦林的结构作为基础支架。通过 Swiss-ADME 评估这些衍生物的药物相似性,然后将它们对接到相关的晶体结构中。该研究的结论是,与它们的母体支架相比,选择了表现出优异结合亲和力的化合物,特别注意到从第二代 NNRTIs、依曲韦林和利匹韦林设计的衍生物有更明显的改进。例如,衍生物 RPV01 和 RPV15 的对接能量值比利匹韦林显着增强,表明在靶向野生型和突变型 HIV RT 方面具有潜在效用。
Murugesan 及其同事28 进行的研究提出了各种药物化学方法,以提高 HIV 治疗的疗效并最大限度地减少耐药性。该研究采用了分子杂交、生物等位置换和高通量筛选。在他们的研究中,他们设法鉴定出对 HIV 野生型和耐药菌株均具有高效力的新型 NNRTI 支架。他们开发了表现出优异选择性和低毒性的 DAPY 衍生物,其中一些化合物在纳摩尔浓度下表现出有效的抑制作用。
最近,由于对药物量子化学特性的实用分析,将计算工具与 计算机研究 一起使用越来越受欢迎29。在本研究中,应用计算机辅助药物设计、密度泛函理论、定性构效关系和分子动力学来发现有效的 NNRTIs。
研究方案
1. 计算细节 - 蛋白质制备
- 单击显示器屏幕上的 Window 图标,然后单击 All Apps。向下滚动以导航到 Schrodinger 文件夹,打开该文件夹,然后单击图 2B 所示的 Maestro 图标,然后选择如图 2B 所示的打开以启动软件。
- 通过导航软件中的 File 选项卡按钮来检索所选的 Protein structure。从弹出的简短菜单中,选择 Get PDB(如图 3A 所示),然后在文本框中输入所选的 PDB 代码 ,如图 3B 所示。单击 下载 按钮,所选的 PDB 文件将显示在项目窗口中。
- 或者,通过在搜索框中输入蛋白质数据库身份码 (PDB ID),将所选蛋白质从蛋白质数据库下载到本地计算机中,然后单击 下载 将 PDB 文件下载到本地计算机中。导航到 File 选项卡,然后选择 Import Structures 选项。在 导入 界面中,找到下载的 PDB 文件,如图 3C 所示,然后选择 导入 按钮,如图 3D 所示。
注意:蛋白质结构将在单独的窗口中以 3D 结构的形式打开,如图 4 所示。 - 导航到软件的右上角,选择任务选项,然后在搜索栏中键入 protein preparation。单击右侧显示屏中的蛋白质制备工作流程,如图 5A 所示。
- 在弹出的 蛋白质制备工作流程 窗口中,将 Job name 写下要保存的 文件名 ,然后单击右下角的绿色 Run 按钮,如图 5B 所示。
- 通过单击右上角的 Jobs 按钮来监视作业的运行情况,如图 5C 所示。
- 选择并右键单击制备的蛋白质并选择 分裂配体,如图 5D 所示。选择分成配体、水等。这是为了将蛋白质成分作为工作区导航器中的独立条目
2. 配体制备
- 通过在搜索栏中输入所选化合物的名称,从 PubChem 数据库30 下载所需的化合物。浏览结构并选择 3 维 (3D) 结构。单击右上方窗口的 Download (下载) 以将结构坐标下载为结构化数据文件 (SDF)。如果 3D 结构不可用,请下载 2D 结构并使用其他工具从 2D 结构生成 3D 结构。
- 单击 Schrodinger 中的 File 选项卡,然后选择 Import Structures,如图 6A 所示。导航到以 SDF 文件格式保存结构的文件位置,以加载要制备的化合物。
- 选择 Schrodinger 软件右上角的 Task。在搜索栏中键入 LigPrep,然后在左侧的右侧窗口中选择 LigPrep,如图 6B 所示。
- 选择 Use Structures from 以从 Workspace 或 Project 表格中选择文件。从 LigPrep 窗口中选择首选选项,将文件保存在本地计算机中,然后单击 run(运行 )提交配体制备作业,如图 6C 所示。
3. 配体的几何和优化
- 打开软件31 对下载的结构进行几何优化。导航到 “文件 ”选项卡(图 7A)并选择 “打开 ”以从 PubChem 数据库中选择下载的 SDF 文件。
注意:文件将在主窗口中加载。点击另一个紫色窗口以可视化相同的化合物。每个已实施的设置都会在紫色窗口中显示结果。 - 导航到 Calculate 选项卡,然后选择 Gaussian Calculation Setup 选项卡。导航到图 7B 所示的 Job type 选项卡,然后选择 Optimization 或 Opt+Freq。
注意:根据原子或化合物的(大小)数量,先优化,然后频率。如果化合物较小,则执行 Opt+Freq。分子越大,与 Optimization 后跟 Frequency 相比,执行 Opt+Freq 所需的时间就越长。 - 导航到 Method ( 方法 ) 选项卡,选择 量子化学方法 ,然后从每个部分的下拉箭头中选择 Kohn-Sham 全局混合交换相关密度泛函、 基集、 电荷和 自旋 。
- 导航到 Title ( 标题 ) 并指定正在调查的化合物的名称。
- 导航到 Link 0 选项卡并指定 Memory limit 和 Shared processors 选择。取消勾选 Full path 框,如图 7C 所示。
- 单击底部的 Edit 按钮以保存 Gaussian 输入文件,如图 3C 所示。将文件保存在首选位置,并使用所选的文件名作为 Gaussian job file (gjf)。
注意:保存高斯输入文件后,将出现一个弹出窗口,在记事本中显示文件的内容。编辑文件的内容,包括设置选项中不可用的选项,例如更改费用和功能。准备好的高斯作业文件将用作输入文件,以便通过本地计算机提交以运行优化和频率计算。
随后,使用 MN15-L 泛函32 和 6-31++G(d,p) 基集33 对这些结构进行几何优化。但是,如果在优化和频率过程的运行中出现问题,则可以在 HPC 的帮助下提交工作。
4. 受体网格生成
- 导航到 Tasks 并选择 受体网格生成34。 图 8A 所示的受体网格生成界面确定了核心晶体配体结合的蛋白质活性位点。单击 Pick 以识别配体 ,并从 图 8B 所示的顶部弹出通知中检查是否存在共结晶配体。
- 从工作区中选择 配体 和/或 残基 ,选定的目标化合物将在工作区中以蓝色突出显示。
- 选择接收器网格生成面板中的设置选项卡以设置 网格框大小。网格框的默认大小为 10 Å x 10Å x 10Å。通过直接输入所需值或在工作区中手动调整框的大小,在 Grid Box 选项卡中修改框尺寸。
注意:或者,如有必要,可以从 Advanced 选项卡设置其他参数,如图 8C 所示。检查所有设置以确保准确性。 - 单击 Run 开始生成网格。完成后,保存生成的栅格文件以供后续的停靠模拟使用。当作业完成时,将显示一条通知消息,如图 8D 所示。
5. 分子对接
- 通过导航到 Tasks、Docking35 和 Ligand Docking(滑行对接)加载蛋白质和准备好的配体,如图 9A 所示。
- 加载上述步骤 4.1 中的网格文件,并使用 图 9B 中的选项中的使用配体从工作区中选择配体。
- 从图 9 C 所示的设置选项卡中选择首选的停靠精度方法(默认停靠精度为标准精度 (SP))。
- 将 力场 设置为 OPLS4 以准确模拟分子相互作用。在 Constraints 选项卡中设置约束(例如,氢键)。
- 查看所有设置并保存停靠作业或文件。单击 RUN 开始停靠过程。
- 此时将显示一条通知,指示作业已完成,并且将打开 Project Table (项目表) 以获取停靠结果。检查 绑定姿势、分数和 交互。
6. 2D-QSAR 模型生成
- 转到 KNIME 社区中心网页,然后在搜索栏中搜索 AutoQsar。选择第二个 AutoQsar 条目,如图 10A 所示。
- 单击 download 工作流图标,并在图 10B 所示的弹出菜单中选择 download workflow。将工作流保存到本地计算机。
- 确保已安装 KNIME36 ,然后从本地计算机打开它。编制一份电子表格 ,其中包含规范的微笑、结构名称、活性/IC50 值 (以微摩尔为单位)和 -log(活性/IC)或任何 选择的化学描述符。将文件以逗号分隔值 (CSV) 格式保存在本地计算机上。
- 导航到 文件 并导入 AutoQSAR 工作流程37。从弹出窗口中,单击 Import KNIME Workflow 选择下载的 AutoQSAR Workflow 工作流,如图 10C 所示,然后选择 Open |下一页 |完成。
- AutoQsar 工作流将显示在左上角的 KNIME Explorer 窗口中。双击工作流,将工作流引入主窗口。
- 双击 分子阅读器(到 MAE) 图标,然后从 “设置 ”选项卡中,单击“ 添加文件” 以添加包含数据输入数据或参数的工作表。
- 选择 Memory Policy 选项卡,然后单击 Write Tables to disc |好的。
- 右键单击 Extract Properties 按钮并选择 Configure,如图 10D 所示。
- 从 排除 窗口选择并过滤所选的分子或化学性质到 包含 窗口。单击 Apply。
- 右键单击 AutoQSAR Build Model ,然后单击 Configure。在弹出的对话框中输入 QSAR 型号名称 。
- 单击选择按钮 ,然后从包含的属性中选择要适合的属性。选择 随机训练集值 (例如,85%:15%)和要保留的几个模型。单击 Apply。
- 右键单击底部的 Molecule Reader 并选择 Configure。从准备好的 CSV Excel 文件中加载测试配体,该文件包含要测试的配体。
- 右键单击 Extract Properties 并将设置设置为您的选择。
- 右键单击顶部的 Molecule Reader 并选择 execute。
注意:橙色点表示流程已开始,绿灯表示流程执行成功。下一步将继续进行,直到成功执行所有节点。 - .执行第二个 Molecule Reader for Test 配体。
- 在成功执行所有节点后,将包含测试和训练集结果的 zip 文件夹保存。
注意:根据交叉验证结果选择性能最佳的模型,例如 SD – 标准差、R2 – 实际和预测活动值之间的训练集相关性以及 Q2 – 测试集实际和预测活动相关性。 - 通过使用评分器或统计节点评估模型的性能来评估工作流性能。
- 要解释结果,请使用 feature importance 标识 main 或 key 描述符。将结果可视化为散点图或条形图,以确定性能和描述符的关系。保存工作流并将结果数据、预测和可视化导出为散点图和/或 CSV 文件。
7. 列举
- 导航到 Task 并搜索 Ligand Designer,如图 11A 所示。
- 从工作区导航器中选择一对 停靠的蛋白质 和 配体 复合物。单击 Ligand designer 窗口中的 Analyze Workspace。要生成和评估新的配体,请从出现的工作流程列表中选择 Isostere scanning ,如图 11B 所示,这意味着通过将片段添加到分子的现有部分来扩展配体的 生长方法 。
- 设置 enumeration 选项后,从出现的 Isostere Scanning 通知窗口中单击 enumerate 。对相同的蛋白质和不同的配体重复步骤 6.2,直到所有配体都运行相同的过程。查看 Projects 表中列举的配体。
- 通过导出设计的配体来保存结构。
注意:枚举方法在模拟完成后生成一组对接分数。 - 单独选择具有更负对接分数的列举化合物,并使用第 4 节中概述的对接程序重新对接它们。
8. HOMO 和 LUMO
- 打开软件并以高斯作业文件格式上传优化的配体。
- 导航到 工具 并选择 红点和绿点中的 MO 编辑器,如图 12A 所示。
注意:枚举方法在模拟完成后生成一组对接分数。 - 在 method 下的 MO 编辑器面板中,通过单击从现有 Chk 或 FChk 文件加载 Mos 来加载 FChk 文件,如图 12B 所示。
- 单击 Visualize 选项卡 | Update。等待 ~10 秒,让当前表面出现,如图 12D 所示。
- 单击突出显示的黄色数字旁边的两个 复选框 之一,以选择要在旁边的窗口中显示的 HOMO 或 LUMO38 表面。
- 右键单击紫色背景,然后选择 File (文件)。单击 save the image file 以保存当前表面的图像,如图 12E 所示。
- 右键单击紫色背景,然后选择 View (查看)。选择显示格式以更改 General 选项卡中的背景、Surface 选项卡中的表面透明度、Text 选项卡中的字体大小和颜色、Molecule 选项卡下的图像质量和表面的首选布局,如图 12F 所示。
- 或者,生成一个 cube 文件,并将 FChk 文件上传到 GaussView 中。导航到 Results 选项卡,然后选择 Surfaces/Contours。单击 cube actions 并加载 cube。单击 Surface Actions 并选择新表面。重复步骤 8.7 以编辑曲面。
注意:HOMO 和 LUMO39 之间的能隙越小(LUMO 和 HOMO 之间的差异),分子的预期反应性就越强。使用方程 1 为每个分子计算能量间隙 (Egap)。
9. 分子动力学模拟 - 系统准备和最小化
- 确保已在本地计算机中安装了 Schrodinger Suite,并在工作区40 中加载蛋白质-配体复合物结构。
- 单击 Task 按钮并选择 Desmond System Builder41。在系统组装商面板中,选择“溶剂化”选项卡,然后选择适合蛋白质-配体复合物的预定义溶剂模型(如图 13A 所示)、盒形(如图 13B)和盒大小计算方法(如图 13C42 所示)。
- 选择 ions(离子 )选项卡,然后单击 recalce(重新计算 ),通过添加反离子并设置 所需的盐浓度来中和系统。
- 选择所选的 OPLS 43,44 作为力场。
- 适当地命名作业,并将作业文件保存在本地计算机上。单击 run (运行 ) 提交作业进行准备。
注意:确保已保存作业名称。使用详细名称。 - 平衡和生产
- 在工作空间中查看项目 系统准备后。从 Workspace Navigator 中选择蛋白质-配体复合物,导航任务,然后选择分子动力学 (Desmond)。
- 从分子动力学面板的工作区加载配体-蛋白质复合物。从 simulation 选项卡中选择所需的模拟时间轴。选择 NPT 作为集成类45。
- 适当地命名作业并写出作业。单击 Close 退出分子动力学窗口。
- 通过本地终端提交写出的分子动力学准备作业。完成后,打开已完成的作业并继续模拟时间,从初始设置的模拟时间线开始,直到所需的模拟时间45,例如 100 ns、200 ns。
注:对其他蛋白质-配体复合物重复步骤 9。- 在 Schrodinger Maestro 软件中打开已完成的作业,如果运行了单独的作业,则合并不同的模拟时间帧。导航到 TASK 按钮并选择 模拟交互图。加载合并的文件或单个文件以可视化轨迹。
- 生成包含可视化效果和其他详细输出结果的报表。
10. 广义 Born 和表面积的分子力学 (MM-GBSA)
- 打开轨迹文件并播放轨迹。可视化蛋白质-配体复合物的平衡位置并记下帧数。通过终端提交作业进行处理。
- 对另一个蛋白质 - 配体复合物重复第 9 节(准备蛋白质分子动力学模拟)。
- 连接/查看输出文件内容以分析生成的结果。读取 ΔG 平均值以获得蛋白质-配体复合物的结合自由能。
- 下载 CSV 文件以可视化不同分子内分子的贡献。
- 要计算复合物的自由结合能,请注意各种热力学和脱溶剂化参数,包括结合能(ΔG结合)、哥伦布溶剂化模型(ΔG结合 Coulumb)、非极性溶剂化项(ΔG结合 Lipo)、氢键校正(ΔG结合 Hbond)、共价结合(ΔG结合 共价)、π-π 堆积校正(ΔG结合 堆积)、广义 Born 静电溶剂化能(ΔG结合 sol GB) 和 van der Waals 相互作用 (ΔG结合 vdW) 进行匹配。
- 通过平均为 MD 模拟中的每个快照确定的 ΔG绑定 值来得出最终的 ΔGbind,如方程 2 所示。
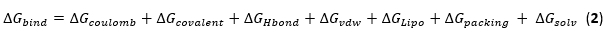
结果
受体网格生成和分子对接
Maestro 的受体网格生成工具用于正确表征随后对接的结合位点。共结晶配体用于定义网格。使用分子对接的 SP 精确 Glide 设置。使用 Schrödinger Maestro 中的 LigPrep 工具准备配体,以便使用 OPLS4 力场进行对接。对于分子动力学模拟,Desmond 中使用了 OPLS4 力场。力场是经典分子模拟的基础,其准确性对于药物发现中蛋白质-配体结合模拟的质量至关重要。对于 OPLS4,使用 Schrödinger Maestro 完成电荷和参数分配。与 OPLS2005 的默认参数相比,OPLS4 参数的应用导致能量和几何比较的显着改进。
这需要对接已知对 HIV-1 靶蛋白具有亲和力的特异性配体,从而能够全面分析配体分子和受体残基之间的相互作用。 表 1 显示了用于 QSAR 建模的化合物分类摘要。
对接 HBY561 后,通过将重新对接的配体与在结晶蛋白 1HQU 的活性位点中发现的配体进行比较来评估对接方案。对接和结晶的 HBY561 结构在补充文件 1(补充图 S1 和补充图 S2)中提供。为了评估停靠姿势和参考结构之间的相似性,小于 2.0 Å 的均方根偏差 (RMSD) 值被广泛认为是可靠停靠结果的标准。此阈值表明预测的结构与实验数据密切相关。在这项研究中,当将参考结构与停靠姿势进行比较时,配体的 RMSD 为 1.27 Å,如补充图 S3 中的红色所示。这表明对接方案足以完成这项工作,因此,所有配体都使用相同的设置对接。检查表 2 中所示的对接分数,相对于共结晶配体 HBY561 (-9.242 eV),依非韦伦和依曲韦林在 -10.432 eV 和 -9.647 eV 处显示出最有利的分数。补充文件 1 中的补充图 S4 显示了原始 HIV-1 蛋白与晶体配体、依曲韦林和依非韦伦之间的配体相互作用图。
氢键、π-π 堆叠和疏水相互作用是导致结合的主要力。HBY561 和指定蛋白 1HQU 之间出现了氢键的特定实例,明确涉及氨基酸残基 LYS101。这种键合模式反映了对配体 Efavirenz 和 Etravirine 的观察结果,如图 14 所示。
此外,除了分子间作用力、氢键和 π-π 堆叠外,疏水相互作用对于涉及 HBY561、Etravirine 和 Efavirenz 的多个蛋白质位置的结合至关重要。在 Efavirenz 中观察到 TYR318 和芳香环之间π-π 堆积。氢键和 π-π 堆叠对于维持配体和蛋白质之间的结合连接都是必不可少的。HBY_561、奈韦拉平、多拉韦林、依非韦伦和依曲韦林的配体相互作用图在 补充文件 1-SupplementalFigureS5 中提供。
这些分子间作用力影响蛋白质-配体相互作用,对于开发降低 HIV-1 中 AMR 的药物至关重要。它们在增强结合亲和力、特异性和作用机制方面的作用有助于设计能够有效靶向和抑制病毒的药物,从而解决 HIV-1 治疗中日益增长的抗菌素耐药性问题。
2D-QSAR 数据集制备
训练和测试阶段涉及 94 种化合物。这些化合物分为四类,每类代表与测试的特定蛋白质相关的配体。训练过程利用了 KNIME AutoQSAR 工作流程,其中 activity 、 HOMO 和 LUMO 被选为本研究的三个描述符。
2D-QSAR 生成
所有 94 个分子的活性值都通过实验数据确定(补充表 S1)。QSAR 模型生成的结果见 表 3。使用 补充表 S2 中的 1 类化合物进行 QSAR 建模,显示添加的 Ar 基团及其各自的活性值。四个等级的结果表明,获得了最高分 0.8223,R2 为 0.815,Q 2 为 0.8182,对应于等级 1。这与之前的目标是 R2 接近 1 和 Q2 大于 0.746 的标准一致。因此,我们选择了 1 类来训练我们的 QSAR 模型。虽然第 3 类和第 4 类的模型分别显示出 0.8172 和 0.6673 的出色相关值 R2 ,但它们与第 1 类的性能不匹配。
根据 Q2 评分 0.8223 和 R2 之间的差异应小于或等于 0.347 的标准,进行互相关以进一步验证所提出的 QSAR 模型的稳定性。我们针对类 1 提出的模型相差 0.0038。 图 15 提供了描述观察到的活动与预测活动的散点图。
HOMO-LUMO 能隙
确定最低未占据的分子轨道和最高的占据的分子轨道之间的能隙,通常称为 HOMO-LUMO 能隙,在表征分子在六种 NNRTI 化合物背景下的化学反应性和动力学稳定性方面起着至关重要的作用。前沿分子轨道在促进与 HIV 蛋白结合位点的电荷转移相互作用中起着关键作用。通过检查振动频率并确认不存在负或虚频,确保能量最小值的确认;随后,获得每个能量最小值的 HOMO 和 LUMO 值。较高的 HOMO 值表示分子作为电子供体的熟练程度,而较低的值表明它充当弱电子受体。 补充图 S6 表示 6 个优化 NNRTI 的 HOMO_LUMO 能隙。此外,由于分子具有很强的电子接受能力和生物活性,HOMO 和 LUMO 能级之间的能量间隙减小会强烈影响研究分子之间发生的分子间电荷转移相互作用48。
如 表 4 所示,能量间隙值的趋势遵循降序:依非韦伦 > 依曲韦林 > HBY-561 > 奈韦拉平 > 地拉韦定 > 多拉韦林 > 利匹韦林。观察到的 Efavirenz 和 Etravirine 的巨大能量间隙意味着对接分数的分析揭示了生物活性与 HOMO-LUMO 间隙之间的相关性。值得注意的是,抗病毒潜力随着 HOMO-LUMO 间隙值的增大而增加。它不仅表明化合物的稳定性,还表明它们与受体形成稳定相互作用的潜力。HOMO-LUMO 差距在理解分子的生物活性方面起着重要作用,尤其是在 HIV-1 药物设计的背景下。
列举
已经创建了各种工具来枚举虚拟库。用于枚举的工具包括 Schrödinger。它依赖于核心跳跃方法,其中通过用试剂化合物的片段替换核心结构上的一个或多个连接来创建文库49。Maestro v13.1 中的枚举工具用于向六个 NNRTI 中的每一个添加自定义侧基或原子。新化合物还用于预测活性。与最初优化的 NNRTI 分子相比,所列分子的预期活性值有所改善,如 表 5 所示。
计数配体活性值显示的改善是由于添加的自定义 R 基团影响了新提议的化合物和原始蛋白质的相互作用力而进行的计数过程。通过优化这些分子并计算它们的振动频率,为量子力学准备了列举的化合物。计算它们的能隙并与 NNRTI 的能隙进行比较。如 表 6 所示,总体观察结果表明,所列化合物比优化的化合物更稳定。
在表 6 中,观察到与优化化合物相比,所列化合物的能量隙显示出相似的趋势。这表明所列分子的化学性质保持不变,无论任何构象旋转。因此,他们能够与蛋白质的氨基酸残基保持重要的分子间作用力。
在按照协议第 6 节中概述的 NNRTI 执行普查过程之前,观察到的输出结果具有来自普查过程的初始对接分数,如表 7 中列举的对接分数列下所示。如方案第 4 节所述,进行分子对接过程以验证所列化合物的拟议对接分数。可以观察到,在重新对接枚举的化合物后,新的对接分数有所提高,如“重新对接的枚举配体”列所示。将列举化合物的重新停靠分数与 表 7 中“原始对接分数”列下表示的原始 NNRTI 的对接分数进行比较。可以观察到,对于被点算的化合物,HBY_561、依曲韦林、依非韦伦和多拉韦林的对接分数优于其等效的优化化合物。然而,Delavirdine 拥有与列举和优化的 Delavirdine 相同的对接分数。
分子动力学
HBY 561 与 LYS101、高电负性 N 原子和 OH 形成三个氢键。晶体配体或第三个分子同时与硫和氢建立两个氢键,并与 GLU138 建立第三个氢键。重要的是,π-π 堆叠和疏水相互作用对所涉及的分子间作用力有显著贡献。此外,额外氢键的存在对于分子动力学至关重要,特别是反映在得到的均方根偏差 (RMSD) 图中。总共观察到 Efavirenz 与非常电负性的氮原子、另一个非芳香环上的氧原子以及中心环处配体高电负性 N 之间的苯环和 TYR318 形成三个氢键。可以看到来自环的 N 与 OH 和 LY101 之间的第二个氢键。依曲韦林与 LYS101 具有三个氢键。多拉韦林与 GLU138 形成氢键。奈韦拉平与 LY101 有两个氢键。
在这种情况下,所有配体共享的氨基酸残基相互作用是 LYS101。尽管它们的结构不同,但它们都与相同的氨基酸残基相互作用。使用协议第 2.8 节中列出的参数进行 MD 模拟,以确定每个配体 (NNRTI 和列出的 NNRTI) 与 1HQU 活性位点结合的好坏程度。 图 16 中描述的配体相互作用表明蛋白质的氨基酸 LY101 与分子 HBY 561、奈韦拉平、依非韦伦和依曲韦林之间存在强大的氢键。从 表 7 中的比较中可以看出,这些强相互作用是每种化合物的高对接分数的原因。
为了评估每个配体的结合功效,包括 NNRTI 和 1HQU 活性位点的 NNRTI,进行了分子动力学模拟 (MDS)。具体来说,选择了四种列举的化合物,这些化合物的对接分数优于原始优化组合。这些选定的化合物经过 MDS 作为验证方法,以检查和观察每个分子在特定时期内与 HIV-1 蛋白的反应,同时考虑在配体存在下的原子间相互作用。
在开始对最近列举的游离寡糖进行 MDS 之前,必须确认模拟方案是否适合我们的系统。为了实现这一目标,第一步涉及运行游离蛋白 1HQU 的 MDS。蛋白质的活性位点内不存在配体(图 17A 和 补充图 S7)。高达 ~60 ns,蛋白质结构存在相当大的波动,产生高达 4.5 Å 的 Cα RMSD 偏移;之后,蛋白质似乎稳定下来,RMSD 波动为 ~3.5 Å,最长可达 200 ns。这种稳定性使我们相信 MD 方案适用于我们的蛋白质-配体复合物,这将在下一节中进行分析。
Etravirine 和点算的 Etravirine 的 200 ns MDS(图 17B,C)显示 Etravirine 的均方根偏差 (RMSD) 波动接近 5.0 Å 和 4.5 Å 的平衡。列举的 Etravirine 显示 RMSD 波动为 4.5 Å,平衡为 3.5 Å。这种稳定性表明,枚举的 Etravirine 可以成为治疗 HIV/AIDS 的潜在 NNRTI 配体。RMSD 小于 5 Å 的轨迹表示活性位点的蛋白质和配体之间存在强大的结合作用。上述所有化合物均采用这一观察结果,但列举化合物中的奈韦拉平和多拉韦林除外(补充文件1:补充图S8、补充图S9、补充图S10和补充图S11)。
图 18 和 图 19 进一步分析了 Etravirine、枚举的 Etravirine 与蛋白质之间的结合。分析的数据包括相互作用、配体-蛋白质和蛋白质-配体接触的直方图。与蛋白质结合的每个相应配体的相互作用接触直方图与蛋白质氨基酸残基和配体之间的相应相互作用力直接相关。LYS101 的高丰度对于 Etravirine 和枚举的 Etravirine 非常明显,并观察到可见的橙色粗带。观察到一条隐约可辨的浅橙色条带位于列举的 Etravirine 图表的下端,与 TYR181 相关。这种对应关系表明 GLU138 之间存在两种分子间吸引力。对配体及其列举形式的这一积极观察结果进行了比较,以分析更好的配体作为潜在的 NNRTI 化合物。根据提供的结果,列举的 Etravirine 具有用于治疗 HIV/AIDS 的潜力。
具有广义 Born 和表面积 (MM-GBSA) 计算的分子力学
在这项研究中,结合自由能 ΔG结合的能量输入的主要来源是范德华 ΔGVdW 相互作用的贡献。列举的依曲韦林的 ΔGVdW 为 -66.146 kcal/mol,高于其已知的 NNRTI 对应物,ΔGVdW 为 -64.669 kcal/mol。列举的 Etravirine 的较高 ΔGHbond 值为 -2.541 kcal/mol,表明配体和蛋白质之间氢吸引力的显着贡献。ΔG库仑 和 ΔG共价 对列举的依曲韦林的贡献(-17.976 和 2.807 kcal/mol)远大于已知的 NNRTI 对应物,后者分别为 -11.196 和 2.491。
Etravirine 和枚举的 Etravirine 与 1HQU 蛋白结合过程中观察到的结果在某种程度上是一致的。由于两个额外的氢键,发现列举的 Etravirine 优于其对应的 Etravirine。该研究还显示,列举的 Etravirine 是与已识别的口袋结合的首选。与依曲韦林 (-89.684 kcal/mol) 相比,列举的依曲韦林的负 ΔG结合 (-80.551 kcal/mol) 表明列举的依曲韦林是 HIV-1 RT 的良好抑制剂。

图 1:美国食品药品监督管理局批准的六种用于人类免疫缺陷病毒 1 的非核苷酸逆转录酶抑制剂的化学结构。 NVP = 奈韦拉平;DLV = 地拉韦定;EFV = 依非韦伦;ETV = 依曲韦林;RPV = 利匹韦林;DOR = 多拉韦林。 请单击此处查看此图的较大版本。
图 2:在本地计算机上的 Windows 上打开 Maestro Schrodinger 应用程序。(A) 在本地计算机中导航到 Maestro Schrodinger 应用程序。(B) 如何在本地计算机上打开和运行 Maestro Schrodinger 应用程序。 请单击此处查看此图的较大版本。

图 3:将 PDB 文件结构从本地计算机导入到 Schrödinger 中的项目窗口。 (A) Maestro Schrodinger 中的导入结构函数。(B) PDB ID 文本框。(C) 在本地计算机中下载的 PDB 文件。(D) Import 按钮允许导入插入的 PDB ID 文件。 请单击此处查看此图的较大版本。
图 4:导入 Schrodinger 项目窗口的 PDB 文件的结构。请单击此处查看此图的较大版本。

图 5:蛋白质制备工作流程。 (A) 蛋白质制备工作流程搜索界面。(B) 保存作业文件名并启动蛋白质制备过程。(C) 正在运行的作业的监控窗口。(D) 将配体分解成其各个组分。 请单击此处查看此图的较大版本。
图 6:配体制备工作流程。 (A) 将结构从本地计算机导入蛋白质制备薛定谔项目窗口。(B) 寻找配体制备过程。(C) 配体制备工作流程窗口。 请单击此处查看此图的较大版本。

图 7: 几何和优化工作流程。 (A) 用于几何优化的 GaussView 菜单窗口。(b) GaussView 的 Calculate 选项卡中的可用作业类型。(C) GaussView 中 Link0 选项卡下的可用选项。 请单击此处查看此图的较大版本。
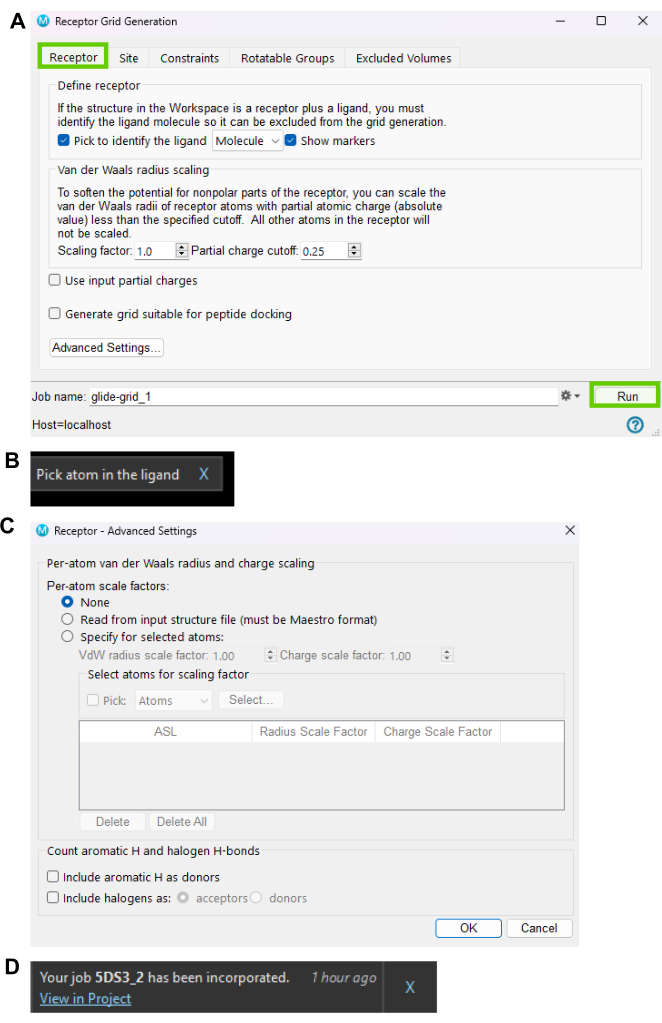
图 8:Glide 新体生成和分子对接工作流程。 (A) 受体网格生成工作流程界面。(B) 用于选择配基内原子的弹出通知。(C) 受体高级设置。(D) 作业完成通知。 请单击此处查看此图的较大版本。
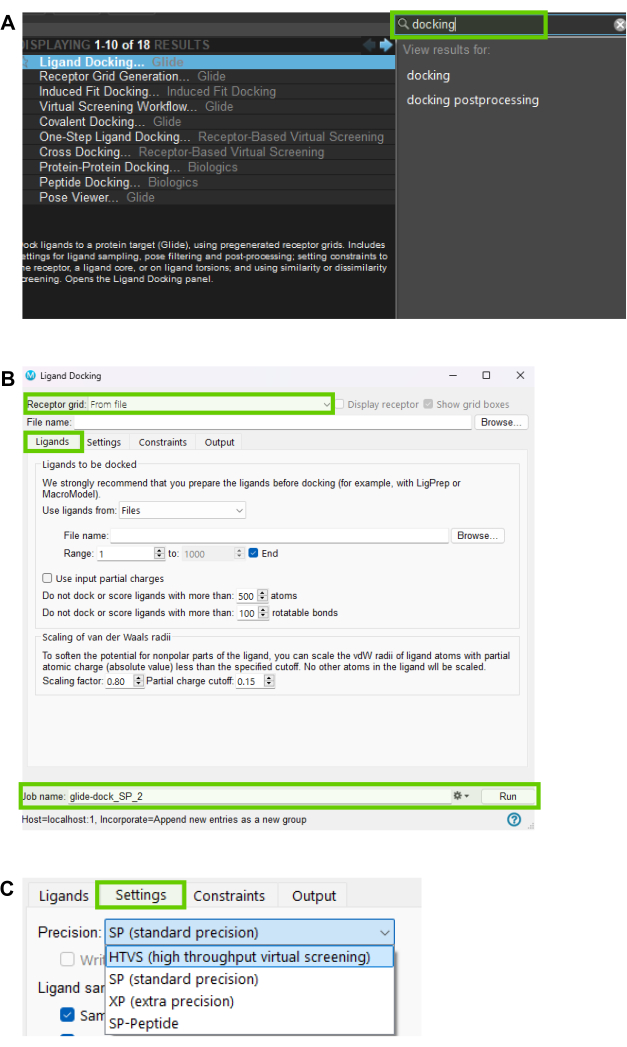
图 9:滑行配体对接。 (A) 滑行配体对接搜索界面。(b) 配体对接界面。(C) 滑行停靠的精度设置。 请单击此处查看此图的较大版本。

图 10:KNIME QSAR 制备工作流程。 (A) 在 KNIME 社区中心网页上搜索 AutoQSAR 节点。(B) 下载按钮以获取 AutoQSAR KNIME 节点。(C) 导入下载的 AutoQSAR KNIME 工作流程。(D) 构建 QSAR 模型时配体的配置设置。 请单击此处查看此图的较大版本。

图 11:在 Maestro Schrodinger 中使用 Ligand Designer 对配体进行计数。 (A) 在 Schrodinger 中搜索 Ligand Designer 选项。(B) 执行普查过程的工作流程列表。 请单击此处查看此图的较大版本。

图 12:HOMO-LUMO 生成工作流程。 (A) 访问 GaussView 中 Tools 选项卡中的分子编辑器选项。(B) 加载现有的 Chk 或 FChk 文件以生成前沿分子轨道。(C) GaussView 中 HOMO 和 LUMO 前沿轨道的图示。(D) 显示 HOMO 和 LUMO 前沿轨道的可视化窗口。(E) 拯救 HOMO 和 LUMO 的前沿轨道。(f) GaussView 中的显示格式界面。 请单击此处查看此图的较大版本。

图 13:分子动力学制备、设置、制备和执行工作流程。 (A) Desmond System Builder:确定刚性水模型的溶剂化选项。(B) 用于确定箱形的 Desmond System Builder 边界选项。(c) Desmond System Builder:计算盒子大小的方法。 请单击此处查看此图的较大版本。
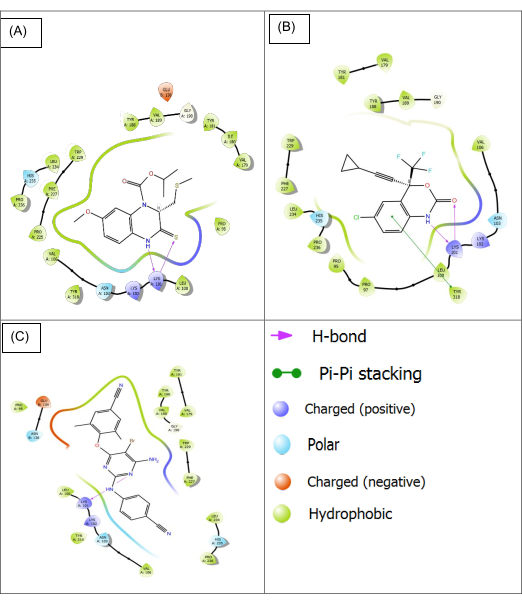
图 14:1HQU 和 3 个顶部对接配体之间的配体相互作用图。 蛋白质 (1HQU) 和 (A) 晶体配体 (HBY561)、(B) 依非韦伦和 (C) 依曲韦林之间的相互作用力。这些力量影响蛋白质-配体相互作用,对于开发降低 HIV-1 抗菌素耐药性的药物至关重要。它们在增强结合亲和力、特异性和作用机制方面的作用有助于设计能够有效靶向和抑制病毒的药物,从而解决 HIV-1 治疗中日益增长的抗菌素耐药性问题。 请单击此处查看此图的较大版本。

图 15:散点图显示了 QSAR 模型第 1 类的观测活动与预测活动。 该图表示作为训练集的类 1 与作为测试集的 NNRTI 化合物之间的拟合,以给出预测活动值。缩写:NNRTI = 非核苷酸逆转录酶抑制剂;QSAR = 定量构效关系。 请单击此处查看此图的较大版本。
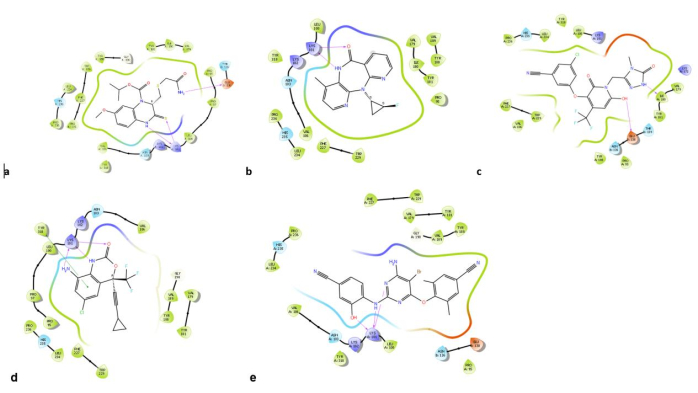
图 16:配体相互作用图。 蛋白质与 (A) 列举的晶体配体 HBY_561,(B) 列举的奈韦拉平,(C) 列举的多拉韦林,(D) 列举的依非韦伦,以及 (E) 列举的依曲韦林之间的相互作用力。 请单击此处查看此图的较大版本。
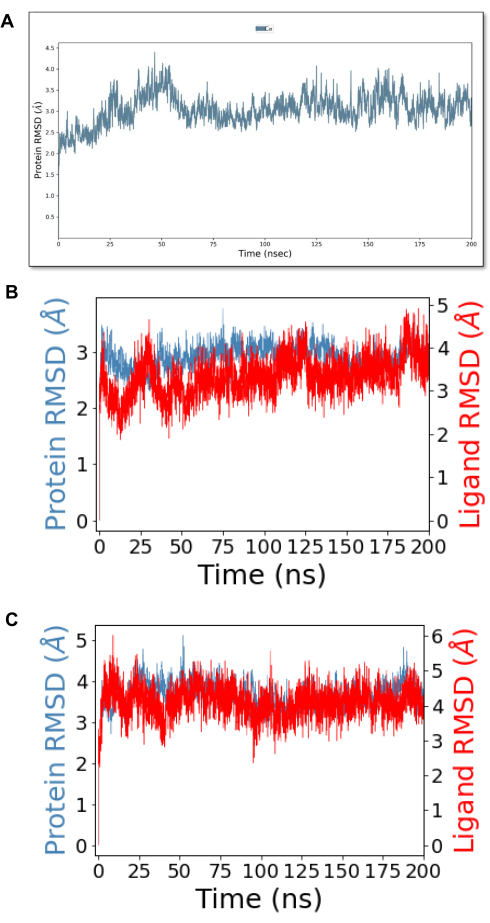
图 17:游离蛋白 Etravirine 和枚举的 Etravirine 的分子动力学模拟相互作用图。 (A) 游离蛋白的分子动力学相互作用图。(B) 依曲韦林的分子动力学相互作用图。(C) 列举的 Etravirine 的分子动力学相互作用图。 请单击此处查看此图的较大版本。
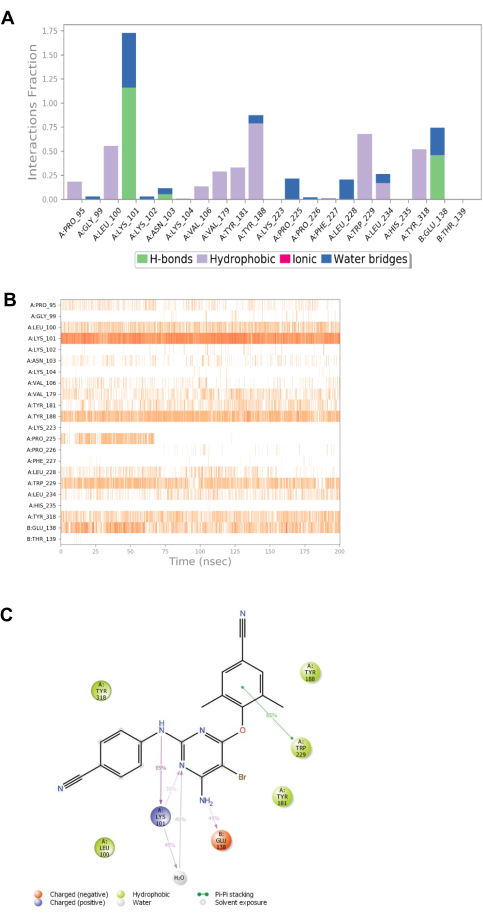
图 18:Etravirine 和蛋白质之间的相互作用接触直方图。 (A) 依曲韦林的蛋白质-配体接触时间表。(B) 蛋白质-配体相互作用随时间变化的时间表,包括 H 键、疏水、离子和水桥接触。(C) 显示配体原子和蛋白质残基之间详细相互作用的示意图。仅显示发生在仿真时间超过 30% (0.00 到 200 ns) 的交互。 请单击此处查看此图的较大版本。
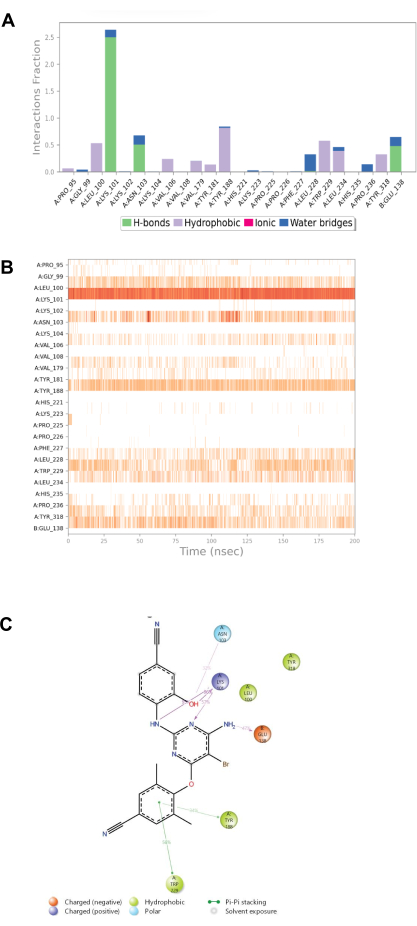
图 19:枚举的 Etravirine 和蛋白质之间的相互作用接触直方图。 (A) 列举的 Etravirine 的蛋白质-配体接触时间表。(B) 蛋白质-配体相互作用随时间变化的时间表,包括 H 键、疏水、离子和水桥接触。(C) 显示配体原子和蛋白质残基之间详细相互作用的示意图。仅显示发生在仿真时间超过 30% (0.00 到 200 ns) 的交互。 请单击此处查看此图的较大版本。
| 类 | 蛋白靶标 | 复合范围 | 所选化合物的总数 |
| 1 | NL4-3 野生型 HIV-1 | (8a1-8e5 – EC50 (nM)a) | 25 |
| 2 | IIIB WT HIV-1 | 13a1-13d6 - EC50 (nM)a | 23 |
| 3 | RES056 NNRTI 抗性菌株 | 13a1-13d6 - EC50 (nM)a | 23 |
| 4 | ROD HIV-2 菌株 | 13a1-13d6 - EC50 (nM)a | 23 |
| 选择用于 QSAR 建模的化合物总数 | 94 |
表 1:QSAR 建模的化合物分类标准总结。 Kang 及其同事50 合成了 94 种二氢呋喃[3,4-d] 嘧啶化合物的数据集,用于靶向各种 HIV 菌株,即 NL4-3 野生型 HIV-1、IIIB WT HIV-1、RES056 NNRTI 耐药菌株和 ROD HIV-2 菌株。这些嘧啶衍生物根据其靶蛋白分为四类,用于准备进行我们的 QSAR 训练。该表总结了这些分子如何根据其实验 EC 50 值分为四类,EC50 值代表药物的效力,作为药物发挥其最大作用的 50% 的浓度。缩写:NNRTI = 非核苷酸逆转录酶抑制剂;QSAR = 定量构效关系。
| 蛋白质名称 | 配体 | 停靠分数 |
| 1HQU | 依非韦伦 | -10.432 |
| 依曲韦林 | -9.647 | |
| HBY_561 | -9.242 | |
| 多拉韦林 | -9.04 | |
| 奈韦拉平 | -8.825 | |
| 利匹韦林 | -7.722 | |
| 地拉韦定 | -6.519 |
表 2:六种优化的 NNRTI 和 HIV-1 蛋白的对接评分。 对接分数适用于正在调查的 6 个 NNRTI。负分越高表示配体与蛋白质之间的结合效果越好。相对于共结晶配体 HBY561 (-9.242) 的对接分数,依非韦伦和依曲韦林在 -10.432 eV 和 -9.647 eV 处显示出最有利的分数。潜在的配体是那些具有更负的对接分数,小于 -9.242 eV 的配体。
| 药品类别 | 活动拟合率为 85% | 列 1 | 列 2 | 列 3 | 列 4 | 列 5 |
| 得分 | 标清 | R2 | 有效值 | 问题2 | Q2 MW (零假设) | |
| 1 | 0.8223 | 0.3268 | 0.815 | 0.2479 | 0.8185 | 0.1462 |
| 2 | 0.5671 | 0.2996 | 0.5067 | 0.1996 | 0.2264 | 0.4736 |
| 3 | 0.8172 | 0.3692 | 0.8114 | 0.1536 | 0.9065 | -1.1532 |
| 4 | 0.6673 | 0.367 | 0.6436 | 0.1709 | 0.8852 | 0.1445 |
| *SD – 标准差, | ||||||
| R2 – 实际活动值和预测活动值之间的训练集相关性, | ||||||
| Q2 – 测试集实际和预测活动相关性。 | ||||||
| RMSE - 均方根误差 | ||||||
表 3:2D-QSAR 模型统计参数。 该表显示了标准差、实际活动值和预测活动值之间的训练集相关性 (R2) 以及每个类的测试集实际和预测活动相关性高分 (Q2)。较高的分数 (R2) 代表类。
| 配体 | 同性恋者 | LUMO | E间隙 |
| 依非韦伦 | -0.2242 | -0.06943 | 0.15477 |
| 依曲韦林 | -0.21408 | -0.08141 | 0.13267 |
| 奈韦拉平 | -0.20602 | -0.07759 | 0.12843 |
| HBY_561 | -0.19687 | -0.0748 | 0.12207 |
| 地拉韦定 | -0.19651 | -0.07958 | 0.11693 |
| 多拉韦林 | -0.22413 | -0.10916 | 0.11497 |
| 利匹韦林 | -0.21343 | -0.11216 | 0.10127 |
表 4:优化后的 NNRTI 的 HOMO-LUMO 能隙。 该表显示了优化 6 个 NNRTI 后获得的 HOMO-LUMO 能隙的结果。
| 配体 | 优化的配体 | 枚举配体 |
| 多拉韦林 | 7.229 | 7.374 |
| 利匹韦林 | 7.302 | 7.279 |
| 依曲韦林 | 7.229 | 7.374 |
| 依非韦伦 | 7.229 | 7.323 |
| 地拉韦定 | 7.302 | 7.302 |
| 奈韦拉平 | 7.229 | 6.988 |
| HBY_561 | 7.229 | 7.323 |
表 5:优化 NNRTI 与相应列举 NNRTI 的预测活性评分。 该表比较了优化配体和枚举配体之间的预测活性评分。活性分数越高,化合物作为潜在药物先导物的能力就越好。
| 配体 | 优化后的能量差距 | 枚举后的能量差距 | 优化化合物和枚举化合物之间的间隙差异 |
| 多拉韦林 | 0.115 | 0.126 | 0.011 |
| 利匹韦林 | 0.101 | 0.127 | 0.030 |
| 依曲韦林 | 0.133 | 0.126 | 0.010 |
| 依非韦伦 | 0.155 | 0.126 | 0.030 |
| 地拉韦定 | 0.117 | 0.126 | 0.010 |
| 奈韦拉平 | 0.128 | 0.126 | 0.002 |
| HBY_561 | 0.122 | 0.126 | 0.004 |
表 6:列举的 NNRTI 的预测能隙与优化后的 NNRTI 的原始能隙的比较。 该表显示了优化配体和列举配体之间的 HOMO-LUMO 能隙以及它们之间的能隙差异的比较。
| 枚举配体 | 5 个属性的规则 | 列 1 | 列 2 | 列 3 | 列 4 | 列 5 | 新增侧组 | 枚举的停靠分数 | 原始对接分数 | 重新对接的枚举配体 |
| 阿洛格 | PSA | HBD | HBA | 分子量 | 城市规划区 (MPO) | |||||
| 多拉韦林 | 2.4 | 125.9 | 2 | 8 | 441.8 | 0.49 | 羟基 | -8.894 | -9.04 | -9.739 |
| 依曲韦林 | 4.4 | 140.9 | 3 | 8 | 451.3 | 0.37 | 羟基 | -10.258 | -9.647 | -10.517 |
| 依非韦伦 | 3.7 | 64.3 | 2 | 3 | 330.7 | 0.75 | 胺 | -10.284 | -10.432 | -11.025 |
| 奈韦拉平 | 2.6 | 58.1 | 1 | 4 | 284.3 | 0.73 | 氟化物 | -9.112 | -8.825 | -9.445 |
| HBY_561 | 2.4 | 93.9 | 2 | 6 | 358.5 | 0.66 | 酰胺 | -9.596 | -9.242 | -10.1 |
| AlogP - (计算的辛醇-水分配系数的对数)。 | ||||||||||
| PSA - (极地表面积) | ||||||||||
| HBD - (氢键供体) | ||||||||||
| HBA - (氢键受体) | ||||||||||
| MW - (分子量) | ||||||||||
| MPO - (多参数优化) | ||||||||||
表 7:原始 NNRTI 和枚举 NNRTI 之间的对接分数比较。 该表显示了枚举的停靠分数之间的比较,这些分数是枚举后的停靠分数。原始对接分数为优化后的 NNRTI 的对接分数。重新停靠的枚举分数是被列举的配体的停靠分数。由于计数过程给出了预测对接分数,因此必须使用与优化配体相同的方法重新对接配体。添加的组是在计数过程中添加到配体中的组。
| 配体 | ΔG绑定 | ΔG库仑 | ΔG共价 | ΔGHbond (Hbond) | ΔG脂肪 | ΔG填料 | ΔG求解器 | ΔGVdW |
| 依曲韦林 | -80.551 | -11.196 | 2.491 | -1.509 | -27.447 | -4.826 | 26.605 | -64.669 |
| 列举的依曲韦林 | -89.684 | -17.976 | 2.807 | -2.541 | -27.652 | -4.211 | 26.034 | -66.146 |
| HBY561型 | -79.664 | -12.261 | 0.994 | -0.521 | -26.052 | -1.278 | 18.624 | -59.169 |
| 列举 HBY561 | -82.719 | -13.443 | 1.534 | -0.603 | -27.053 | -1.210 | 20.600 | -62.544 |
| 依非韦伦 | -71.372 | -12.984 | 1.151 | -0.834 | -25.353 | -1.379 | 14.472 | -46.44 |
| 列举的 Efavirenz | -79.125 | -18.602 | 1.753 | -2.159 | -25.409 | -1.198 | 16.619 | -50.129 |
表 8:1HQU 上选定配体的 MMGBSA reXts。 该表显示了具有广义 Born 和表面积计算的分子力学,其中显示了蛋白质-配体复合物的平均结合自由能 (ΔGbind)。该表显示了最初优化的配体之间的比较,这些配体相对于晶体配体及其列举的对应物显示出良好的对接分数。所选化合物具有较高的对接分数和良好的分子动力学模拟 RMSD 平衡波动,也应通过显示(ΔG结合) 的最负结合自由能来满足 MMGBSA 计算。在这种情况下,枚举的 Etravirine 满足两个计算参数。
补充文件 1:本研究中获得的其他 rXts。请点击此处下载此文件。
讨论
DFT26 方法用于分析各种嘧啶衍生物的电子性质和稳定性,这在类似研究中并非如此。使用 DFT 可以更深入地了解量子水平的分子相互作用,从而增强 NNRTI 的设计过程。在本研究中,我们选择了 6 个活性值未知的 NNRTIs。我们从 PubChem 数据库中检索了它们的分子结构,并使用 Gaussian 16 修订版 C0133 量子力学软件包中的明尼苏达 15 局部 (MN15-L)32 泛函和 6-31++G (d,p)2 基集进行了几何优化。为了设置仿真输入文件,我们使用了 GaussView 6.026。然后将量子结构对接到 HIV-1 蛋白的活性位点。
该研究采用了 QSAR 方法51,该方法旨在根据 NNRTI 的化学结构预测 NNRTI 的生物活性。该模型是使用 94 种二氢呋喃[3,4-d] 嘧啶衍生物的数据集生成的,能够识别影响抗病毒活性的关键官能团。整合稳健的 QSAR 模型是一项至关重要的进步,因为它有助于在设计过程的早期过滤化合物,从而提高药物发现的有效性。
考虑了新化合物的预测活性,并验证了对接分数。先进的分子对接技术探索了 NNRTIs 与 HIV-1 逆转录酶之间的结合相互作用。这得到了较长时间 (200 ns) 的分子动力学模拟的补充,提供了对药物-蛋白质复合物在生理条件下的稳定性和行为的见解。分子动力学52 验证了对接研究的结果。通过模拟药物-酶复合物随时间的行为,我们可以评估静态对接研究中可能无法捕获的动态稳定性和相互作用53。这种方法对 NNRTI 在生物系统中的表现提供了更现实的评估。模拟结果表明,Etravirine 产生的 RMSD 波动约为 4.5 Å,而列举的 Etravirine 产生的 RMSD 波动为 3.5 Å。Etravirine 和枚举的 Etravirine 之间存在各种相似之处,枚举的化合物在蛋白质的活性位点内具有增强的氨基酸相互作用。较低的 RMSD、改进的氨基酸相互作用和最高的结合自由能都表明,列举的 Etravirine 可以作为 HIV/AIDS 治疗的可行替代品。
使用计数技术54 生成立体异构体并执行等排体扫描是本研究的一个值得注意的方面。这种方法允许研究人员通过系统地修改现有结构来探索潜在 NNRTI 的更广阔的化学空间,这可能导致发现具有更高疗效和更低耐药性的化合物。
该研究整合了源自量子力学计算的 HOMO-LUMO 分析,以评估化合物的反应性和稳定性。在类似的研究中,这一方面经常被忽视55,但它为影响生物活动的电子特性提供了有价值的见解
在本研究中使用 MMGBSA 方法估计列举的 NNRTI 作为潜在的 HIV-1 逆转录酶 (RT) 抑制剂的结合自由能提出了一个新的方面,增强了这项工作对 HIV 治疗开发的重要性和潜在影响。
与对应的 Etravirine 相比,MMGBSA 在本研究中的应用提供了对列举的 Etravirine 的结合自由能的更准确估计。通过计算结合自由能值,该研究定量评估了列举的 Etravirine 的结合亲和力,其比标准 Etravirine 高得多 (9.133 kcal/mol)。结合能计算中的这种详细程度有助于更细致地了解结构修饰如何影响 NNRTI 的功效,这在可能仅依赖于定性评估的类似研究中通常缺乏。
报道的 ΔG共价 值为 1.477 kcal/mol,进一步表明了所列化合物的相互作用强度。比较方法具有创新性,因为它不仅确定了有前途的候选药物,而且还将其置于现有疗法的更广泛背景下,为未来的 NNRTI 开发提供了基准。
表 8 中显示的比较还表明,列举的 Etravirine 可以成为用作替代 ART 的潜在化合物,因为与 Etravirine、HBY56、Etravirine 及其列举的对应物相比,其结合自由能 (ΔG结合) 是最高的。
我们的研究结果可能会显着影响 HIV 感染的新型治疗药物的开发。此外,这种方法可能允许确定最有前途的抗 HIV 候选药物。这些发现可能为新型 HIV-1 NNRTI 的合理设计铺平道路。
将 MMGBSA 与其他计算方法(如分子对接、QSAR 建模和分子动力学模拟)相结合,增强了研究结果的稳健性。这种集成方法可以对化合物进行全面评估,从结构优化到生物环境中的动态行为。这种方法在 NNRTI 研究中相对较新,NNRTI 研究通常侧重于孤立的方法,而没有对药物设计过程的整体看法。该研究强调了几个有希望的结果,并增强了对 NNRTI 和逆转录酶之间分子相互作用的理解,这可以为未来的药物设计工作提供信息。
虽然 NNRTI 开发领域的其他研究通常侧重于单一计算方法或基本对接分析,但这项研究通过将量子化学计算与分子动力学和 QSAR 建模相结合,采用系统枚举方法扩大了 NNRTI 的潜在化学空间,并利用全面的 计算机模拟 框架,该框架不仅可以预测结合亲和力,还可以评估药物-酶相互作用的稳定性和动力学。
总体而言,这项研究的新颖性在于其多方面的方法,利用先进的计算技术来解决 HIV 治疗中耐药性的挑战,从而为开发更有效的 NNRTI 铺平了道路。研究结果强调了开发更有效的 NNRTI 的潜力,这些 NNRTI 可以克服当前疗法的局限性。
该协议中描述的方法的一些未来应用包括以下内容。
与机器学习集成
未来的研究可以将 MMGBSA 与机器学习算法相结合,以提高绑定自由能估计的预测能力。通过在大型数据集上训练模型,研究人员可以提高预测的准确性和可靠性,从而更好地识别有前途的 NNRTI 候选者。
在基于片段的药物设计中的应用
MMGBSA 可有效用于基于片段的药物设计,其中小分子片段经过优化以提高结合亲和力。这种方法可能导致鉴定对 HIV-1 具有增强效力和选择性的新型 NNRTIs。
耐药机制探索
该技术可用于研究 NNRTIs 与 HIV-1 突变株的结合相互作用。通过比较 NNRTI 与野生型和耐药性变体的结合自由能,研究人员可以深入了解耐药机制,并为下一代抑制剂的设计提供信息。
药代动力学特性评价
MMGBSA 还可用于评估 NNRTI 的药代动力学特性,例如溶解度和渗透性。通过了解这些特性与结合亲和力的关系,研究人员可以优化候选药物以获得更好的治疗特征。
披露声明
作者声明,他们没有已知的竞争性经济利益或个人关系,这些利益或个人关系似乎可能会影响本文报告的工作。
致谢
作者要感谢高性能计算中心 (CHPC) 提供计算资源,并感谢约翰内斯堡大学化学科学系。
材料
| Name | Company | Catalog Number | Comments |
| GaussView | GaussView | V6.1.1 | |
| KNIME | KNIME | V4.7.1 | |
| Schrodinger Maestro V13.6 | SCHRODINGER INC. | License release year 2023-2 |
参考文献
- Fauci, A. S. Lane, H. C. Four decades of HIV/AIDS - much accomplished, much to do. N Engl J Med. 383 (1), 1-4 (2020).
- Kumar, V., Kishor, S., Ramaniah, L. M. Chemical reactivity analysis of deoxyribonucleosides and deoxyribonucleoside analogues (NRTIs): A first-principles density functional approach. J Mol Model. 18, 3969-3980 (2012).
- Shafer, R. W. Vuitton, D. A. Highly active antiretroviral therapy (haart) for the treatment of infection with human immunodeficiency virus type 1. Biomed Pharmacother. 53 (2), 73-86 (1999).
- Ginat, D. T., Schaefer, P. W. Highly active antiretroviral therapy (HAART). In Neuroimaging Pharmacopoeia. Ginat, D. T., Small, J. E., Schaefer, P. W. (Eds) 229-238 (2022).
- Murray, C. J. L. et al. Global burden of bacterial antimicrobial resistance in 2019: A systematic analysis. Lancet. 399 (10325), 629-655 (2022).
- Razzaque, M. S. Commentary: Microbial resistance movements: An overview of global public health threats posed by antimicrobial resistance, and how best to counter. Front Public Health. 8, 629120-629120 (2021).
- UNAIDS. Global HIV & AIDS statistics - Fact sheet. https://www.unaids.org/en/resources/fact-sheet (2023).
- Li, D. et al. HIV-1 pretreatment drug resistance and genetic transmission network in the southwest border region of China. BMC Infect Dis. 22 (1), 741 (2022).
- WHO. Fact sheet: HIV drug resistance. https://www.who.int/news-room/fact-sheets/detail/hiv-drug-resistance (2023).
- Hunt, G. M. et al. Prevalence of HIV-1 drug resistance amongst newly diagnosed HIV-infected infants age 4-8 weeks, enrolled in three nationally representative PMTCT effectiveness surveys, South Africa: 2010, 2011-12 and 2012-13. BMC Infect Dis. 19 (Suppl 1), 787-787 (2019).
- Koay, W. L. A., Kose-Otieno, J., Rakhmanina, N. HIV drug resistance in children and adolescents: Always a challenge? Curr Epidemiol Rep. 8 (3), 97-107 (2021).
- Wang, Z. et al. Contemporary medicinal chemistry strategies for the discovery and development of novel HIV-1 non-nucleoside reverse transcriptase inhibitors. J Med Chem. 65 (5), 3729-3757 (2022).
- Mbayo, V., Sookan, T. Correction to: Effects of a resistance training programme in people living with HIV in Zimbabwe. Sport Sci Health. 16 (4), 775 (2020).
- Krawczyk, C. S. et al. Factors associated with delayed initiation of HIV medical care among infected persons attending a Southern HIV/AIDS clinic. South Med J. 99 (5), 472-481 (2006).
- Namasivayam, V. et al. The journey of HIV-1 non-nucleoside reverse transcriptase inhibitors (NNRTIs) from lab to clinic. J Med Chem. 62 (10), 4851-4883 (2019).
- Patel, P. H. Zulfiqar, H. Reverse transcriptase inhibitors. In StatPearls. Treasure Island (FL)StatPearls Publishing (2023).
- Tan, J. J. et al. Therapeutic strategies underpinning the development of novel techniques for the treatment of HIV infection. Drug Discov Today. 15 (5-6), 186-197 (2010).
- Liu, N. et al. Novel HIV-1 non-nucleoside reverse transcriptase inhibitor agents: Optimization of diarylanilines with high potency against wild-type and rilpivirine-resistant e138k mutant virus. J Med Chem. 59 (8), 3689-3704 (2016).
- Anta, L. et al. Rilpivirine resistance mutations in HIV patients failing non-nucleoside reverse transcriptase inhibitor-based therapies. AIDS. 27 (1), 81-85 (2013).
- Sarafianos, S. G. et al. Structure and function of HIV-1 reverse transcriptase: Molecular mechanisms of polymerization and inhibition. J Mol Biol. 385 (3), 693-713 (2009).
- Vingerhoets, J. et al. Tmc125 displays a high genetic barrier to the development of resistance: Evidence from in vitro selection experiments. J Virol. 79 (20), 12773-12782 (2005).
- Ripamonti, D., Bombana, E., Rizzi, M. Rilpivirine: Drug profile of a second-generation non-nucleoside reverse transcriptase hiv-inhibitor. Expert Rev Anti-infect Ther. 12 (1), 13-29 (2014).
- Lambert-Niclot, S. et al. Prevalence of pre-existing resistance-associated mutations to rilpivirine, emtricitabine and tenofovir in antiretroviral-naive patients infected with B and non-B subtype HIV-1 viruses. J Antimicrob Chemother. 68 (6), 1237-1242 (2013).
- Wainberg, M. A. Combination therapies, effectiveness, and adherence in patients with HIV infection: Clinical utility of a single tablet of emtricitabine, rilpivirine, and tenofovir. HIV AIDS (Auckl). 5, 41-49 (2013).
- Namasivayam, V. et al. The journey of HIV-1 non-nucleoside reverse transcriptase inhibitors (NNRTIs) from lab to clinic. J Med Chem. 62 (10), 4851-4883 (2018).
- Jordaan, M. A., Ebenezer, O., Damoyi, N., Shapi, M. Virtual screening, molecular docking studies and DFT calculations of FDA approved compounds similar to the non-nucleoside reverse transcriptase inhibitor (NNRTI) efavirenz. Heliyon. 6 (8), e04642 (2020).
- Soltani, A. et al. Application of molecular docking for the development of improved HIV-1 reverse transcriptase inhibitors. Curr Comput Aided Drug Des. 17 (4), 538-549 (2021).
- Vanangamudi, M., Palaniappan, S., Kathiravan, M. K., Namasivayam, V. Strategies in the design and development of non-nucleoside reverse transcriptase inhibitors (NNRTIs). J Viruses. 15 (10), 1992 (2023).
- Mohapatra, R. K. et al. Computational investigations of three main drugs and their comparison with synthesized compounds as potent inhibitors of SARS-CoV-2 main protease (Mpro): DFT, QSAR, molecular docking, and in silico toxicity analysis. J King Saud Univ Sci. 33 (2), 101315-101315 (2021).
- Kim, S. et al. Pubchem substance and compound databases. Nucleic Acids Res. 44 (D1), D1202-D1213 (2016).
- Ofem, M. I. et al. Synthesis, spectral characterization, and theoretical investigation of the photovoltaic properties of (E)-6-(4-(dimethylamino) phenyl) diazenyl)-2-octyl-benzoisoquinoline-1, 3-dione. BMC Chem. 16 (1), 109 (2022).
- Yu, H. S., He, X., Truhlar, D. G. MN15-L: A new local exchange-correlation functional for kohn-sham density functional theory with broad accuracy for atoms, molecules, and solids. J Chem Theory Comput. 12 (3), 1280-1293 (2016).
- Sasitha, T. John, W. J. Design, docking, and DFT investigations of 2,6-bis(3,4-dihydroxyphenyl)-3-phenethylpiperidin-4-one. Heliyon. 7 (2), e06127-e06127 (2021).
- Maurya, S. K., Maurya, A. K., Mishra, N., Siddique, H. R. Virtual screening, ADME/T, and binding free energy analysis of anti-viral, anti-protease, and anti-infectious compounds against nsp10/nsp16 methyltransferase and main protease of SARS CoV-2. J Recept Signal Transduct Res. 40 (6), 605-612 (2020).
- Singh, A. K. et al. Current insights and molecular docking studies of HIV-1 reverse transcriptase inhibitors. Chem Biol Drug. 103 (1), e14372 (2024).
- Kralj, S., Jukič, M., Bren, U. Comparative analyses of medicinal chemistry and cheminformatics filters with accessible implementation in konstanz information miner (KNIME). Int J Mol Sci. 23 (10), 5727 (2022).
- Bastikar, V., Bastikar, A., Gupta, P. P. Quantitative structure-activity relationship-based computational approaches. Computational Approaches for Novel Therapeutic and Diagnostic Designing to Mitigate SARS-CoV-2 Infection. 191-205 (2022).
- Mazouin, B., Schöpfer, A. A., Von Lilienfeld, O. A. Selected machine learning of HOMO-LUMO gaps with improved data-efficiency. Mater Adv. 3 (22), 8306-8316 (2022).
- Singh, V. K. et al. In silico design, synthesis and anti-HIV activity of quinoline derivatives as non-nucleoside reverse transcriptase inhibitors (NNRTIs). Comput Biol Chem. 98, 107675 (2022).
- Lanka, G. et al. Pharmacophore-based virtual screening, 3D QSAR, docking, ADMET, and MD simulation studies: An in silico perspective for the identification of new potential HDAC3 inhibitors. Comput Biol Med. 166, 107481 (2023).
- Singh, K. D. Muthusamy, K. Molecular modeling, quantum polarized ligand docking and structure-based 3D-QSAR analysis of the imidazole series as dual AT1 and ETA receptor antagonists. Acta Pharmacol Sin. 34 (12), 1592-1606 (2013).
- Salo-Ahen, O. M. et al. Molecular dynamics simulations in drug discovery and pharmaceutical development. Processes. 9 (1), 71 (2020).
- Raniolo, S. Limongelli, V. Improving small-molecule force field parameters in ligand binding studies. Front Mol Biosci. 8, 760283 (2021).
- Lu, C. et al. OPLS4: Improving force field accuracy on challenging regimes of chemical space. J Chem Theory Comput. 17 (7), 4291-4300 (2021).
- Goswami, N., Singh, A., Bharadwaj, S., Sahoo, A. K., Singh, I. K. Targeting neuroblastoma by small-molecule inhibitors of human ALYREF protein: Mechanistic insights using molecular dynamics simulations. J Biomol Struct Dyn. 42 (3), 1352-1367 (2023).
- Chirico, N. Gramatica, P. Real external predictivity of QSAR models. Part 2. New intercomparable thresholds for different validation criteria and the need for scatter plot inspection. J Chem Inf Model. 52 (8), 2044-2058 (2012).
- Tabti, K., Sbai, A., Maghat, H., Lakhlifi, T., Bouachrine, M. Computational exploration of the structural requirements of triazole derivatives as colchicine binding site inhibitors. ChemistrySelect. 8 (26), e202301707 (2023).
- Miar, M., Shiroudi, A., Pourshamsian, K., Oliaey, A. R., Hatamjafari, F. Theoretical investigations on the HOMO-LUMO gap and global reactivity descriptor studies, natural bond orbital, and nucleus-independent chemical shifts analyses of 3-phenylbenzo[D]thiazole-2(3H)-imine and its para-substituted derivatives: Solvent and substituent effects. J Chem Res. 45 (1-2), 147-158 (2020).
- Saldivar-Gonzalez, F., Huerta-García, C., Medina-Franco, J. Chemoinformatics-based enumeration of chemical libraries: A tutorial. J Cheminf. 12, 64-64 (2020).
- Kang, D. et al. Identification of dihydrofuro[3,4-d]pyrimidine derivatives as novel HIV-1 non-nucleoside reverse transcriptase inhibitors with promising antiviral activities and desirable physicochemical properties. J Med Chem. 62 (3), 1484-1501 (2019).
- Viira, B., Garcia-Sosa, A. T., Maran, U. Chemical structure and correlation analysis of HIV-1 NNRT and NRT inhibitors and database-curated, published inhibition constants with chemical structure in diverse datasets. J Mol Graph. 76, 205-223 (2017).
- Javed, M. R. CADD and molecular dynamic simulations: Potential impacts to conventional medicines. Comb Chem High Throughput Screen. 25 (4), 658-659 (2022).
- Jiang, X. et al. Exploiting the tolerant region i of the non-nucleoside reverse transcriptase inhibitor (NNRTIi) binding pocket. Part 2: Discovery of diarylpyrimidine derivatives as potent HIV-1 NNRTIs with high FSP3 values and favorable drug-like properties. Eur J Med Chem. 213, 113051 (2021).
- Zhang, T., Jiang, S., Li, T., Liu, Y., Zhang, Y. Identified isosteric replacements of ligands' glycosyl domain by data mining. ACS Omega. 8 (28), 25165-25184 (2023).
- Sule, L., Gupta, S., Jain, N., Sapre, N. S. In silico induction of missense mutation in NNRTI protein: Computational modelling and stability study of modelled proteins. J Math Chem. 62, 2776-2797 (2024).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。