Method Article
探索通过脂肪吞噬对脂滴分解代谢的调节
摘要
脂肪吞噬是一种选择性形式的自噬,涉及脂滴的降解。这个过程中的功能障碍与癌症的发展有关。然而,确切的机制尚不完全清楚。该协议描述了定量成像方法,以更好地了解自噬、脂质代谢和癌症进展之间的相互作用。
摘要
巨自噬,通常称为自噬,是一种高度保守的细胞过程,负责细胞成分的降解。这个过程在禁食、细胞应激、细胞器损伤、细胞损伤或细胞成分老化等情况下尤为突出。在自噬过程中,细胞质的一部分被封闭在称为自噬体的双膜囊泡内,然后与溶酶体融合。这种融合后,自噬体的内容物在溶酶体的促进下发生非选择性批量降解。然而,自噬也表现出选择性功能,靶向特定细胞器,包括线粒体、过氧化物酶体、溶酶体、细胞核和脂滴 (LD)。脂滴被磷脂单层包裹,该单层将中性脂质与细胞质分离,保护细胞免受过量甾醇和游离脂肪酸 (FFA) 的有害影响。自噬与各种疾病有关,包括神经退行性疾病、代谢紊乱和癌症。具体来说,脂肪吞噬 - 脂滴的自噬依赖性降解 - 在调节不同代谢状态下的细胞内 FFA 水平中起着至关重要的作用。该调节支持膜合成、信号分子形成和能量平衡等基本过程。因此,脂肪吞噬受损会增加细胞对死亡刺激的脆弱性,并导致癌症等疾病的发展。尽管其重要性,但控制癌细胞中脂肪吞噬调节的脂滴代谢的确切机制仍然知之甚少。本文旨在描述共聚焦成像采集和定量成像分析方案,这些方案能够研究与癌细胞代谢变化相关的脂肪吞噬。通过这些方案获得的结果可能揭示了自噬、脂质代谢和癌症进展之间错综复杂的相互作用。通过阐明这些机制,可能会出现对抗癌症和其他代谢相关疾病的新型治疗靶点。
引言
自噬是一个通用术语,用于描述细胞将其成分转运到溶酶体进行降解的分解代谢过程。迄今为止,已鉴定出三种类型的自噬:微自噬、巨自噬和伴侣介导的自噬 1,2,3。巨自噬,以下简称自噬,是调节细胞稳态的重要途径。这种平衡的破坏会导致病理状况的发展4。
自噬是一个复杂的过程,涉及多个步骤。第一步是自噬诱导,由各种刺激触发,例如生长因子(胰岛素和胰岛素样生长因子)的退出、病原性感染、细胞能量水平降低 (ATP)、细胞外或细胞内应激(例如缺氧、内质网 (ER) 应激、氧化应激)和营养缺乏(氨基酸、葡萄糖)5.第二步涉及吞噬泡的形成,其中膜分离从 ER、质膜和线粒体开始。从头形成涉及按顺序募集的胞质蛋白的保守机制6,例如 Ser/Thr 激酶 Unc-51 样激酶-1 复合体(酵母中的 ULK1:ATG1)、Beclin-1 和 VPS347。ULK1 复合体形成后,III 类磷脂酰肌醇 3-激酶 (PI3K) 复合体 I 被募集到分离膜 (IM) 中,该膜在货物的初始隔离中发挥作用8。此外,ULK1 复合物具有将 ATG9 募集到分离膜 (IM) 的能力,这是必不可少的步骤,因为 ATG9 囊泡被认为是促进 IM 扩增的膜载体9。
两种泛素样 (Ubl) 偶联系统对于扩增过程至关重要:微管相关蛋白 1 轻链 3 (LC3-I) 系统和 ATG12 系统10。在偶联之前,LC3 前体发生裂解。然后,胞质 LC3-I 与磷脂酰乙醇胺 (PE) 偶联,产生膜相关 LC3-PE (LC3-II),从而促进自噬体的形成11。在此过程中,货物必须内化到形成的双膜自噬体中。自噬可以内化随机靶标进行降解,或通过特异性自噬受体(如 p62/SQSTM112)捕获选择性货物。最后一步是形成的自噬体与溶酶体融合,导致自噬溶酶体形成。尽管自噬溶酶体形成的确切机制仍然难以捉摸,但膜栓系复合物、RAS 相关 GTP 结合蛋白和可溶性 N-乙基马来酰亚胺敏感因子附着蛋白受体 (SNARE) 蛋白参与了这一融合过程13。此外,微管细胞骨架系统对于将成熟的自噬体和溶酶体从随机起始位点运输到核周区域以形成自噬溶酶体至关重要 14,15,16。在自噬溶酶体中,随机或选择性隔离的货物被溶酶体蛋白酶蛋白水解降解17。
自噬过程在所有真核生物中都是保守的,并且在通过细胞质更新调节细胞内条件方面至关重要。它可以去除错误折叠或聚集的蛋白质,消除细胞内病原体,并清除受损的细胞器。据报道,几种细胞器,包括内质网、线粒体、过氧化物酶体、溶酶体、细胞核和 LDs,是自噬的靶标 16,18,19,20,21,22。LD 起源于 ER,是脂质和能量稳态的重要储存细胞器。它们独特的结构由中性脂质的疏水核心组成,该核心被嵌入特定蛋白质的磷脂单层包围。这些液滴可以通过膜接触位点与各种细胞器相互作用23。此外,自噬有助于回收初级资源以维持最佳细胞条件。例如,LD 的降解可通过脂肪酸β氧化24 产生 ATP。
自噬与多种疾病有关,包括神经退行性疾病、代谢紊乱和癌症17。自噬可以促进或抑制癌症中的肿瘤生长,具体取决于环境25,26。例如,Beclin 1 +/- 小鼠在肺、肝和乳腺组织等器官中表现出自发性淋巴瘤和癌的高发病率。相反,肠上皮细胞中自噬相关基因 Atg7 的缺失会减弱由结直肠癌中原发性肿瘤抑制因子大肠腺瘤性息肉病 (APC) 的缺失驱动的肿瘤生长27,28。因此,自噬相关基因的缺失会对肿瘤生长产生相反的影响。
癌细胞必须产生能量来维持其生长、分裂和存活29。它们对脂质具有很高的亲和力,脂质用于结构成分的生物合成和能量产生30。癌细胞可以根据环境条件调整其新陈代谢。例如,当宫颈癌来源的 HeLa 细胞中的糖酵解受到抑制时,氧化磷酸化增加以获得生存所需的 ATP31。细胞中的脂质不以非酯化 FFA 的形式存在,因为它们在高浓度下具有潜在的细胞毒性。相反,细胞将 FFA 和胆固醇储存为中性、惰性生物分子,例如 LDs32 中的甾醇酯和甘油三酯。因此,脂肪吞噬可以通过提供 FFA 来产生能量来促进癌症代谢,这是癌症研究的一个新兴领域。然而,上调癌细胞中线粒体 FA 氧化的途径仍然知之甚少。FFA 的摄取和积累已被证明可以增强不同癌症类型的侵袭性 33,34,35。脂质代谢重编程是癌症代谢重编程的标志,作为管理肿瘤微环境中不良生理情景的适应性反应起着关键作用36,37。事实上,在许多人类癌症中观察到 LDs 积累,包括肺癌、乳腺癌和前列腺癌,并且与侵袭性和不良临床预后有关 38,39,40。
鉴于自噬和 LDs 在癌症代谢中的相关性以及知之甚少的机制,必须建立研究它们对癌症发展贡献的方案。本研究描述了一种通过共聚焦成像采集和定量成像分析方案来评估脂肪吞噬的方案,以研究癌细胞中的脂质代谢变化。
研究方案
本研究是使用上皮腺癌 HeLa 细胞 (CCL2, ATCC) 进行的。该方案侧重于研究活细胞中脂肪吞噬诱导过程中的脂滴 (LD),以量化表达野生型 (p62/SQSTM1-S182S) 和自噬受体 p62/SQSTM1 的两个位点特异性突变体的细胞中 LD 数变化和 LD-自噬体相互作用的时间过程16。磷酸化缺陷型 (p62/SQSTM1-S182A) 的表达增加了 LD 的数量,而磷酸化模拟型 (p62/SQSTM1-S182E) 的表达减少了 LD的数量 16。首先,描述了一种使用共聚焦显微镜分析活细胞中 LD 的方法。然后,使用机器人共聚焦显微镜解释无偏、全自动图像采集和分析的方案。材料 表中提供了本研究中使用的试剂和设备的详细信息。
1. 共聚焦活细胞成像
- 细胞培养
- 在 37 °C 和 5% CO2 的 Dulbecco 改良 Eagle 培养基 (DMEM) 中培养细胞,补充有 10% v/v 胎牛血清 (FBS)、1,000 U/mL 青霉素、100 μg/mL 链霉素和两性霉素 B。维持细胞直到它们达到不超过 80% 的汇合度。
- 用 1 mL 1x 磷酸盐缓冲盐水洗涤细胞,使用 0.25% v/v 胰蛋白酶/EDTA 收集,并在室温下以 100 x g 离心 5 分钟。
- 使用移液管弃去上清液。
- 将细胞重悬于含 10% v/v FBS 的 DMEM 培养基中,并将(0.05 x 106 个细胞/孔)接种到 35 mm 玻璃底培养皿中。
- 使用 TransIT-LT1 转染或 Lipofectamine 2000 试剂转染细胞。使用每种 p62/SQSTM1-mcherry 磷酸化变体和野生型 p62/SQSTM1-mcherry16 的 1 μg 质粒 DNA。
- 在进行后续处理之前,将细胞在 37 °C 和 5% CO2 下孵育 48 小时。
注:转染后 16-24 小时,p62/SQSTM1 形成大缩合物,而不是离散的 p62/SQSTM1 自噬体。这些大的冷凝物在 48 小时标记处大部分消散,从而可以更精确地观察自噬体。
- 细胞器标记
- 在玻璃底培养皿中用1x PBS(预热至37°C)洗涤细胞两次。
- 将细胞与稀释至 0.5 μM 的 BODIPY 493/503 在补充有 10 mM HEPES 的 DMEM 培养基中孵育,并在 37 °C、5% CO2 下保持 30 分钟。BODIPY 405 和 BODIYPY 633 也可以使用相同的结果。
- 检查 p62/SQSTM1 变体的表达是否诱导 LDs 数量和总荧光强度的变化。
- 用 1x PBS(室温)洗涤细胞,并将它们维持在不含红酚的 DMEM 中,补充有 10 mM ES 合格的 HEPES 缓冲液用于活细胞成像。
注意:BODIPY 493/503 发出亮绿色荧光,便于双重荧光标记。然而,在某些条件下,例如重复激发,它会发出红色荧光,当与红色标记物结合使用时,可能会导致错误的解释(错误的共定位)。有关 550 nm 激发荧光基团的更多信息,请参阅 Ohsaki 等人41 的文章。
- 活细胞成像中的共聚焦显微镜图像采集
注:LD 的图像是使用具有 63 倍油浸物镜 (NA 1.4) 的共聚焦显微镜获取的。使用兼容的图像采集软件来捕获图像。使用自动温度控制系统将温度保持在 37 °C。
注:脂滴 (LD) 的图像是使用具有 63 倍油浸物镜 (NA 1.4) 的共聚焦显微镜获取的。使用兼容的图像采集软件来捕获图像。使用自动温度控制系统将温度保持在 37 °C。- 将多线氩气激光器调整为 10% 工作功率,488 nm 激光线的工作效力为 1%-2%,总激光功率为 0.1%-0.2%。
- 为了最大限度地减少细胞损伤和探针光漂白,将 568 nm 激光调整至 3%-5% 效力。
- 将软件中的图像采集设置调整为 1024 x 1024 像素的分辨率。该系统使用以 600 Hz 运行频率的混合探测器,平均为 2。
- 在 478-494 nm(LD 的绿色发射)和 600-625 nm 波长(mCherry 的红色发射)之间调整光谱范围。
- 对于 488 nm 波长,将针孔大小调整为 1 艾里单位 (AU)。
- 将细胞与 8-Br-cAMP (100 mM) 一起孵育,以激活 PKA 激酶并增加 LDs 降解16。
- 以 5 分钟的间隔捕获荧光图像,37 °C 持续 60 分钟。
- 图像分析
- 使用 ImageJ 打开包含单个实时图像序列的堆栈文件。使用 ROI 管理器进行手动单元格分割。保存区域以供下一步使用。
- 打开堆栈文件并调用每个图像的已保存区域。调整 LDs 通道阈值。
- 通过在 ROI 管理器上选择 multi-measure 来测量总荧光强度。
注:在自噬过程中,总荧光强度和 LD 的数量会发生显著变化。此外,在自噬诱导过程中,LD 和溶酶体之间的相互作用增加。
- 接触和共定位分析
- 在 37 °C 下以 1 秒的间隔进行多光谱捕获 5 分钟。
- 将多线氩气激光器设置为 10% 效力,488 nm 激光线的工作功率为 0.1%-0.5%,以最大限度地减少细胞损伤和探针光漂白。将 568 nm 激光调整至 3%-5% 效力。
- 点击 ComDet V:ComDet V 0.5.3 <插件<检测粒子。定义点必须被视为共定位的最小距离。
- 为两个通道分别选择定义粒子的参数,例如其大小(mm 或像素单位)和阈值。将提供一个摘要,其中包含每次每个通道中的粒子数和相应的共定位百分比,可以导出到 Excel(请参阅 补充文件 1)。
注:建议将 ComDet V 插件用于椭圆形的细胞器,例如脂滴、内体、过氧化物酶体或溶酶体。不建议将其与线粒体等细胞器共定位。“ComDet V”插件(ImageJ FIJI 分布)允许通过粒子分析来分析时间共定位。要在斐济安装它,请复制 URL http://sites.imagej.net/Ekatrukha/,转到“帮助”、“更新”,选择“管理更新站点 - 添加更新站点 ”并粘贴 URL。应用更改;建议使用两个通道。如果有更多频道,请将它们分开,然后使用 Merge 重新加入。
- LD 的动态运动
- 定义微珠和细胞的感兴趣区域 (ROI) 并保存它们。转到 plugins 并选择 Tracking 和 TrackMate。
- 打开 TrackMate。该插件的第一个窗口提供有关捕获图像的时间间隔和图像分辨率的信息。要确认时间间隔而不进行更改,请单击 NEXT。
- 选择 LoG 检测器 ,然后选择 下一步。在 LoG 检测器配置中,根据颗粒的直径 (0.8-1.0 微米) 过滤颗粒,并设置适当的阈值。执行 预览 以确认正确的参数,然后单击 下一步。
- 设置初始 Threshold (阈值),这是一个质量阈值,用于限制要分析的点的数量。当跟随飞机上的许多点时,这是必需的,这很有挑战性。不要使用过滤器;按 NEXT 而不更改参数。
- 选择视图:选择 HyperStack Displayer 并单击 NEXT。
- 为平面中速度恒定的粒子选择 Linear motion LAP 跟踪器 。
- 定义开始新轨道时两个点之间的最大距离。根据图像,建议在 0.5 -1 微米之间。
- 设置与候选点的预测位置的最大距离以进行分析。
- 将 Max Frame Gap (最大帧间隙) 设置为跟随可能从焦平面上消失的点的最长时间。我们建议 2 个时间点。
- 选择 Display Options (显示选项)。摘要表(包括最小速度、最大速度、平均速度、中速和轨道位移)将导出到 Excel(请参阅 补充文件 2)。
- 选择 Plot Features。要直观地表示结果,请转到 Tracks (轨迹)。选择要在 X 轴上表示的度量(速度和位移),并将载荷点放置在 Y 轴上。
- 选择一个 Action 以保存显示点移动的视频。这允许可视化显示显着变化的点。
注:增强自噬可优化细胞通量并减少 LD 值,突出其在代谢调节和细胞稳态中的关键作用16。提到的插件可用于 ImageJ 软件(FIJI 发行版)。也可以使用等效的软件。对于所有荧光强度测量,都使用了积分荧光密度(ImageJ 中的“RawIntDen”),考虑到面积,它考虑了每个图像像素的总荧光。
2. 固定细胞中的全自动共聚焦图像采集和图像分析
注意:该方法允许评估多个条件并为每个条件开发一式三份测量,提高平均测量值的置信度,并能够确定标准偏差或标准误差以进行实验之间的统计区分。该方法的工作流程如图 1 所示的流程图所示。
- 细胞培养
- 在 37 °C 和 5% CO2 的 Dulbecco 改良 Eagle 培养基 (DMEM) 中培养细胞,补充有 10% v/v 胎牛血清 (FBS)、1,000 U/mL 青霉素、100 μg/mL 链霉素和两性霉素 B。
- 用 1 mL 的 1x 磷酸盐缓冲盐水 (PBS) 洗涤细胞,使用 0.25% v/v 胰蛋白酶/EDTA 收获。
- 将细胞收集在 15 mL 离心管中。
- 在室温下以 100 x g 离心细胞 5 分钟。
- 用移液管小心吸出上清液并保留细胞沉淀。
- 将细胞沉淀重悬于含 10% v/v FBS 的 DMEM 培养基中,并接种到光学底部 96 孔板(0.05 x 106 个细胞/孔)中。
注意:在进行后续处理之前,将细胞在 37 °C 和 5% CO2 下孵育 24 小时。
- 通过血清饥饿诱导自噬
- 取出细胞培养基,将细胞在无血清的 DMEM 培养基中孵育,以去除血清并诱导自噬 6 小时。
- 使用巴弗洛霉素 A1 200 nM 抑制 LD 脂肪吞噬。二酰基甘油酰基转移酶-1 (DGAT1) 抑制剂 T863 (50 μM) 抑制 LD 生物发生。使用肉碱棕榈酰转移酶 I (CPT1) 抑制剂 Etomoxir (100 μM) 抑制 FFA 转化为酰基肉碱。此外,使用 ATGLstatin (10 μM) 可以抑制脂肪分解。
- 细胞固定
- 用冰冷的 PBS-CM(200 μL/孔)洗涤细胞 3 次。
- 用在补充有 0.1 mM CaCl2 和 1 mM MgCl2 (PBS-CM) 的 PBS 中稀释的 4% 多聚甲醛在室温下固定细胞 15 分钟16。
- 用 PBS-CM (200 μL/孔) 洗涤细胞 3 次。
- 在室温下用 PBS-CM 中的 0.2% Triton X-100 透化 15 分钟。
- 用 PBS-CM 洗涤细胞 3 次。
- 细胞器标记
- 将细胞与稀释为 0.5 μM 的 BODIPY 493/503 和 PBS-CM 中的 DAPI (125 mg/mL) 在 37 °C 下孵育 30 分钟。
- 用 PBS-CM 洗涤 3 次。
- 将固定和染色的细胞保存在每孔 200 μL PBS 中,直到图像采集。
注意:96 孔板可以在 4 °C 下储存数周,避光和脱水。不需要使用常用的抗淬灭封片剂进行封片。
- 自动共聚焦显微镜(固定细胞)
- 进行细胞核染色以进行自动图像分割16.
注意:图像采集使用带有 40 倍 (NA 1.1) 水浸物镜的转盘显微镜。使用 305 nm LED 照明(例如:355-385 nm;Em:430-500 nm),BODIPY 493/503 采集使用 488 nm LED 照明(例如:460-490 nm;Em:550-550 nm)。
- 进行细胞核染色以进行自动图像分割16.
- 图像分析
注:图像分析16 使用具有专用 LD 分析模块的兼容软件进行。本研究中使用的软件(参见 材料表)具有用于图像分割和定量的现成解决方案(RMS 脂质液滴分析)算法组合。- 通过选择 DAPI 通道来识别细胞核,并根据信号和背景调整阈值。
- 识别细胞质。根据 LDs 荧光通道选择合适的方法。
- 通过调整参数来识别斑点:半径 (1-1.5 μm)、对比度 (0.2-0.25)、未校正的斑点到区域强度 (0.4-0.6)、距离 (0.3-0.4 μm)、斑峰半径 (0.2-0.25 μm)。
- 计算形态学特性:选择 群体 LDs、Region Spot 和 标准 方法,然后选择 Area。
- 计算强度属性:选择 LDs 通道、 群体 LDs、Region Spot 和 标准 方法。
- 计算属性:选择“所有细胞群”,按相关群体 LD 选择方法,然后选择 LD 的数量、面积和强度(平均值和总和;总和是积分荧光密度/强度)。这将产生与细胞相关的 LD 测量值。
- 通过选择标准输出、对象计数、以 SD 平均值表示的每个单元的 LDs 数±、每个区域的平均 LDs、每个区域的总 LDs、每个区域的 LD 分数和总 LDs 强度来定义结果。
结果
共聚焦活细胞成像
LD 是动态的,并与 p62/SQSTM1 阳性自噬体瞬时相互作用。当诱导脂肪吞噬时,这些相互作用会减少 LD 的数量及其总荧光强度。该方案使用自噬受体 p62/SQSTM1 的磷酸化突变版本来检查这些影响16。
LD 的数量和荧光强度受脂肪吞噬调节,取决于 p62/SQSTM1 的表达变体。p62/SQSTM1-S182A 的表达增加了脂滴的数量及其荧光强度,而 p62/SQSTM1-S182E 的表达降低了脂滴的数量及其总荧光强度。这种机制是通过 p62/SQSTM1-S182A 抑制脂肪吞噬或 p62/SQSTM1-S182E 激活其发生的,从而导致 LDs 降解的抑制或激活。定量变化包括表达 48 小时后 LDs 增加约 20%-25%,总荧光强度增加 30%-35%。相反,48 小时后 LDs 降低 50%-60%,荧光强度降低 60%(图 2)。
与 p62/SQSTM1-S182 野生型对照相比,PKA 激活还降低了 LDs 的数量和总荧光强度,因为增强脂肪吞噬会增加 LDs 降解。定量变化显示,在脂肪吞噬诱导的 PKA 激活 30 分钟后,总荧光强度降低 20%-25%。p62/SQSTM1-S182A 的表达通过阻断脂肪吞噬导致 LDs 减少,PKA 激活 30 分钟后 LDs 的总荧光强度降低不到 10%(图 3)。
当 p62/SQSTM1-S182E 表达诱导脂肪吞噬时,与对照细胞相比,LDs 和自噬体之间的瞬时接触增加,并且 LDs 速度增加。相比之下,表达 p62/SQSTM1-S182A 的细胞显示 LDs 和自噬体之间的接触减少,LDs 速度降低。当脂肪吞噬被阻断时,LD/自噬体接触减少 40%-50%,p62/SQSTM1-S182A 表达未观察到 LD 速度变化。当诱导脂肪吞噬时,LDs/自噬体接触增加 35%-50%,p62/SQSTM1-S182E 表达时 LDs 速度增加 100%-120%(图 4)。
在固定池中完成自动共聚焦图像采集和图像分析
LDs 参数受脂肪吞噬介导的 LDs 代谢调节。通过血清剥夺诱导脂肪吞噬将减少 LDs 的数量、单位面积的 LDs、单位面积的总 LDs、单位面积的分数和总 LDs 强度。巴弗洛霉素会阻断脂肪吞噬引起的变化。依托莫昔将增加 LDs 的数量、每个区域的 LDs 数量、每个区域的 LDs 总数、每个区域的 LDs 分数以及 LDs 的总强度。T863 将减少 LDs 数量、每个区域的 LDs、每个区域的 LDs 总数、每个区域的 LDs 分数和总 LDs 强度。
血清剥夺会增加脂肪吞噬,需要溶酶体活性来增加 FFA 的可用性。CPT1 将 FFA 转移到线粒体。T863 抑制 DGAT1 阻断 LD 形成。LDs 数量、单位面积的 LDs、单位面积的总 LDs、单位面积的 LDs 分数和总 LDs 强度变化将取决于细胞类型,范围从 20% 到 60% 不等(图 1)。
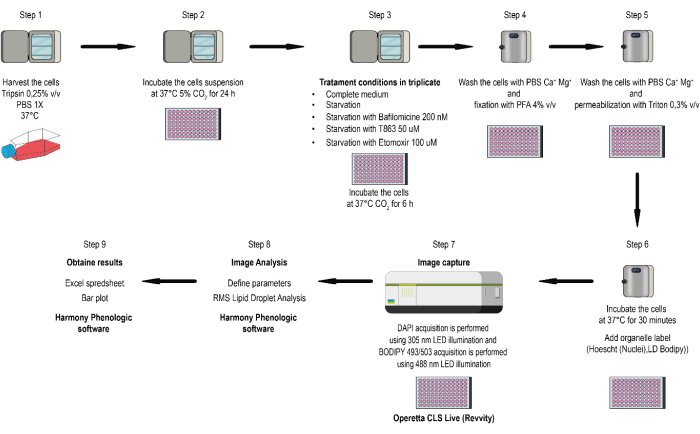
图 1:研究流程图。 将细胞在37°C和5%CO2 的DMEM,10%v / v FBS,1,000U / mL青霉素,100μg/ mL链霉素和两性霉素B中培养。使用0.25%v / v胰蛋白酶/ EDTA收获并以100× g 离心5分钟。然后吸出上清液。将细胞(0.05 x 106 个细胞/孔)悬浮在含有10%v / v FBS的DMEM培养基中,并接种到光学底部96孔板中。使用配备 40 倍 (N.A 1.1) 水浸物镜的活体转盘显微镜进行图像采集。使用 305 nm LED 照明(例如:355-385 nm;Em:430-500 nm),BODIPY 493/503 采集使用 488 nm LED 照明(例如:460-490 nm;Em:550-550 nm)。图像分析使用现象学软件与 RMS 脂滴分析算法组合进行图像分割和定量。 请单击此处查看此图的较大版本。

图 2:脂肪吞噬激活/抑制开关 LD 的数量及其总荧光。 转染细胞表达野生型或噬脂缺陷型 p62/SQSTM1-mcherry 变体 (S182A),并对 LDs 进行染色(如图所示)。在 PKA 激活期间,通过 8-Br-cAMP (100 mM) 每隔 5 分钟获取活细胞图像,并测量总荧光强度。PKA 激活降低了 LD 强度。自噬缺陷型 p62/SQSTM1 版本的表达降低了 PKA 反应。PKA 的作用通过使用 PKA 抑制剂 PKI(50 mM,肉豆蔻酰化 PKI 肽)是可逆的。数据是 SEM ±平均值。该图是从 Tapia 等人 16 修改而来的。 请单击此处查看此图的较大版本。
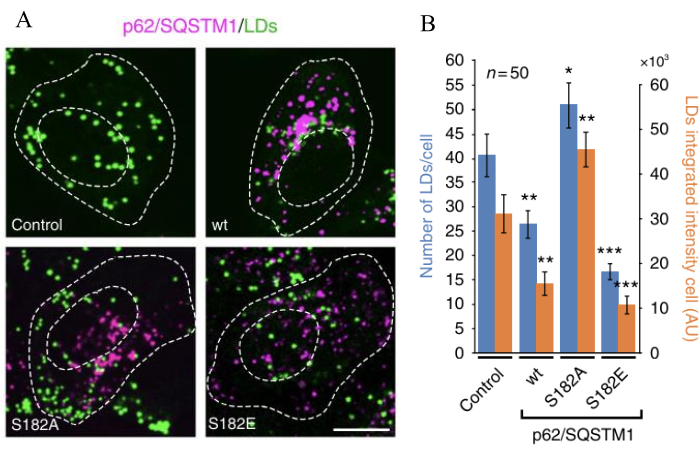
图 3:脂肪吞噬激活或抑制开关 LD 的数量及其总荧光。 (A) 转染细胞以表达指定的 p62/SQSTM1-mcherry 变体(品红色),并对 LD 进行染色和定量(绿色)。比例尺:10 μm。(B) 在对照(未转染细胞)和表达 p62/SQSTM1-cherry-wt(野生型)或指定的 S182A 和 S182E 磷酸化变体版本的 p62/SQSTM1-cherry 中测量 LD 的数量和荧光强度。数据是 SEM ±平均值。*p < 0.05;**p < 0.01;p < 0.001 (学生 t 检验)。与对照细胞进行比较,进行所有 t 检验 。该图是从 Tapia 等人 16 修改而来的。 请单击此处查看此图的较大版本。
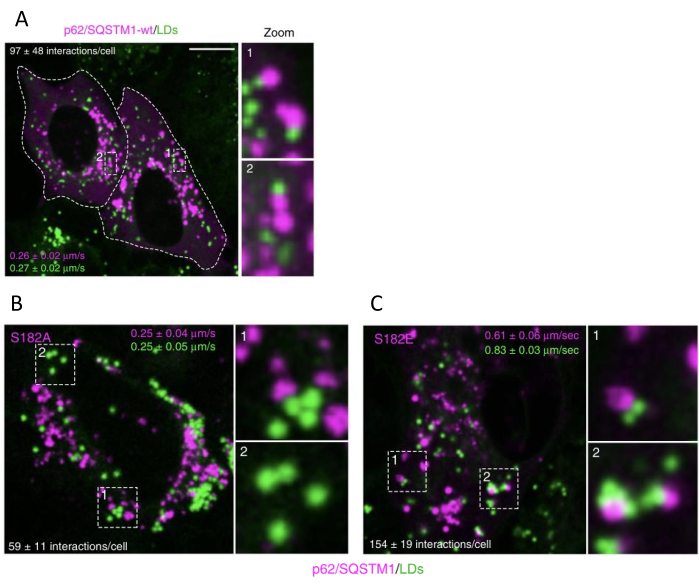
图 4:脂肪吞噬激活或抑制改变了 LDs 速度和自噬体相互作用的数量。 (A) 转染 p62/SQSTM1-cherry-wt(野生型)或指定的 S182A (B) 和 S182E (C) 磷酸化变体版本,然后进行 LD 染色。获取活细胞图像,并测量 LDs 和 p62/SQSTM1-cherry-wt 点相互作用的数量和移动速度。比例尺:10 μm。数据是 SEM ±平均值。该图是从 Tapia 等人 16 修改而来的。 请单击此处查看此图的较大版本。
补充文件 1:交互电子表格。 瞬时转染细胞以表达 p62/SQSTM1-mcherry-wt (野生型),然后对脂滴 (LDs) 进行染色。获得活细胞图像,并量化 LDs 与 p62/SQSTM1-mcherry-wt 点之间的相互作用次数。 请点击此处下载此文件。
补充文件 2:排水量和速度电子表格。 瞬时转染细胞以表达 p62/SQSTM1-mcherry-wt (野生型),随后进行脂滴 (LDs) 染色。然后捕获活细胞图像以量化 LDs 和 p62/SQSTM1-mcherry-wt 点之间的相互作用次数,以及测量运动速度和位移。 请点击此处下载此文件。
讨论
定量成像技术,如共聚焦显微镜和图像分析方案,为脂肪吞噬过程中 LD 的动力学提供了有价值的见解 16,42,43。这些技术能够实时可视化和定量 LD,从而可以分析它们的数量、大小以及与其他细胞器的相互作用16。然而,该方案中最关键的步骤之一是正确的 LDs 标记,为了实现这一目标,需要使用具有检测范围的液相 LDs 标志物,这些标志物允许以高精度和特异性量化 LDs 的微小差异。基于 SPLICS 报告基因44 开发了一种更定量的细胞器-细胞器接触方法。
BODIPY 493/503 标记已被广泛使用,但存在实验局限性,例如低 FAs 浓度的检测受限和荧光发射的变化可能导致对结果的误解41。其他荧光染料,如尼罗红,由于其荧光发射范围较宽,为 550-750 nm,因此难以用于双重标记45。另一方面,BODIPY 558/568 C12(红色 C12)可以很好地替代检测活细胞和固定细胞中 LD 中的低 FA 含量46。然而,根据细胞类型,它必须以适当的浓度使用,因为它也可以标记其他细胞器(如内质网和线粒体)中包含的 FA。
本文详细介绍了自动图像采集和分析方案,简化了活细胞和固定细胞中脂肪吞噬的研究。这种方法考虑了以前分子方法的缺陷并解释了其局限性,通常与选择 LDs 标记物有关47,48。通过该协议,除了使用适当的图像采集设备和软件外,还可以对 LD 及其与溶酶体的相互作用进行精确和实时的定量。这种方法提供了可靠、无偏倚和稳健的数据,对于理解脂肪噬对癌细胞模型中 LDs 代谢的作用至关重要。
总之,先进的成像技术与自动图像采集和定量分析方法相结合,简化和加快了数据收集,同时提供了可靠的结果。从这些数据中获得的见解增强了我们对细胞器生物学和脂质代谢之间相互作用的理解。未来的研究应进一步阐明癌细胞中脂肪吞噬调节的分子机制,并探索其在癌症治疗中的治疗潜力。
披露声明
作者没有需要披露的利益冲突。
致谢
Operetta 机器人共聚焦显微镜由 Fondo de Equipamiento Mediano (FONDEQUIP) 第 EQM220072 期资助。C.L. 得到了 Vicerrectoria de Investigación y Doctorados (VRID) 和圣塞巴斯蒂安大学博士奖学金的支持。C.S. 得到了 Agencia Nacional de Investigación y Desarrollo (ANID) 奖学金的支持。D.T. 和 J.C. 得到了 Fondo Nacional de Desarrollo Científico y Tecnológico (FONDECYT) N°1221374 资助。
材料
| Name | Company | Catalog Number | Comments |
| 35 mm glass-bottom dishes | MatTek | P35G-1.5-14-C | |
| Bafilomycin A1 | Tocris | 1334 | 200 nM |
| BODIPY 493/503 | Invitrogen | D3922 | 0.5 mM |
| CaCl2 | Merck | 102378 | 0.1 mM |
| ComDet V Plugin | ImageJ | ImageJ FIJI | |
| DAPI | Invitrogen | D1306 | 125 mg/mL |
| Dulbecco’s Modified Eagle’s Medium (DMEM) | Gibco | 12800017 | |
| ES-qualified HEPES buffer | Cytiva HyClone AdvanceSTEM | SH3085101 | 10 mM |
| Etomoxir | SigmaAldrich | E1905 | 100 mM |
| Fetal Bovine Serum | Cytiva HyClone AdvanceSTEM | SH3039603 | 10% v/v |
| Forma Series II Water-Jacketed CO2 Incubator | Thermo Scientific | 3111 | 37 °C, 5% CO2 |
| Harmony Phenologic software | Revvity | image analysis software | |
| HeLa cells | ATCC | CCL-2 | Maintain cells at a low passage number, ideally between 8 and 10, to ensure optimal cellular characteristics. |
| HEPES | Merck | 110110 | 10 mM |
| High-speed clinical centrifuge | DLAB | DM0412 | |
| Immersion Oil | Leica | 11513859 | |
| MgCl2 | Merck | 814733 | 1 mM |
| Operetta CLS Live spinning-disk microscope | Revvity | HH16000020 | |
| Optical bottom 96-well plates | Thermo Scientific | 165305 | |
| Paraformaldehyde | Electron Microscopy Sciences | 157-8 | 4%v/v |
| penicillin/streptomycin/Amphotericin B | Biological Industries | 030331b | (1000 µ/mL, 100 mg/mL, 100 mg/mL) |
| Phosphate-buffered saline (PBS) | Sartorius | 020235A | 1x |
| Red-phenol free DMEM | Gibco | 31053028 | |
| T863 | Merck | SML0539 | 50 mM |
| TCS SP8 Leica confocal microscope | Leica Microsystems | ||
| TransIT-LT1 Transfection Reagent | Mirus | MIR 2304 | |
| Triton X-100 | Merck | T9284 | 0.20% |
| Trypsin/EDTA | Gibco | 252000056 | 0.25% v/v |
| UNO-TEMP controller | Okolab | OK-H401-T-CONTROLLER | 37 °C |
参考文献
- Yamamoto, H., Matsui, T. Molecular mechanisms of macroautophagy, microautophagy, and chaperone-mediated autophagy. J Nippon Med Sch. 91 (1), 2-9 (2024).
- Mejlvang, J., et al. Starvation induces rapid degradation of selective autophagy receptors by endosomal microautophagy. J Cell Biol. 217 (10), 3640-3655 (2018).
- Kaushik, S., Cuervo, A. M. The coming of age of chaperone-mediated autophagy. Nat Rev Mol Cell Biol. 19 (6), 365-381 (2018).
- Galluzzi, L., Pietrocola, F., Levine, B., Kroemer, G. Metabolic control of autophagy. Cell. 159 (6), 1263-1276 (2014).
- He, C., Klionsky, D. J. Regulation mechanisms and signaling pathways of autophagy. Annu Rev Genet. 43, 67-93 (2009).
- Suzuki, K., Ohsumi, Y. Current knowledge of the pre-autophagosomal structure (pas). FEBS Lett. 584 (7), 1280-1286 (2010).
- Neufeld, T. P. Contribution of ATG1-dependent autophagy to tor-mediated cell growth and survival. Autophagy. 3 (5), 477-479 (2007).
- Mizushima, N., Yoshimori, T., Ohsumi, Y. The role of ATG proteins in autophagosome formation. Annu Rev Cell Dev Biol. 27, 107-132 (2011).
- Mari, M., et al. An ATG9-containing compartment that functions in the early steps of autophagosome biogenesis. J Cell Biol. 190 (6), 1005-1022 (2010).
- Itakura, E., Mizushima, N. Characterization of autophagosome formation site by a hierarchical analysis of mammalian ATG proteins. Autophagy. 6 (6), 764-776 (2010).
- Kabeya, Y., et al. LC3, GABARAP AND GATE16 localize to autophagosomal membrane depending on form-ii formation. J Cell Sci. 117 (Pt 13), 2805-2812 (2004).
- Ahmad, R., et al. P62/SQSTM1 binds with claudin-2 to target for selective autophagy in stressed intestinal epithelium. Commun Biol. 6 (1), 740 (2023).
- Bento, C. F., et al. Mammalian autophagy: How does it work. Annu Rev Biochem. 85, 685-713 (2016).
- Jahreiss, L., Menzies, F. M., Rubinsztein, D. C. The itinerary of autophagosomes: From peripheral formation to kiss-and-run fusion with lysosomes. Traffic. 9 (4), 574-587 (2008).
- Pu, J., Guardia, C. M., Keren-Kaplan, T., Bonifacino, J. S. Mechanisms and functions of lysosome positioning. J Cell Sci. 129 (23), 4329-4339 (2016).
- Tapia, D., et al. KDEL receptor regulates secretion by lysosome relocation- and autophagy-dependent modulation of lipid-droplet turnover. Nat Commun. 10 (1), 735 (2019).
- Ichimiya, T., et al. Autophagy and autophagy-related diseases: A review. Int J Mol Sci. 21 (23), 8974 (2020).
- Ciechanover, A. Proteolysis: From the lysosome to ubiquitin and the proteasome. Nat Rev Mol Cell Biol. 6 (1), 79-87 (2005).
- Khaminets, A., et al. Regulation of endoplasmic reticulum turnover by selective autophagy. Nature. 522 (7556), 354-358 (2015).
- Ashrafi, G., Schwarz, T. L. The pathways of mitophagy for quality control and clearance of mitochondria. Cell Death Differ. 20 (1), 31-42 (2013).
- Koerver, L., et al. The ubiquitin-conjugating enzyme ube2ql1 coordinates lysophagy in response to endolysosomal damage. EMBO Rep. 20 (10), e48014 (2019).
- Deosaran, E., et al. NBR1 acts as an autophagy receptor for peroxisomes. J Cell Sci. 126 (Pt 4), 939-952 (2013).
- Olzmann, J. A., Carvalho, P. Dynamics and functions of lipid droplets. Nat Rev Mol Cell Biol. 20 (3), 137-155 (2019).
- Xu, C., Fan, J. Links between autophagy and lipid droplet dynamics. J Exp Bot. 73 (9), 2848-2858 (2022).
- White, E. The role for autophagy in cancer. J Clin Invest. 125 (1), 42-46 (2015).
- Amaravadi, R. K., Kimmelman, A. C., Debnath, J. Targeting autophagy in cancer: Recent advances and future directions. Cancer Discov. 9 (9), 1167-1181 (2019).
- Levy, J., et al. Intestinal inhibition of ATG7 prevents tumour initiation through a microbiome-influenced immune response and suppresses tumour growth. Nat Cell Biol. 17 (8), 1062-1073 (2015).
- Trentesaux, C., et al. Essential role for autophagy protein ATG7 in the maintenance of intestinal stem cell integrity. Proc Natl Acad Sci U S A. 117 (20), 11136-11146 (2020).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (review). Oncol Lett. 4 (6), 1151-1157 (2012).
- Martin-Perez, M., Urdiroz-Urricelqui, U., Bigas, C., Benitah, S. A. The role of lipids in cancer progression and metastasis. Cell Metab. 34 (11), 1675-1699 (2022).
- Shiratori, R., et al. Glycolytic suppression dramatically changes the intracellular metabolic profile of multiple cancer cell lines in a mitochondrial metabolism-dependent manner. Sci Rep. 9 (1), 18699 (2019).
- Danielli, M., Perne, L., Jarc Jovicic, E., Petan, T. Lipid droplets and polyunsaturated fatty acid trafficking: Balancing life and death. Front Cell Dev Biol. 11, 1104725 (2023).
- Butler, L. M., et al. Lipids and cancer: Emerging roles in pathogenesis, diagnosis and therapeutic intervention. Adv Drug Deliv Rev. 159, 245-293 (2020).
- Nagarajan, S. R., Butler, L. M., Hoy, A. J. The diversity and breadth of cancer cell fatty acid metabolism. Cancer Metab. 9 (1), 2 (2021).
- Vasseur, S., Guillaumond, F. Lipids in cancer: A global view of the contribution of lipid pathways to metastatic formation and treatment resistance. Oncogenesis. 11 (1), 46 (2022).
- Yang, K., et al. The role of lipid metabolic reprogramming in tumor microenvironment. Theranostics. 13 (6), 1774-1808 (2023).
- Tan, Y., et al. Metabolic reprogramming from glycolysis to fatty acid uptake and beta-oxidation in platinum-resistant cancer cells. Nat Commun. 13 (1), 4554 (2022).
- Safi, R., Menendez, P., Pol, A. Lipid droplets provide metabolic flexibility for cancer progression. FEBS Lett. 598 (10), 1301-1327 (2024).
- Iwahashi, N., et al. Lipid droplet accumulation independently predicts poor clinical prognosis in high-grade serous ovarian carcinoma. Cancers (Basel). 13 (20), 5251 (2021).
- Luo, W., et al. Adding fuel to the fire: The lipid droplet and its associated proteins in cancer progression. Int J Biol Sci. 18 (16), 6020-6034 (2022).
- Ohsaki, Y., Shinohara, Y., Suzuki, M., Fujimoto, T. A pitfall in using bodipy dyes to label lipid droplets for fluorescence microscopy. Histochem Cell Biol. 133 (4), 477-480 (2010).
- Rambold, A. S., Cohen, S., Lippincott-Schwartz, J. Fatty acid trafficking in starved cells: Regulation by lipid droplet lipolysis, autophagy, and mitochondrial fusion dynamics. Dev Cell. 32 (6), 678-692 (2015).
- Nguyen, T. B., Olzmann, J. A. Lipid droplets and lipotoxicity during autophagy. Autophagy. 13 (11), 2002-2003 (2017).
- Giamogante, F., et al. A SPLICS reporter reveals-synuclein regulation of lysosome-mitochondria contacts which affects TFEB nuclear translocation. Nat Commun. 15 (1), 1516 (2024).
- Greenspan, P., Mayer, E. P., Fowler, S. D. Nile red: A selective fluorescent stain for intracellular lipid droplets. J Cell Biol. 100 (3), 965-973 (1985).
- Nguyen, T. B., et al. DGAT1-dependent lipid droplet biogenesis protects mitochondrial function during starvation-induced autophagy. Dev Cell. 42 (1), 9-21.e5 (2017).
- Murugan, S., Amaravadi, R. K. Methods for studying autophagy within the tumor microenvironment. Adv Exp Med Biol. 899, 145-166 (2016).
- Mallela, S. K., et al. Detection and quantification of lipid droplets in differentiated human podocytes. Methods Mol Biol. 1996, 199-206 (1996).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。