Method Article
跟踪实验进化人群中结构变异的动态
摘要
我们开发了一种具有成本效益的方法来跟踪非单核苷酸多态性等位基因动力学,该方法可以轻松适应实验进化冷冻档案。将三重PCR技术与自动平行毛细管电泳相结合,以量化实验进化过程中插入等位基因的相对频率。
摘要
结构变异(SV)(即缺失、插入、重复和倒置)现在已知在表型变异中起重要作用,因此在疾病确定或适应新环境等过程中起重要作用。然而,单核苷酸变异比SV受到更多的关注,可能是因为它们更容易检测,并且它们的表型效应更容易预测。短读长深度测序技术的发展极大地改善了SV的检测,但从汇集测序(poolseq)数据中量化其频率在技术上仍然复杂且昂贵。
在这里,我们提出了一种相当简单且便宜的方法,该方法允许研究人员跟踪SV等位基因频率的动态。作为应用的一个例子,我们跟踪细菌实验进化群体中插入序列(IS)的频率。该方法基于围绕结构变异边界的引物三重体设计,使得通过复制野生型(WT)和衍生等位基因产生的扩增子在大小上相差至少5%,并且它们的扩增效率相似。然后通过平行毛细管电泳确定每个扩增子的数量,并将其归一化为校准曲线。该方法可以很容易地扩展到其他结构变异(缺失、重复和倒置)的频率定量,以及自然人群(包括患者体内病原体群体)的池-序列方法。
引言
结构变异(SV)是基因组序列的改变,通常影响50 bp或更多。所描述的 SV 的四类是大插入、大删除、反转和重复。直到最近,就其表型效应及其作为疾病遗传决定因素的作用或其对适应的贡献而言,对单核苷酸变异(SNV)的关注比对结构变异的关注更多。这可能是因为检测SNV和预测其表型效应更容易。然而,短读长深度测序技术极大地改善了SV的检测,至少在单个个体或克隆基因组中是这样1。同时,它们的表型效应得到了更好的表征,并且已经记录了许多关于它们作为人类疾病2,3或适应新环境4的遗传决定因素的例子。
缺失和插入通常是由于移动遗传元件(MGE)插入引起的,比单核苷酸多态性(SNP)更具破坏性,并导致移码突变和蛋白质结构修饰。基因内的缺失和MGE插入几乎总是导致基因失活,当插入序列(IS)包含启动子或终止序列时,插入到非编码区域会导致相邻基因的抑制或组成性表达5。虽然必需基因的敲除对细菌适应性有明显的不利影响,但在某些情况下,非必需基因的缺失是有益的。尽管有其固有的成本,但重复也可能是有利的,并参与适应,因为它们导致基因剂量的变化;根据条件6,特定蛋白质活性的增加可能是有利的。
微生物实验进化种群通常从克隆开始。这种遗传多样性的最初缺乏,加上试管的"封闭环境"特征,导致通过水平基因转移和重组获得基因的进化潜力非常有限。在这些特定条件下,对缺失、重复和基因组内 MGE 插入的适应的贡献尤为重要;细菌通常通过功能丧失突变(主要是由于缺失或MGE插入)来适应,影响在稳定,通常营养丰富,单一栽培的人工环境中无用的基因7。在运行时间最长的大 肠杆菌 进化实验中,IS150插入在50,000代后进化的人群中特别频繁,IS元件占突变的35%,这些突变在保留其祖先点突变率的种群中达到高频率8。
进化和重新测序研究将实验进化和下一代测序(NGS)技术相结合,以研究细菌如何在表型和基因组水平上适应不同的环境条件和压力,例如不同的碳和能源,抗生素和渗透应激9,10,11.这些研究通常仅在实验终点获得进化群体或克隆的基因组信息,在某些情况下,在许多中间时间点12,13,14。这些数据提供了对适应给定环境所涉及的基因和途径的见解,但很少允许研究人员随着时间的推移跟踪从头出现和扫荡等位基因的动态。
遵循这些动态的一种方法是选择有限数量的分离感兴趣的等位基因(因为它们影响的基因的功能,因为它们在独立群体中平行扫描等),并使用扩增子测序来量化等位基因比例,在同一测序运行中汇集许多时间点15。该方法已成功用于跟踪实验16 和自然17 微生物种群中小尺寸变异(SNP或1 bp插入缺失)的动力学。然而,在较大的插入缺失或MGE插入的情况下,扩增子的大小差异会引起PCR效率差异,从而扭曲了读取和等位基因比例之间的关系。在某些情况下,两个等位基因之间的大小差异优于扩增子的经典长度。在这里,我们将三重态PCR技术与自动平行毛细管电泳相结合,以根据大小判别量化插入等位基因的相对频率。这种方法允许利用未充分利用的实验时间点来确定新兴突变等位基因的动力学,并以具有成本效益的方式跟踪其固定或丢失的频率。我们应用这种方法来跟踪通过IS10插入突变的新兴 mutS-等位基因,为突变的基因型提供超突变表型。
这种方法需要两个靶等位基因,大小差异为≥5%。首先,引物三联体被设计成产生相似大小的片段,它们共享一个共同的引物。其次,优化PCR条件,并使用野生型(WT)和突变gDNA的混合物产生校准曲线。最后,通过PCR扩增样品,并通过平行定量毛细管电泳定量每个等位基因的相对频率。
研究方案
建立此协议需要精确了解祖先序列中的插入、删除、反转或复制点。该信息通常通过末端或中间点样本的全基因组测序(WGS)获得。在下面的协议中,给出了每个步骤插入突变情况的一般原则,以及一个代表性案例,其中遵循大肠杆菌实验进化群体中 IS10 插入 mutS 基因的频率。在该群体中,终点群体的WGS确定在位置2,463和2,471之间插入了1,329 bp IS10,导致该插入位点重复。该方法适用于其他三种SV类型,讨论中给出了每种情况的具体情况。
1. 三重引物的设计
- 使用经典引物设计实践(18-24 bp,40%-60% GC 含量,尽可能以 G/C 对开始/结束,Tm 差异< 5 °C)产生引物 FW1 和 RV1。设计引物以扩增突变等位基因插入位点周围WT等位基因上的短扩增子(图1)。
注意:扩增子大小的范围为100 bp至高达3,000 bp,与毛细管电泳中使用的DNA大小阶梯一致。在本例中,扩增了155 bp扩增子。此处选择的小片段尺寸可防止整个IS10插入序列的脱靶扩增(参见第2节)。 - 在插入序列中设计第二个正向引物FW2,以产生比WT扩增子大或小约5%的第二个扩增子(图1)。这5%的尺寸差异是平行毛细管电泳装置可以可靠地区分的最小尺寸差异。因此,设计引物时,使两个扩增子的大小差异高于但尽可能接近相对阈值。
注意:确保尽量减少Tm 差异和引物二聚体的形成。在本例中,设计了第二个正向引物以产生226 bp扩增子,比WT扩增子大71 bp。在这个代表性的例子中,引物序列如下:
FW1:AAAGCATTTCGCCGAACGCC
RV1: GCGATAAATCCACTCCAGCGCC
FW2: AGTTCGCTTAGGCATGGAAG

图 1:mutS WT 基因和突变 mutS IS10 插入的三重引物设计图谱。黑色三角形代表mutS基因中的IS10插入位点。WT基因为蓝色,IS10为橙色。引物 FW1 和 RV1 标记 IS10 插入位点并产生 155 bp WT 扩增子。RV1引物和IS10内引物FW2产生第二个226 bp扩增子。请点击此处查看此图的大图。
2. PCR条件的优化
- 培养固定WT和突变等位基因克隆的过夜培养物。
- 使用任何试剂盒提取DNA。
- 量化脱氧核糖核酸。
- 通过将WT和突变DNA的提取稀释至5ng / μL来制备DNA样品。 以50/50的比例混合两个DNA样品。
- 使用 2x 即用型 PCR 预混液、0.5 μM FW1 引物、1 μM RV1 引物和 0.5 μM FW2 引物,在 20 μL 反应体积中扩增三种 DNA 样品(WT、突变体、50/50 混合物)中的 10 ng。使用2%琼脂糖凝胶通过经典电泳迁移PCR产物,并确定最佳PCR条件。
注意:应尽量减少伸长时间,以防止在突变等位基因上形成FW1和RV1扩增子。应调整退火温度,以尽量减少等位基因的偏置扩增和非特异性扩增。- 要遵循此示例中的程序,请使用以下设置:98°C持续10秒,然后进行25个循环,98°C持续1秒,58°C持续15秒,72°C循环8秒,最后伸长步骤在72°C下持续1分钟。
注意:伸长时间减少到8秒,以防止突变等位基因上的前向引物和RV1引物的>1,000 bp产物扩增(具有IS10插入的mutS )。
- 要遵循此示例中的程序,请使用以下设置:98°C持续10秒,然后进行25个循环,98°C持续1秒,58°C持续15秒,72°C循环8秒,最后伸长步骤在72°C下持续1分钟。
3. 校准曲线
- 以 10/90、25/75、40/60、50/50、60/40、75/25 和 90/10 的比例混合两个 DNA 样品 WT 和突变体。
注意:生物重复由独立的过夜细菌培养物制备。 - 使用优化的PCR条件扩增(参见第2节)。
- 量化扩增子产物。
- 将 PCR 产物稀释至 0.1 ng/μL。
- 准备平行毛细管电泳仪。
- 混合新鲜凝胶和染料(NGS定量分析试剂盒(22、33或55);本例中HS NGS片段1-6,000 bp)。
注意:有关详细说明,请参阅平行毛细管电泳HS NGS片段指南(参见 材料表)。
- 混合新鲜凝胶和染料(NGS定量分析试剂盒(22、33或55);本例中HS NGS片段1-6,000 bp)。
- 更换毛细管储存溶液和入口缓冲液,并将冲洗缓冲板放在平行毛细管电泳仪的正确抽屉位置。
- 将 2 μL HS 稀释剂标记物添加到 96 孔板中每个稀释样品的 22 μL 中。
- 将HS NGS定量分析试剂盒中的大小分子量标准(DNA大小分子量标准;范围1-6,000 bp)添加到96孔板的一个孔中。
- 将96孔板放入平行毛细管电泳仪的正确抽屉中,然后在平行毛细管电泳仪软件上选择 运行 。
- 使用数据分析软件分析结果,该软件检测并识别尺寸阶梯的每个峰,将样品的每个峰分配给其已知的实际大小。
- 使用定量试剂盒时,使用软件通过积分峰下面积来确定每个片段的DNA数量,就像在色谱数据分析中一样。同样,将样品与标准品中的已知量进行比较,以量化样品中的每个峰,并计算样品中检测到的不同峰之间的比率。
- 构建校准曲线(图2),将突变等位基因(DNA混合物)的已知比例与使用平行毛细管电泳仪器测量的比例联系起来。该校准曲线允许评估和校正方法的可靠性,以消除轻微的扩增偏差。
4. 样品制备
- 在标准条件下生长时间点样品过夜。
- 提取DNA。
- 量化脱氧核糖核酸。
- 使用优化的PCR条件扩增样品(参见第2节)。
- 在平行毛细管电泳仪中运行样品(参见步骤3.5-3.10)。
5. 等位基因定量
- 使用软件从平行毛细管电泳仪器数据中提取突变等位基因量,并通过在校准曲线上绘制这些值来计算实际比例。
结果
使用从祖先克隆中提取的DNA和从第1,000代S2.11群体中分离的超突变子克隆,我们建立了 图2所示的校准曲线。实验室制备的DNA混合物和平行毛细管电泳仪器测量的实际突变比例通过斜率为1.0706的线性关系连接,R2 为0.9705。此外,生物学重复之间存在良好的一致性;标准曲线的九个点的标准差在0.61和17.74之间。

图 2:校准标准曲线。观察到的WT/突变DNA混合物的比例与预期比例。误差条表示生物学重复之间的标准误差。请点击此处查看此图的大图。
从从356代到990代的19个时间点提取DNA,通过平行毛细管电泳扩增和定量。Prosize软件用于识别和定量每个样品中的每个扩增子。使用校准曲线将这些结果转换为等位基因比例。
图3所示的结果揭示了超突变子诱导mutS等位基因的非单调轨迹。第 680 代(第 102 天)是突变等位基因数量足以通过平行毛细管电泳检测到的第一个时间点。突变等位基因的频率迅速增加,在第713代(33代后)达到66.7%。突变等位基因频率随后停滞不前,到766代为76%,尽管之前频率迅速增加。出乎意料的是,突变等位基因频率在13代(2天)的过程中从76%降低到49%,之后它增加到固定。
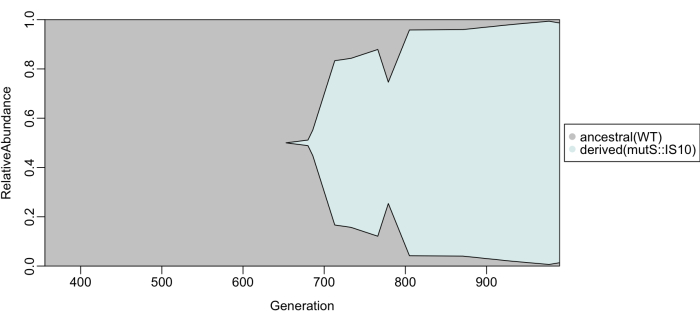
图 3:祖先 (WT) 和衍生 (mutS::IS10) 等位基因在 1,000 代实验进化过程中的动力学。祖先等位基因频率为灰色,派生的等位基因频率为蓝色。请点击此处查看此图的大图。

图 4:用于定量四种结构变体的引物设计和扩增子位置。 放大的碎片以黑白描绘。箭头代表其杂交位点的引物;虚线箭头表示引物杂交但不参与任何片段扩增,要么是因为没有成对的引物(重复和倒置),要么是延长时间缩短以避免大扩增子的扩增(插入和缺失)。 请点击此处查看此图的大图。
讨论
在这里,我们提出了一种具有成本效益的方法,该方法可以跟踪实验进化群体中新兴适应性SV等位基因的动力学。该方法将经典PCR技术和自动平行毛细管电泳相结合,可以确定两个等位基因的相对数量。一旦建立,它允许并行定量许多样品中的等位基因比例,并且比WGS便宜得多。该方法可被视为等效于非SNP突变的扩增子测序,并可解决大SV扩增子测序的技术局限性。我们已经表明,该方法使用克服PCR偏差的校准曲线准确量化了非SNP突变体的比例(图2)。
表征SV等位基因动力学可用于通过其平行时间动力学识别相关的突变,或计算选择系数并识别其随时间的变化。在我们在这里介绍的应用示例中,我们通过终点WGS确定了超突变体在各种实验进化大 肠杆菌 种群中分离或固定18。这些群体被认为是超突变体,因为它们在甲基错配修复(MMR)基因之一中具有破坏性突变(IS插入),并且在1000代的实验进化中积累的突变比不携带MMR基因突变的突变多得多(24.38±1.75对126.7±19.57)。已知MMR基因的突变会导致突变率增加,使细菌能够采样比其基线突变率允许的更多的突变12,13。然而,突变率的大幅增加也意味着有害突变的更快积累和更大的突变负荷14,15。据预测,突变体在适应新环境的人群中具有短暂的优势,因为在这种情况下,有益突变的比例更大。在高突变率下产生的有益遗传背景允许突变体通过搭便车在它们产生的有益突变上在种群中繁殖19.超突变子传播的时间动力学已经通过模拟进行了研究,并且使用我们在这里提出的方法,可以跟踪实验人群中IS介导的突变子等位基因的动力学19。
在我们的代表性结果中,SV是IS插入,但这里描述的方法可以很容易地扩展到其他类型的SV(见 图4)。三重引物设计遵循与插入定量完全相同的缺失定量逻辑:两个引物位于缺失点周围,第三个引物位于缺失序列内。对于反转和复制,两个引物位于其中一个断点周围,第三个引物位于倒置/复制片段内,靠近另一个断点并指向片段的外部。对于插入、缺失和倒置,每个扩增子都特定于其中一个等位基因,无论是祖先的还是衍生的。对于重复,其中一个扩增子对两个等位基因是共性的,另一个是衍生等位基因特有的。在根据校准曲线计算等位基因的比例时,必须考虑到这一点。
最后,我们开发了这种方法来跟踪细菌实验进化群体中的SV,其中群体水平的"宏基因组"测序是常见的做法。然而,该方法也可以应用于以pool-seq方法进行的多细胞生物的实验进化群体,或应用于宿主内病毒或微生物病原体采样17。
此方法有一些限制。首先,被比较的等位基因的大小必须相差至少5%。这阻止了我们跟踪由非常小的插入或缺失产生的等位基因的动态。幸运的是,这些等位基因动力学之后可能会进行扩增子测序。其次,必须知道衍生等位基因的序列,以便围绕插入或删除或断点设计引物。第三,每个位点都需要引物设计和优化。因此,该方法适用于跟踪有限数量的目标等位基因对。如果要在同一人群中跟踪许多等位基因,我们建议使用多个时间点的NGS。
披露声明
作者没有利益冲突需要披露。
致谢
这项工作得到了ERC HGTCODONUSE(ERC-2015-CoG-682819)对S.B.的支持,这项工作中使用的数据(部分)是通过蒙彼利埃进化科学研究所的GenSeq技术设施在ANR"Investissements d'avenir"计划(ANR-10-LABX-04-01)的支持下产生的。
材料
| Name | Company | Catalog Number | Comments |
| 96 Well Skirted PCR Plate | 4titude | 4Ti - 0740 | PCR |
| Agarose molecular biology grade | Eurogentec | EP-0010-05 | Agarose gel electrophoresis |
| Agilent DNF-474 HS NGS Fragment Kit Quick Guide for the Fragment Analyzer Systems | Agilent | PDF instruction guide | |
| Buffer TBE | Panreac appliChem | A4228,5000Pc | Agarose gel electrophoresis |
| Calibrated Disposable Inoculating Loops and Needles | LABELIANS | 8175CSR40H | Bacterial culture |
| Dneasy Blood and Tissue Kit | Qiagen | 69506 | DNA extraction |
| Electrophoresis power supply | Amilabo | ST606T | Agarose gel electrophoresis |
| Fragment Analyzer Automated CE System | Agilent | Parallel capillary electrophoresis | |
| Fragment DNA Ladder | Agilent | DNF-396, range 1-6000bp | Parallel capillary electrophoresis |
| GENTAMICIN SULFATE SALT BIOREAGENT | Sigma-Aldrich | G1264-1G | Bacterial culture |
| High Sensitivity diluent marker | Agilent | DNF-373 | Parallel capillary electrophoresis |
| High Sensitivity NGS quantitative analysis kit | Agilent | DNF-474 | Parallel capillary electrophoresis |
| Ladder quick load 1 kb plus DNA ladder | NEB | N0469S | Agarose gel electrophoresis |
| LB Broth, VegitoneNutriSelect Plus | Millipore | 28713 | Bacterial culture |
| Master Mix PCR High Fidelity Phusion Flash | Thermo Fisher Scientific | F548L | PCR |
| Primers | Eurogentec | PCR | |
| Prosize data analysis software v.4 | Agilent | V.4 | Parallel capillary electrophoresis |
| Qubit assays | Invitrogen | MAN0010876 | DNA quantification |
| Qubit dsDNA HS Assay Kit | LIFE TECHNOLOGIES SAS | Q32854 | DNA quantification |
| Thermocycler | Eppendorf | Ep gradients | PCR |
| UVbox, eBOX VX5 | Vilber Lourmat | Agarose gel electrophoresis visualisation | |
| Water for injectable preparation | Aguettant | PROAMP | PCR |
参考文献
- Mahmoud, M., et al. Structural variant calling: the long and the short of it. Genome Biology. 20 (1), 246 (2019).
- Bragg, D. C., et al. Disease onset in X-linked dystonia-parkinsonism correlates with expansion of a hexameric repeat within an SVA retrotransposon in TAF1. Proceedings of the National Academy of Sciences. 114 (51), 11020-11028 (2017).
- Stransky, N., Cerami, E., Schalm, S., Kim, J. L., Lengauer, C. The landscape of kinase fusions in cancer. Nature Communications. 5, 4846 (2014).
- Tenaillon, O., et al. The molecular diversity of adaptive convergence. Science. 335 (6067), 457-461 (2012).
- Vandecraen, J., Chandler, M., Aertsen, A., Van Houdt, R. The impact of insertion sequences on bacterial genome plasticity and adaptability. Critical Reviews in Microbiology. 43 (6), 709-730 (2017).
- Andersson, D. I., Gene Hughes, D. amplification and adaptive evolution in bacteria. Annual Review of Genetics. 43, 167-195 (2009).
- Bailey, S. F., Bataillon, T. Can the experimental evolution programme help us elucidate the genetic basis of adaptation in nature. Molecular Ecology. 25 (1), 203-218 (2016).
- Consuegra, J., et al. Insertion-sequence-mediated mutations both promote and constrain evolvability during a long-term experiment with bacteria. Nature Communications. 12 (1), 980 (2021).
- Burch, C. L., Romanchuk, A., Kelly, M., Wu, Y., Jones, C. D. Genome-wide determination of barriers to horizontal gene transfer. bioRxiv. , (2022).
- Slomka, S., et al. Experimental evolution of Bacillus subtilis reveals the evolutionary dynamics of horizontal gene transfer and suggests adaptive and neutral effects. Genetics. 216 (2), 543-558 (2020).
- Choudhury, D., Saini, S. Evolution of Escherichia coli in different carbon environments for 2,000 generations. Journal of Evolutionary Biology. 32 (12), 1331-1341 (2019).
- Tenaillon, O., et al. Tempo and mode of genome evolution in a 50,000-generation experiment. Nature. 536 (7615), 165-170 (2016).
- Behringer, M. G., et al. Escherichiacoli cultures maintain stable subpopulation structure during long-term evolution. Proceedings of the National Academy of Sciences. 115 (20), 4642-4650 (2018).
- Voordeckers, K., et al. Adaptation to high ethanol reveals complex evolutionary pathways. PLoS Genetics. 11 (11), 1005635 (2015).
- Levy, S. F., et al. Quantitative evolutionary dynamics using high-resolution lineage tracking. Nature. 519 (7542), 181-186 (2015).
- Bruger, E. L., Marx, C. J. A decade of genome sequencing has revolutionized studies of experimental evolution. Current Opinion in Microbiology. 45, 149-155 (2018).
- Grubaugh, N. D., et al. An amplicon-based sequencing framework for accurately measuring intrahost virus diversity using PrimalSeq and iVar. Genome Biology. 20 (1), 8 (2019).
- Bedhomme, S., et al. Evolutionary changes after translational challenges imposed by horizontal gene transfer. Genome Biology and Evolution. 11 (3), 814-831 (2019).
- Tenaillon, O., Toupance, B., Le Nagard, H., Taddei, F., Godelle, B. Mutators, population size, adaptive landscape and the adaptation of asexual populations of bacteria. Genetics. 152 (2), 485-493 (1999).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。
我们使用 cookie 来增强您在我们网站上的体验。
继续使用我们的网站或单击“继续”,即表示您同意接受我们的 cookie。