Method Article
Verwendung von Mikrofluidik und Fluoreszenzmikroskopie zur Untersuchung der Montagedynamik von einzelnen Actin-Filamenten und -Bündeln
In diesem Artikel
Zusammenfassung
Wir präsentieren Protokolle für einfache mikrofluidische Aktinfilament-Assays in Kombination mit der Fluoreszenzmikroskopie, die es ermöglichen, einzelne Aktinfilamente in Echtzeit genau zu überwachen und sie nacheinander verschiedenen Proteinlösungen auszusetzen.
Zusammenfassung
Um die komplexen molekularen Mechanismen zu entschlüsseln, die den Auf- und Abbau von Aktinfilamenten regulieren, ist es ein großer Vorteil, einzelne Reaktionen live unter gut kontrollierten Bedingungen zu überwachen. Zu diesem Zweck sind in den letzten 20 Jahren Live-Single-Filament-Experimente entstanden, die hauptsächlich die Total-Internal-Reflection-Fluoreszenz-Mikroskopie (TIRF) verwenden, und haben eine Fülle von Schlüsselergebnissen geliefert. Um die Möglichkeiten dieser Experimente weiter auszubauen und wiederkehrende problematische Artefakte zu vermeiden, haben wir 2011 einfache Mikrofluidik in diese Assays eingeführt. Diese Studie beschreibt unser grundlegendes Protokoll, bei dem einzelne Aktinfilamente an einem Ende an der passivierten Deckglasoberfläche verankert sind, sich an der Strömung ausrichten und nacheinander verschiedenen Proteinlösungen ausgesetzt werden können. Wir stellen auch die Protokolle für spezifische Anwendungen vor und erklären, wie kontrollierte mechanische Kräfte dank des viskosen Widerstands der fließenden Lösung angewendet werden können. Wir heben die technischen Vorbehalte dieser Experimente hervor und stellen kurz mögliche Entwicklungen vor, die auf dieser Technik basieren. Diese Protokolle und Erklärungen, zusammen mit der heutigen Verfügbarkeit von einfach zu bedienenden Mikrofluidikgeräten, sollten es Nicht-Spezialisten ermöglichen, diesen Assay in ihren Labors zu implementieren.
Einleitung
Der Auf- und Abbau von Aktinfilamenten und Aktinfilamentnetzwerken wird durch mehrere biochemische Reaktionen gesteuert und hängt vom mechanischen Kontext ab. Um Einblick in diese komplexen Mechanismen zu erhalten, ist es von unschätzbarem Wert, einzelne Reaktionen auf einzelne Filamente (in ausreichend großer Anzahl) beobachten zu können. In den letzten Jahrzehnten hat sich die Beobachtung dynamischer Aktinfilamente in Echtzeit, meist unter Verwendung der TIRF-Mikroskopie (Total Internal Reflection Fluorescence), zu einer Schlüsseltechnik entwickelt und eine beeindruckende Liste von Ergebnissen geliefert, die mit biochemischen Assays in Bulk-Lösung nicht hätten erzielt werden können1.
Um dies zu erreichen, muss man fluoreszierend markierte Aktinfilamente nahe an der Oberfläche des Mikroskop-Deckglases halten, während man sie Lösungen von aktinbindenden Proteinen (ABPs) aussetzt, die auch fluoreszierend markiert werden können. Auf diese Weise können Ereignisse an einzelnen Filamenten unter gut kontrollierten biochemischen Bedingungen überwacht und so die Reaktionsgeschwindigkeiten quantifiziert werden. Es sollten jedoch eine Reihe spezifischer Einschränkungen in Betracht gezogen werden. Die künstliche Aufrechterhaltung von Filamenten in der Nähe der Oberfläche, oft dank mehrerer Verankerungspunkte oder durch die Verwendung eines Überfüllungsmittels wie Methylcellulose, kann ihr Verhalten verändern (z. B. Pausen in ihrer Polymerisation und Depolymerisationverursachen 2). Die Verfolgung der Kontur jedes Filaments kann eine Herausforderung darstellen, insbesondere wenn sich im Laufe der Zeit neue Filamente oder Filamentfragmente im Sichtfeld ansammeln. Die Reaktionen finden in einem endlichen Volumen statt, in dem die Konzentration von Aktinmonomeren und ABPs im Laufe der Zeit variieren kann, was es möglicherweise schwierig macht, genaue Geschwindigkeitskonstanten abzuleiten. Schließlich ist die Erneuerung oder Änderung der Lösung von ABPs in weniger als 30 s schwierig zu erreichen und führt oft zu einem inhomogenen Proteingehalt in der Probe.
Vor etwas mehr als 10 Jahren, inspiriert von dem, was bereits getan wurde, um einzelne Desoxyribonukleinsäure (DNA) Stränge3 zu untersuchen, führten wir eine neue Technik ein, die auf Mikrofluidik basiert, um einzelne Aktinfilamente zu beobachten und zu manipulieren4. Es erlaubt einem, die oben genannten Einschränkungen klassischer Single-Filament-Techniken zu umgehen. In diesen Mikrofluidik-Assays werden Aktinfilamente aus Spektrin-Aktin-Samen gezüchtet, die auf dem Deckglas adsorbiert sind. Filamente werden somit von einem Ende nur am Boden der mikrofluidischen Kammer verankert und schwanken über der Oberfläche, ohne zu kleben. Filamente richten sich an den Fluss eingehender Lösungen aus, erleichtern so die Überwachung ihrer Konturlänge und halten sie in einem flachen Bereich über dem Deckglas, in dem TIRF verwendet werden kann. Verschiedene Lösungen fließen gleichzeitig in die Kammer, ohne zu mischen, und die Filamente können ihnen nacheinander und schnell ausgesetzt werden.
Hier schlagen wir eine Reihe grundlegender Protokolle vor, um Einzelaktin-Filament-Mikrofluidik-Assays im Labor einzurichten. Deckgläser und Mikrofluidikkammern können im Voraus (in einem halben Tag) vorbereitet werden, und das Experiment selbst, bei dem mehrere biochemische Bedingungen getestet werden können, ist in weniger als einem Tag abgeschlossen.
Protokoll
1. Mikrofluidische Kammervorbereitung
- Wählen Sie eine SU-8 Masterform mit mehreren Kammermustern. Typische Kammern sind kreuzförmig mit drei Einlässen und einem Auslass, 20 μm hoch und 800 μm breit (Abbildung 1). Solche Master-Formen können von externen Unternehmen gekauft oder in akademischen Labors (z. B. Gicquel, Y. et al.5) hergestellt werden.
- Legen Sie Klebeband um den Rand der Form.
- Legen Sie ~50 cm langes, 19 mm breites, transparentes Standard-Büroband (siehe Materialtabelle) auf eine Bank, klebrig mit der Seite nach oben. Platzieren Sie die Form vertikal an einem Ende und entlang der Mittellinie des Bandes.
- Rollen Sie die Form zum anderen Ende des Bandes, um einen 1 cm langen Rand um die Form zu erzeugen. Falten Sie das Klebeband über den Boden der Form.
- Bereiten Sie Polydimethylsiloxan (PDMS) -Lösung vor.
- In einer Einweg-Wiegeschale 25-30 g PDMS-Basis (Table of Materials) direkt gießen. Fügen Sie 10% Gewicht / Gewicht PDMS-Härter (Tabelle der Materialien) mit einer Einweg-Pasteur-Pipette aus Kunststoff hinzu.
- Manuell und gründlich mit einem Kunststoffstab mischen. Stellen Sie sicher, dass der Härter gut in die PDMS-Basis integriert ist, auch wenn durch Rühren viele Blasen entstehen.
- Entgasen Sie die PDMS-Lösung in einem Vakuum-Exsikkator (Table of Materials) für mindestens 5 min bei Raumtemperatur (RT). Blasen dehnen sich aus, steigen an die Oberfläche und platzen, wenn das Vakuum gebrochen wird.
- Gießen Sie die PDMS-Lösung über die SU-8-Form. Verwenden Sie einen Kunststoffstäbchen, um so viel wie möglich von der Mischung zu kratzen und zu übertragen.
- Entgasen Sie PDMS zum zweiten Mal (5 min im Vakuum-Exsikkator). Stellen Sie sicher, dass Sie die meisten Blasen loswerden (ein paar kleine Blasen an der oberen Oberfläche sind in Ordnung).
- Legen Sie die Form mindestens 5 h lang bei 70 °C in einen Ofen, damit sich das PDMS vernetzen und verfestigen kann.
- Entfernen Sie feste PDMS-Kammern aus der Form.
ACHTUNG: Siliziumwafer für SU-8-Formen sind extrem zerbrechlich, daher ist bei der Trennung des PDMS von Wafern große Vorsicht geboten. Arbeiten Sie auf einer harten, ebenen Oberfläche und halten Sie den Wafer flach auf der Oberfläche.- Machen Sie mit einer Rasierklinge einen kreisförmigen Schnitt im PDMS, etwa 1 cm vom Rand der Form entfernt. Alle Muster müssen mindestens 0,5 cm im Schnitt sein. Den zentralen PDMS-Block vorsichtig mit sanftem Zug abziehen.
VORSICHT: Halten Sie beim Abziehen die SU-8-Form flach auf der Tischplatte, um ein Brechen zu verhindern. - Legen Sie PDMS auf saubere Aluminiumfolie, die geformte Oberfläche, die der Aluminiumfolie zugewandt ist, um ihre Oberfläche vor Staub zu schützen und Muster besser sichtbar zu machen.
- Machen Sie mit einer Rasierklinge einen kreisförmigen Schnitt im PDMS, etwa 1 cm vom Rand der Form entfernt. Alle Muster müssen mindestens 0,5 cm im Schnitt sein. Den zentralen PDMS-Block vorsichtig mit sanftem Zug abziehen.
- Wählen und schneiden Sie eine Kammer mit einer Rasierklinge, die mindestens 0,5 cm vom Muster entfernt ist. Der resultierende PDMS-Block ist etwa 0,5 cm hoch, 1,5 cm breit und 3 cm lang. Durchbohren Sie drei Einlässe und einen Auslass mit einem Biopsiestempel 0,75 mm I.D. (Table of Materials).
- Reinigen Sie die PDMS-Kammer mit hochreinem Ethanol (Table of Materials) und trocknen Sie sie mit einer Sicherheitsblaspistole (Table of Materials) an der Luft. Legen Sie das PDMS mit dem Muster nach oben in eine saubere Petrischale und schließen Sie die Schale mit ihrem Deckel.
2. Glasabdeckglasreinigung
HINWEIS: Hier wird ein Standard-Deckglasreinigungsverfahren beschrieben, das auf einer Reihe von Beschallungsschritten basiert. Andere Glasabdeckglasreinigungsverfahren wurden in vielen anderen Publikationen beschrieben, die ähnlich zufriedenstellende Ergebnisse erzielen können 6,7,8,9.
- Legen Sie 10-20 Deckgläser (40 mm lang) auf einen Halter aus Polytetrafluorethylen (PTFE) (Materialverzeichnis). Beschallen Sie die Deckgläser in 0,5 l 2% iger Glasreinigungslösung (Materialtabelle) in einem 1 L Glasbecherglas (35 °C, 30 min).
- Entsorgen Sie die Glasreinigungslösung und spülen Sie die Deckgläser in mindestens drei aufeinanderfolgenden 0,5 L Bädern ausgiebig mit dH2O ab.
- Bereiten Sie 0,5 L 2 M KOH in einem 1 L Glasbecherglas vor. Beschallen Sie die Deckgläser in KOH (RT, 30 min). KOH entsorgen und die Deckgläser mit dH2O in mindestens drei 0,5 L Bädern abspülen.
ACHTUNG: Verwenden Sie geeignete Laborsicherheitsausrüstung (Handschuhe, Brille und Laborkittel). - Übertragen und beschallen Sie die Deckgläser in 0,5 l hochreinem Ethanol (RT, 30 min). Deckgläser können bis zu 2 Wochen in Ethanol aufbewahrt werden. Schließen Sie das Becherglas mit thermoplastischer Folie (Table of Materials), um eine Verdunstung zu verhindern. Vor Gebrauch das Deckglas mit Luftstrom trocknen.
3. PDMS-Kammermontage
- Die Heizplatte auf 100 °C vorheizen. Legen Sie bis zu drei gereinigte PDMS-Kammern und Glasabdeckungen in eine saubere Petrischale. Legen Sie die geöffnete Petrischale in einen tiefen UV-Reiniger (λ = 185 nm, siehe Materialtabelle) und setzen Sie sie 3-5 min UV-Licht aus.
HINWEIS: Alternativ können PDMS-Kammern und Deckgläser 30 s lang Luft oder Sauerstoffplasma ausgesetzt werden. - Positionieren Sie die PDMS-Kammer vorsichtig über dem Deckglas. Stellen Sie sicher, dass die beiden in Kontakt gebrachten Oberflächen direkt UV-Strahlen ausgesetzt waren. Das PDMS haftet automatisch am Glas und die Kammer wird deutlich sichtbar.
- Um die an der PDMS-Coverslip-Schnittstelle eingeschlossene Luft zu entfernen, drücken Sie die Oberfläche sehr vorsichtig mit einem Finger an. Für eine engere Verklebung stärker über Ecken und Seiten drücken. Stellen Sie sicher, dass die Decke der Kammer nicht mit der Glasoberfläche in Berührung kommt.
- Stellen Sie die Kammer mit dem Glasboden zur heißen Platte bei 100 °C für 5 min. Nach diesem Schritt werden die Glas-PDMS-Bindungen dauerhaft und Kammern können nur einmal verwendet werden. Verwenden Sie die Kammer sofort oder bewahren Sie sie bis zu einer Woche in einer sauberen Petrischale auf.
4. [OPTIONAL] Direkte Passivierung und Funktionalisierung
HINWEIS: Je nach Anwendung können Kammern entweder nach dem Anschluss an das mikrofluidische Steuergerät (siehe Materialtabelle) oder durch manuelles Einspritzen von Lösungen direkt in die Kammer mit einer Pipette vor dem Anschluss an das mikrofluidische Gerät passiviert und funktionalisiert werden. Letzteres bietet den Vorteil, weniger Reagenz zu verbrauchen und eine mögliche Kontamination zu vermeiden, indem die Lösung durch den Polyetheretherketonschlauch (PEEK) des mikrofluidischen Geräts fließt. In allen folgenden Schritten werden Lösungen injiziert, indem die Pipettenspitze direkt in den Auslass gesteckt wird. Um Blasen in der Kammer zu vermeiden, stellen Sie sicher, dass ein winziges Tröpfchen aus der Pipettenspitze herausragt, wenn Sie die Spitze in den Auslass der PDMS-Kammer stecken. Entfernen Sie ebenfalls die Pipettenspitze, bevor das gesamte Volumen injiziert wurde.
- Injizieren Sie 20 μL PLL-PEG (1 mg/ml in phosphatgepufferter Kochsalzlösung (PBS)). Inkubieren Sie für mindestens 1 Stunde (oder über Nacht) bei RT. Um eine Verdunstung zu verhindern, legen Sie die PDMS-Kammer in eine feuchte Box (z. B. eine leere Kippbox mit Wasser im unteren Fach und die PDMS-Kammer auf der Kipphalteplattform).
- Injizieren Sie 20 μL 100 pM Spektrin-Aktin-Samen (in F-Puffer, siehe Tabelle 1 und Tabelle 2). Warten Sie nicht länger als 1 min. Passen Sie die Samenkonzentration und das Timing an, um die Samenoberflächendichte abzustimmen, hoch genug für große Statistiken und niedrig genug, damit sich Filamente nicht überlappen.
HINWEIS: Wenn keine Spektrin-Aktin-Samen verfügbar sind, verwenden Sie alternativ biotin-funktionalisierte kurze Filamentsegmente, die auf einem streptavidinbeschichteten Deckglas 9,10 immobilisiert werden. - [OPTIONAL] Injizieren Sie 20 μL 5% Rinderserumalbumin (BSA) in den F-Puffer. Lassen Sie bei RT für 10 min.
- [OPTIONAL] Injizieren Sie 20 μL 1 mg/ml β-Casein in F-Puffer. Lassen Sie bei RT für 10 min.
HINWEIS: Befolgen Sie die Schritte 4.3 und/oder 4.4, um die Kammer weiter zu passivieren. Die Wahl der Passivierung hängt von den verwendeten Proteinen ab und funktioniert nicht bei allen ABPs gleich gut. Bei alleiniger Verwendung von Aktin ist PLL-PEG oder BSA ausreichend.
5. Mikrofluidisches Gerät anschließen
HINWEIS: Verwenden Sie ein druckbasiertes mikrofluidisches System mit bis zu vier Kanälen, um die Durchflüsse in der mikrofluidischen Kammer zu steuern (Abbildung 1A, siehe Materialtabelle). Um Blasenbildung in den mikrofluidischen Schläuchen zu vermeiden und die Strömungsstabilität zu stören, entgasen Sie alle Lösungen. Geben Sie 5 ml dH20 und 10 ml F-Puffermaterial in einen Vakuum-Exsikkator, der an eine Vakuumpumpe angeschlossen ist (Endvakuum <250 mbar) und entgasen Sie für mindestens 1 h bei RT.
- Spüleinlässe + Auslassschläuche mit dH2O (500 μL, 300 mbar).
- Füllen Sie alle 2 mL Reservoirröhrchen (siehe Materialtabelle) mit 300 μL F-Puffer. Stellen Sie den Druck auf 300 mbar ein und lassen Sie fünf bis acht Tropfen verschwenden. Wiederholen Sie den Vorgang für jeden Kanal und stellen Sie den Druck auf 0 ein.
- Schließen Sie den Auslass an und spülen Sie die Kammer ausgiebig ab.
- Stellen Sie den Druck für das Vorratsrohr 4 (Auslass) auf 50 mbar ein. Sobald ein Tröpfchen aus dem Schlauchende austritt, schließen Sie den Schlauch an den Auslass der PDMS-Kammer an. Die Flüssigkeit füllt sich in der Kammer auf und kommt aus allen Einlässen.
- [OPTIONAL] Wenn die Kammer direkt passiviert wurde (Abschnitt 4), stellen Sie den Druck auf 100 mbar ein, um die Kammer mit 50-100 μL F-Puffer (3-5 min) zu spülen. Entfernen Sie die überschüssige Flüssigkeit an den Einlässen mit einem Reinigungstuch.
- Druck auf 20 mbar einstellen.
- Schließen Sie Einlässe an.
- Stellen Sie den Druck für das Reservoirrohr 1 auf 50 mbar ein. Um das Einbringen von Luftblasen zu vermeiden, stellen Sie sicher, dass ein Tröpfchen aus dem Schlauch und dem PDMS-Einlass kommt.
- Verbinden Sie den Schlauch mit dem Einlass 1 (die beiden Tröpfchen verschmelzen beim Anschließen). Stellen Sie den Druck auf 30 mbar ein.
- Wiederholen Sie die Schritte 5.4.1-5.4.2, um die Eingänge 2 und 3 anzuschließen.
- Stellen Sie den Druck aller Einlässe auf 20 mbar und den Ausgangsdruck auf 0 mbar ein. Stellen Sie sicher, dass die Durchflussraten in den Einlässen ungefähr gleich sind (siehe Abschnitt Fehlerbehebung).
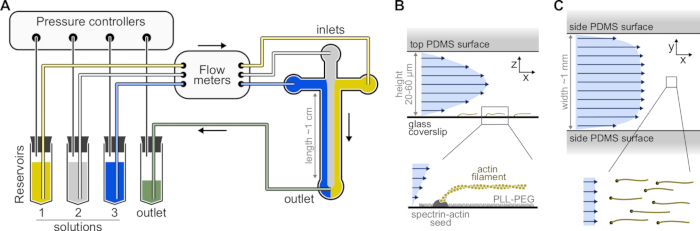
Abbildung 1: Injektion von Lösungen durch eine mikrofluidische Kammer . (A) Standard-Mikrofluidik-Aufbau für Einzelaktin-Filament-Experimente. Proteinlösungen, die in Reservoirs 1-3 platziert werden, werden durch Anpassung des Drucks in der Gasphase in die Kammer gedrückt. Die erzeugten Durchflussmengen werden von Durchflussmessern gemessen. Innerhalb der mikrofluidischen Kammern vermischen sich die Lösungen nicht und nehmen je nach den angewendeten relativen Drücken (hier gleicher Druck auf alle Einlässe) den Raum ein. Typische Abmessungen: Reservoirrohre enthalten bis zu 2 ml Lösung. PEEK-Schläuche (0,25 mm Innendurchmesser) verbinden die Behälter mit den Durchflussmessern (nach 10 cm Schlauch) und dann mit der PDMS-Kammer (nach weiteren 70 cm). Siliziumröhren und Edelstahl-Rohrkupplungen werden verwendet, um die PEEK-Schläuche mit den PDMS-Einlässen zu verbinden. Der mikrofluidische Hauptkanal ist 20-60 μm hoch, etwa 1 mm breit und 1 cm lang. (B,C) Strömungsprofile innerhalb der mikrofluidischen Kammer. (B) Das Fluid erzeugt ein parabolisches Profil über die Kammerhöhe: v(z) = 6z(h-z)R/h3w, wobei h und w die Kammerhöhe und -breite und R die Gesamtdurchflussrate ist. Boden: Einzelaktin-Filament, polymerisiert aus oberflächenverankerten Spektrin-Aktin-Samen. (C) Wenn die Kammerbreite wesentlich größer als ihre Höhe ist, ist die Strömung in der gesamten Kammer nahezu gleichmäßig, außer an den PDMS-Oberflächen, wo sie auf Null geht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
6. Konfiguration des Setups mit Standard-Durchflussraten
HINWEIS: Das computergesteuerte Drucksystem ermöglicht eine einfache und präzise Einstellung der Drücke aller an die PDMS-Kammer angeschlossenen Ein- und Ausgänge, also die Kontrolle der ein- und ausgehenden Durchflussraten. Voreingestellte Konfigurationen können mit einem einzigen Mausklick gespeichert und ein-/ausgeschaltet werden. Im Folgenden finden Sie die empfohlenen Konfigurationen (sofern nicht anders angegeben, ist der Ausgangsdruck auf 0 mbar eingestellt). Die erwarteten Durchflussraten für diese voreingestellten Konfigurationen finden Sie in Tabelle 3. Die hier angegebenen Drücke müssen je nach Kammergeometrie und Systemkonfiguration angepasst werden.
-
Ändern: Verwenden Sie diese Voreinstellung, wenn Sie ein oder mehrere Reservoirs ändern. Es erzeugt einen milden Rückfluss in den interessierenden Schläuchen, um das Einbringen von Blasen zu verhindern.
- Stellen Sie alle Eingangsdrücke auf 12 mbar und den Ausgangsdruck auf 5 mbar ein (Abbildung 2B).
-
High Flow 'All': Verwenden Sie diese Voreinstellung, um schnell drei Lösungen parallel zu injizieren. Sie erreichen die Kammer innerhalb von 4 Minuten.
- Stellen Sie alle Eingangsdrücke auf 150 mbar ein.
-
High Flow 'x': Verwenden Sie diese Voreinstellung, um schnell eine Lösung zu injizieren. Es erreicht die Kammer innerhalb von 3 Minuten (Abbildung 3A-C).
- Stellen Sie den Eingangsdruck 'x' auf 150 mbar (~15 μL/min) ein. Der Druck in den anderen Einlässen wird auf etwa 100 mbar eingestellt, so dass die resultierende Durchflussrate in diesen Einlässen ~500 nL/min beträgt.
-
Mid Flow 'Alle': Verwenden Sie diese Voreinstellung, um das System anzuhalten.
- Stellen Sie alle Einlässe auf 20 mbar ein (Abbildung 2A).
-
Mid Flow 'x': Verwenden Sie diese Voreinstellung, damit die Lösung 'x' den größten Teil der Hauptkanalbreite ausfüllen kann (siehe Abbildung 2C,D), während die anderen Einlasslösungen auf die Kanalseiten beschränkt werden. Aktinfilamente in der Kammer werden somit nur dem biochemischen Zustand ausgesetzt, der durch die Lösung "x" auferlegt wird.
- Stellen Sie den Eingangsdruck 'x' auf 12 mbar ein. Stellen Sie den Druck in den anderen Einlässen ein und stellen Sie ihn auf ~ 9 mbar ein, so dass ihre jeweiligen Durchflussraten ~ 150 nL / min betragen.
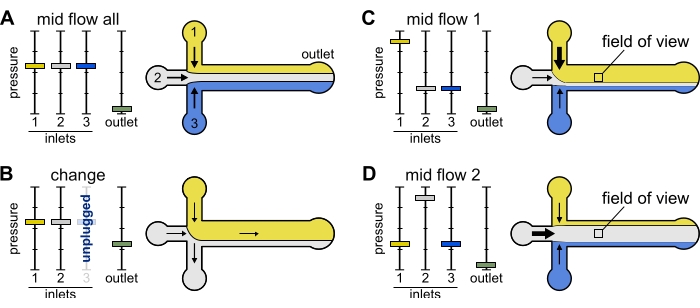
Abbildung 2: Der Druck, der auf jedes Reservoir ausgeübt wird, steuert die Aufteilung / räumliche Verteilung von Lösungen innerhalb der mikrofluidischen Kammer. (A) Bei gleichem Druck, der auf die Reservoirs ausgeübt wird, nimmt jede Lösung ein Drittel der Kammer ein. (B) Beim Wechsel eines Reservoirrohrs (hier Reservoir 3) sinkt der effektive Druck auf Null, wodurch ein Rückfluss entsteht. (C,D) Die Erhöhung des relativen Drucks auf eines der Reservoirs ermöglicht die Exposition der Glasoberfläche gegenüber einer einzigen Lösung. Das Sichtfeld in der Mitte der Kammer kann sequentiell den Lösungen 1 und 2 ausgesetzt werden, indem zwischen der Konfiguration Mid Flow 1 (C) und Mid Flow 2 (D) abgewechselt wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
7. Lösung 'x' ändern
HINWEIS: Wie in Abbildung 3A-C gezeigt, ist es wichtig zu bedenken, dass Lösungen Minuten benötigen, um von einem Reservoirrohr zum Hauptkanal der Kammer zu fließen. Diese minimale "Totzeit" wird durch das im Schlauch enthaltene Flüssigkeitsvolumen und das Strömungsprofil innerhalb des Schlauches auferlegt (Abbildung 3A-C).
- Bereiten Sie 200-300 μL Lösung in einem neuen Reservoirrohr vor. Setzen Sie den Druck auf Einstellung ändern (siehe Abschnitt 6).
- Schrauben Sie das Reservoirrohr des Einlasses 'x' ab. Die Lösung im Schlauch fließt langsam rückwärts, von der Kammer zur freien Schlauchspitze. Der gemessene Durchfluss wird negativ (Abbildung 2B).
- Sobald sich an der Schlauchspitze ein winziger Tröpfchen gebildet hat, schrauben Sie das neue Rohr mit der frischen Lösung ein. Sobald das Rohr richtig auf das Drucksystem angezogen ist, kehrt die Durchflussrate des Einlasses in den positiven Bereich zurück.
- Stellen Sie die Druckeinstellung auf High Flow 'x' ein.
- Abhängig von der mikrofluidischen Konfiguration und der Kammergeometrie warten Sie 3-5 Minuten, bis die Lösung den Schlauch vollständig gefüllt und die Kammer erreicht hat.
- [OPTIONAL] Befolgen Sie diesen Prozess, indem Sie den Anstieg der Fluoreszenz im Laufe der Zeit messen (Abbildung 3C).
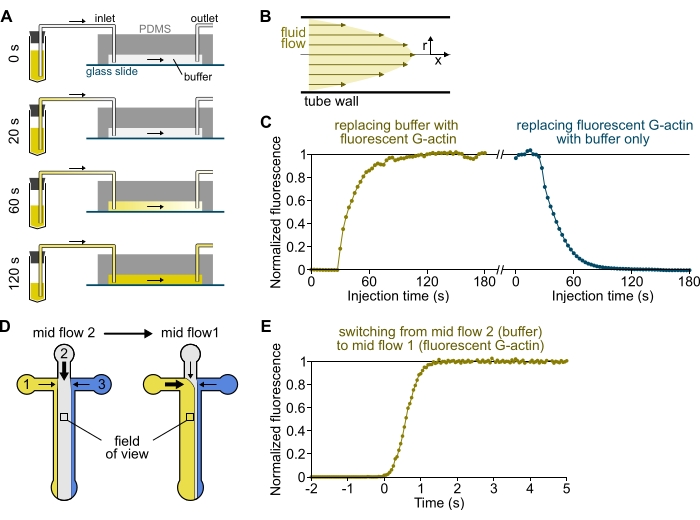
Abbildung 3: Verzögertes Eintreffen von Lösungen aus den Reservoirs in die PDMS-Kammer und schnelle Änderung der biochemischen Bedingungen. (A-C) Verzögerte Ankunft von Lösungen aus den Reservoirs in der PDMS-Kammer. (A) Abhängig von der Kammergeometrie, der Rohrlänge und dem angelegten Druck an den Einlässen erfolgt der Ersatz einer Lösung durch eine andere nicht augenblicklich. Nach dem Wechsel des Reservoirrohrs in ein Rohr, das eine fluoreszierende Lösung (0 min) enthält, füllt sich die Lösung schrittweise in den Schlauch (0,4 min) und die PDMS-Kammer (1-2 min). Das indikative Timing wird für einen angelegten Druck von 150 mbar, einen 80 cm langen PEEK-Schlauch und eine 1600 μm breite, 20 μm hohe PDMS-Kammer angegeben. (B) Das parabolische Strömungsprofil innerhalb des PEEK-Schlauchs erzeugt einen effektiven Fluoreszenzgradienten entlang des radialen Schlauchprofils und innerhalb der Kammer (siehe auch Abbildung 1B). (C) Das verzögerte Eintreffen von Lösungen kann quantifiziert werden, indem das Hintergrundepifluoreszenzsignal in der Kammer als Funktion der Zeit gemessen wird. Versuchsbedingungen: 0,5 μM 10% Alexa-568-markiertes G-Aktin wird mit 150 mbar über einen Durchflussmesser und einen 80 cm PEEK-Schlauch injiziert. (D,E) Rasche Veränderung der biochemischen Bedingungen. (D) Muster der eingehenden Lösungen in zwei Mid-Flow-Bedingungen. (E) Erhöhung der Hintergrundfluoreszenz als Anzeige der Aktinkonzentration. Die Zeit t = 0 wird als Beginn der Fluoreszenzzunahme eingestellt. Lösung 1: 0,5 μM 10% Alexa-488-markiertes G-Aktin, Lösung 2: F-Puffer. (C,E) PDMS-Kammer: 20 μm hoch und 1600 μm breit. Die Epifluoreszenzintensität, ~ 2 μm über der Oberfläche, wurde quantifiziert, indem das Signal über das gesamte Sichtfeld gemittelt wurde, normalisiert auf 0 in Abwesenheit von Fluorophor und 1 bei maximaler Intensität. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
8. Einfaches Einzelfilament-Experiment: Adenosindiphosphat (ADP)-Aktin Widerhakenend-Depolymerisation
HINWEIS: In diesem Abschnitt wird von einer nicht funktionalisierten Kammer ausgegangen (nur Abschnitt 5). Wenn die Kammer direkt funktionalisiert wurde (Abschnitt 4), beginnen Sie mit Schritt 8.4.
- Oberflächenfunktionalisierung mit Aktinfilamentsamen:
- Ändern Sie die Lösung 3 in200 μL von 50 pM Spektrin-Aktin-Samen 11 im F-Puffer (siehe Abschnitt 7).
HINWEIS: Alternativ, wenn Spektrin-Aktin-Samen nicht verfügbar sind, kann man biotin-funktionalisierte kurze Filamentsegmente verwenden, die auf einem streptavidinbeschichteten Deckglas immobilisiert werden (siehe 9,10 für Details). - Für 2 min mit High Flow 3 injizieren.
HINWEIS: Passen Sie die Konzentration und Zeit in Abhängigkeit von der endgültigen Samendichte an.
- Ändern Sie die Lösung 3 in200 μL von 50 pM Spektrin-Aktin-Samen 11 im F-Puffer (siehe Abschnitt 7).
- Oberflächenneutralisierung:
- Wechseln Sie Röhrchen 3 mit 300 μL von 5% BSA im F-Puffer.
- Inject für 5 min bei High Flow 3, gefolgt von 5 min bei Mid Flow 3. Reduzieren Sie während dieses zweiten Schritts den Druck in den Kanälen 1 und 2 auf 7-8 mbar, um einen Gegenstrom von ~-100 nL / min zu erhalten, so dass die gesamten Kammeroberflächen BSA-passiviert sind.
HINWEIS: Da die BSA-Lösung viskoser ist, müssen die Drücke entsprechend angepasst werden.
- Wechseln Sie Rohr 3 auf F-Puffer und spülen Sie den Kanal (5 min, High Flow 3).
- Bereiten Sie die folgenden 200-300 μL-Lösungen vor, wobei alle Proteine in F-Puffer verdünnt werden:
Einlass 1, Polymerisationslösung: 1 μM 10% Alexa-488 markiertes G-Aktin, 1 μM Profilin (Tabelle 1).
Einlass 2, alternde Lösung: 0,15 μM 10% Alexa-488 markiert G-Actin.
Einlass 3, Depolymerisationslösung: nur F-Puffer.
HINWEIS: Profilin wird hier verwendet, um spontane Keimbildung zu verhindern und eine konstante Konzentration von G-Aktin aufrechtzuerhalten. - Wechseln Sie die Rohre 1 bis 3 (Abschnitt 7). Injizieren Sie mit der Voreinstellung High Flow All für 3-4 min. Die drei Lösungen haben nun den PEEK-Schlauch gefüllt und die Kammer erreicht (Abbildung 3A). Die Glasoberfläche kann jeder Einlasslösung ohne Totzeit ausgesetzt werden (<1 s, Abbildung 3D,E).
- Schalten Sie das Mikroskop ein. Stellen Sie die Einstellungen ein: 150 mW 488 nm Anregungslaser bei 10% -20% Leistung, 100-200 ms Kamerabelichtungszeit, 200-300 nm TIRF-Eindringtiefe, 60x Objektiv. Diese Einstellungen werden im gesamten Manuskript verwendet.
- Filamentpolymerisation (Abbildung 4A):
- Stellen Sie die Druckeinstellung auf Mid Flow 1 für ~ 10 min.
- [OPTIONAL] Rekordpolymerisation (1 Rahmen/20 s, TIRF). Filamente sollten bei etwa 10 Untereinheiten/Sekunde (sub/s)1,12 polymerisieren.
- Filteralterung: Stellen Sie die Druckeinstellung für 15 min auf Mid Flow 2 ein. Bei der kritischen Konzentration, 0,15 μM G-Aktin, bleibt die Filamentlänge konstant und die Filamente werden zu >99% ADP-F-Aktin4.
- Depolymerisation (Abbildung 4A):
- Starten Sie die Aufnahme mit 1 Bild/5 s im Epifluoreszenzmodus. Da es in den Kanälen 2 und 3 einen sehr niedrigen Fluoreszenzhintergrund gibt, ist es nicht notwendig, TIRF zu verwenden.
- Wechseln Sie nach ein bis zwei Frames zu Mid Flow 3. Filamente sollten bei etwa 10 sub/s depolymerisieren (Referenz12).
- Um das Experiment zurückzusetzen, brechen Sie alle fluoreszierend markierten Filamente, indem Sie sie kontinuierlich mit maximaler Leistung für ~ 2 min dem Laser aussetzen. Um verschiedene Bedingungen zu testen, wechseln Sie die Lösungen 1, 2 oder 3 und injizieren Sie sie (High Flow, 3-4 min). Wiederholen Sie die Schritte 8.7-8.9.
9. Andere Einzelfilament-Experimente
- Testen der Wechselwirkungen von ABPs mit F-Actin
HINWEIS: Die Mikrofluidik wurde erfolgreich eingesetzt, um die Aktivität mehrerer seitenbindender ABPs wie Cofilin, Tropomyosin und Arp2/3 zu quantifizieren. Gemäß dem Protokoll in Abschnitt 8:- Ändern Sie Kanal 3 in das fluoreszierende ABP, das im F-Puffer von Interesse ist. Einspritzen (High Flow 3, 3 min).
- Filamentpolymerisation: Stellen Sie die Druckeinstellung für 10 min auf Mid Flow 1 ein.
- ABP-Bindung: Starten Sie die Erfassung mit TIRF. Passen Sie die Bildrate in Abhängigkeit von der ABP-Konzentration an. Wechseln Sie nach 1-2 Frames zu Mid Flow 3.
HINWEIS: Je nach ABP kann es auch möglich sein, schnell (z.B. für weniger als 5 s) auf Mid Flow 2 umzusteigen, um die Hintergrundfluoreszenz bei der Aufnahme eines Bildes weiter zu reduzieren. - ABP unverbindlich: Während Sie die Akquisition fortsetzen, wechseln Sie zu Mid Flow 2.
- Polymerisation mit Formin am freien Widerhaken
HINWEIS: Es wurde gezeigt, dass Formine die Polymerisation von Filament-Stacheln beeinflussen. Die Mikrofluidik ist besonders geeignet, um Forminbindungs- und Unbindungsraten und deren Einfluss auf die Filamentdehnung zu messen.- Bereiten Sie die folgenden Lösungen vor:
Kanal 1: 10 nM Formin im F-Puffer (Tabelle 1).
Kanal 2: 1 μM 10% Alexa-488 markiertes G-Actin, 4 μM Profilin.
Kanal 3: F-Puffer. - Wechseln Sie die Rohre 1, 2 und 3 (Abschnitt 7). Injizieren Sie mit der Voreinstellung High Flow All für 3-4 min.
- Filamentpolymerisation einleiten: Stellen Sie die Druckeinstellung für 2 min auf Mid Flow 2 ein.
- Forminbindung an Filament-Widerhakende: Stellen Sie die Druckeinstellungen für 30 s auf Mid Flow 1 ein.
- Formin-vermittelte Polymerisation: Stellen Sie die Druckeinstellung auf Mid Flow 2 ein. Mit Formin mDia1 an ihrem Stachelende sollten Filamente bei etwa 50 sub/s13,14,15 polymerisieren.
- Bereiten Sie die folgenden Lösungen vor:
- Polymerisation/Depolymerisation aus oberflächenverankertem Formin
HINWEIS: Es hat sich gezeigt, dass die Polymerisations- und Depolymerisationsraten von forminverzierten Stachelköpfen von der Spannung abhängen, die auf das Filament ausgeübt wird. In der Mikrofluidik erzeugt die Reibung der Fluidströmung entlang der Filamentseite eine Spannung, die proportional zur Filamentlänge und zur Durchflussrate14,16 ist.- Verwenden Sie das in Abschnitt 8 beschriebene Verfahren, indem Sie die Schritte 8.1, 8.2 und 8.3 für die Oberflächenpassivierung ersetzen durch:
- Wechseln Sie Röhrchen 3 auf 1 μg/ml Anti-His-Antikörper im F-Puffer. Für 2 min mit High Flow 3 injizieren.
- Wechseln Sie Rohr 3 mit 5% BSA im F-Puffer. Inject für 5 min bei High Flow 3, gefolgt von 5 min bei Mid Flow 3. Reduzieren Sie während dieses zweiten Schritts den Druck in den Kanälen 1 und 2 auf 7-8 mbar, um einen Gegenstrom von ~-100 nL / min zu erhalten, so dass die gesamten Kammeroberflächen BSA-passiviert sind.
- Wechseln Sie Röhre 3 auf 100 nM His-tagged formin in F-buffer. Für 5 min mit High Flow 3 injizieren. Wechseln Sie Rohr 3 mit F-Puffer. 5 Minuten lang mit High Flow 3 injizieren, um sicherzustellen, dass keine Formine im Schlauch verbleiben.
- Bereiten Sie die folgenden Lösungen vor und injizieren Sie sie (je 200-300 μL, in F-Puffer):
Kanal 1: 1 μM 10% Alexa-488 markiert G-Actin.
Kanal 2: 1 μM unmarkiertes G-Aktin, 4 μM Profilin.
Kanal 3: Nur F-Puffer. - Filamentkeimbildung: Setzen Sie die oberflächenverankerten Formine G-Aktin aus (Einstellung Mid Flow 1).
- Filamentpolymerisation: Setzen Sie die Kammer mit Mid Flow 2 Profilin-Aktin aus.
- Erfassung starten: 1 Frame/2 s, Epifluoreszenz. Bei Formin mDia1 sollten Filamente bei 50-80 sub/s polymerisieren, abhängig von der Filamentlänge und der Durchflussrate14.
- Filament-Depolymerisation: Beginn der Erfassung (1 Rahmen/4 s, Epifluoreszenz). Nach 1-2 Frames legen Sie die Filamente dem F-Puffer, Mid Flow 3, aus. Bei Formin mDia1 sollten Filamente bei 5-15 sub/s depolymerisieren, abhängig von der Filamentlänge und dem Durchfluss14.
- Verwenden Sie das in Abschnitt 8 beschriebene Verfahren, indem Sie die Schritte 8.1, 8.2 und 8.3 für die Oberflächenpassivierung ersetzen durch:
- Aktinfilamente mit unbeschrifteten Segmenten
HINWEIS: Die Aktinfluoreszenzmarkierung erzeugt mehrere Artefakte, wie z. B. Pausen während der Depolymerisation17 und veränderte Tropomyosinbindung18. Eine Problemumgehung für diese Artefakte besteht darin, Mikrofluidik zu verwenden, um Filamente mit unbeschrifteten Segmenten zusammenzustellen.- Bereiten Sie die folgenden Lösungen vor und injizieren Sie sie (200-300 μL im F-Puffer):
Kanal 1: 1 μM unmarkiertes G-Aktin, 1 μM Profilin.
Kanal 2: 0,3 μM 10% Alexa-488 markiert G-Actin. - Setzen Sie die Oberfläche nacheinander Kanal 2 (5 min), Kanal 1 (10 min) und Kanal 2 (15 min) aus, um ADP-Aktin-unmarkierte Segmente mit fluoreszierend markierten Segmenten an jedem Ende zu erzeugen.
- Bereiten Sie die folgenden Lösungen vor und injizieren Sie sie (200-300 μL im F-Puffer):
- Geriffelte endverankerte Filamente mit Gelsolin
HINWEIS: Bei Spektrin-Aktin-Samen polymerisieren Filamente an ihrem freien Widerhakenende, während das spitze Ende durch den Spektrin-Aktin-Samen stabilisiert wird. Eine Alternative besteht darin, Filamente mit einem mit Widerhaken versehenen Endverschließer wie Gelsolin zu verankern.- Bereiten Sie eine F-Aktinlösung von 4 μM 10% Alexa-488 markiertem G-Aktin in 20 μL F-Puffer vor. Lassen Sie das Aktin spontan bei RT für mindestens 30 min auf der Bank nukleieren und polymerisieren. Wickeln Sie das Rohr in Aluminiumfolie, um es vor Licht zu schützen.
- In der Zwischenzeit wird die mikrofluidische Kammer vorbereitet und die Oberfläche mit einer Mischung aus 5% BSA und 1% Biotin-BSA passiviert (siehe Schritt 8.2).
- Spülkanal 3 mit F-Puffer (2 min bei High Flow 3). Injizieren Sie 10 μg/ml Neutravidin in F-Puffer (4 min bei High Flow 3).
- Wechseln Sie die Röhren zu:
Kanal 1: 10 nM Biotin-Gelsolin (Tabelle 1).
Kanal 2: F-Puffer.
Kanal 3: 0,4 μM vorpolymerisiertes F-Aktin. - Injizieren Sie alle Lösungen zusammen mit der Einstellung High Flow All für 3 min.
- Setzen Sie die gesamte Kammer Gelsolin aus (Mid Flow 1, 30 s).
- Befestigen Sie Filamente an der Oberfläche (Low Flow 3: Kanal 3 bei 3 mbar, Kanäle 1 und 2 bei ~2 mbar, für ca. 2 min).
- [OPTIONAL] Wenn die Filamentdichte zu niedrig ist, wiederholen Sie die Schritte 9.5.6 und 9.5.7.
- Depolymerisation am spitzen Ende: Beginn der Erfassung (1 Rahmen/30 s, Epifluoreszenz). Nach 1-2 Frames stellen Sie Filamente nur dem Puffer aus, Mid Flow 2. Filamente sollten bei etwa 0,2 sub/s depolymerisieren.
10. Fascin-induzierte Filamentbündelbildung und -demontage durch ADF/Cofilin
HINWEIS: Um Aktinfilamentbündel zu bilden, achten Sie auf eine ausreichend hohe Filamentsamendichte an der Oberfläche der Kammer. Bei Exposition gegenüber Fascin-Protein werden benachbarte Filamente, die seitlich schwanken, dynamisch durch Fascin-Proteine vernetzt. Da sich Fascin schnell von der Filamentseite19 löst, muss Faszin ständig in der Hauptflusslösung vorhanden sein, um die Filamentbündelung aufrechtzuerhalten.
- Führen Sie die Schritte 8.1-8.3 aus.
- Bereiten Sie die folgenden Lösungen vor (200-300 μL im F-Puffer):
Kanal 1, Polymerisationslösung: 1 μM 10% Alexa-488 markiertes G-Aktin, 1 μM Profilin.
Kanal 2, Bündelungslösung: 200 nM Faszin (Tabelle 1), 0,15 μM 10% Alexa-488 markiertes G-Aktin.
Kanal 3, Demontagelösung: 200 nM ADF/Cofilin (Tabelle 1), 100 nM Fascin, 0,15 μM 10% Alexa-488 markiertes G-Aktin. - Wechseln Sie die Rohre 1 bis 3 (Abschnitt 7). Injizieren Sie mit der Voreinstellung High Flow All für 3-4 min.
- Filamentpolymerisation: Stellen Sie die Druckeinstellung für ~ 10 min auf Mid Flow 1 ein. Die Polymerisation kann mit TIRF abgebildet werden.
- Filamentbündelung (Abbildung 4C): Starten Sie die Bilderfassung (1 Bild/5 s, Epifluoreszenz). Stellen Sie nach 1-2 Frames die Druckeinstellung auf Mid Flow 2 und beobachten Sie die Filamentbündelung.
- Bundle-Fragmentierung: Starten Sie die Bildaufnahme (1 Frame/5 s, Epifluoreszenz). Stellen Sie nach 1-2 Frames die Druckeinstellung auf Mid Flow 3 ein und beobachten Sie die Cofilin-induzierte Demontage von einzelnen Filamenten und Bündeln.
11. Reinigungsverfahren für mikrofluidische Geräte
HINWEIS: Um eine Kontamination von einem Experiment zum anderen zu vermeiden, ist es wichtig, alle Schläuche und Durchflussmesser nach jedem Experiment umfassend zu reinigen und vollständig zu trocknen.
- Trennen Sie alle Schläuche von der PDMS-Kammer und entsorgen Sie die Kammer.
- Um PEEK-Schläuche und Durchflussmesser zu reinigen, kleben Sie die Schlauchenden in ein leeres 15-ml-Kunststoffrohr und injizieren Sie die folgenden Lösungen bei maximalem Druck, bis das Reservoir fast leer ist:
400 μL F-Puffer.
400 μL von 0,5 m NaOH.
400 μL reines Wasser.
200 μL Isopropanol. - Durch einen leeren Behälter ersetzen und Luft blasen, bis die Schläuche vollständig trocken sind (~ 2-4 min, maximaler Druck).
12. Bildanalyse
HINWEIS: Während sich dieses Manuskript auf die Methode konzentriert, einzelne Aktinfilamente in der Mikrofluidik zusammenzusetzen, zu manipulieren und zu visualisieren, wird hier eine kurze Methode zur Analyse erworbener Filme bereitgestellt. Die Analyse wird auf 16-Bit-Bildern mithilfe von ImageJ gemäß Abschnitt 8 durchgeführt.
- Die Bildbehandlung ist minimal:
- Importieren Sie den Polymerisations- oder Depolymerisationsbildstapel.
- [OPTIONAL] Homogenisieren Sie die Bildintensität mit der Funktion Hintergrund subtrahieren (Standardeinstellungen (z. B. 'Rollende Kugelradius' = 50 Pixel)). Dies ist besonders nützlich, wenn sich die Hintergrundfluoreszenz im Laufe eines Films ändert oder wenn die Fluoreszenzbeleuchtung über dem Sichtfeld nicht homogen ist.
- Helligkeit und Kontrast anpassen (Hintergrund nahe Null, Filamente nahe Maximum).
- Filament-Kymograph erstellen:
- Wählen Sie ein Filament aus, das nicht pausiert, bricht oder sich löst. Wählen Sie nicht aufgrund des Verhaltens aus. Zeichnen Sie eine Linie 1-2 Pixel darüber (Geradeaus-Werkzeug ). Speichern Sie die Filamentnummer (Hinzufügen im ROI-Manager).
- Wenden Sie die Funktion Reslice an (Slice-Anzahl: 5 Pixel). Berechnen Sie die maximale Intensität (Funktion Zprojection).
- Messen Sie die Polymerisations-/Depolymerisationsrate:
- Zeichnen Sie auf dem Kymographen eine Linie entlang des Filament-Stachelkopfendes (Werkzeug "Gerade Linie ", Abbildung 4A). Messen Sie die Linienbreite und -höhe (Funktion Messen).
- Wiederholen Sie die Schritte 12.2-12.3 über mehrere Filamente. Berechnen Sie die Polymerisations-/Depolymerisationsraten (Abbildung 4A):
 , wobei v die Rate (in sub/s), w die Linienbreite (Pixel), pix die Pixelgröße (nm), h die Zeilenhöhe (Frames) und dt die Zeit zwischen den Frames (in Sekunde) ist. Dabei entspricht 2,7 nm dem effektiven Beitrag einer Aktin-Untereinheit zur Filamentlänge.
, wobei v die Rate (in sub/s), w die Linienbreite (Pixel), pix die Pixelgröße (nm), h die Zeilenhöhe (Frames) und dt die Zeit zwischen den Frames (in Sekunde) ist. Dabei entspricht 2,7 nm dem effektiven Beitrag einer Aktin-Untereinheit zur Filamentlänge.
Ergebnisse
Für alle oben beschriebenen Experimente sollten fluoreszierend markierte Aktinfilamente deutlich sichtbar sein, mit gutem Kontrast, was auf eine geringe Hintergrundfluoreszenz von der Oberfläche hinweist (Abbildung 4, siehe Ergänzende Datei 1 zur Fehlerbehebung bei häufigen Problemen). Aktinfilamente sollten auch nicht an der Oberfläche haften: Wenn die dominante Durchflussrate niedrig ist, sollten die seitlichen Schwankungen der Aktinfilamente bei der Beobachtung bei lebendiger Betrachtung wahrnehmbar sein und es ermöglichen, klar festzustellen, dass sie nur an einem ihrer Enden verankert sind. In ähnlicher Weise sollten bei der Verwendung von TIRF-Bildgebung ihre vertikalen Schwankungen durch Intensitätsänderungen entlang ihrer Länge und Zeit sichtbar sein. Abhängig von den angewendeten Durchflussraten muss möglicherweise die TIRF-Eindringtiefe angepasst werden, um die Bildqualität der von TIRF erfassten Aktinfilamente zu optimieren.
Wenn Filamente Polymerisationsbedingungen ausgesetzt werden (siehe Abschnitt 8), sollte die Filamentdehnung regelmäßig erfolgen (d. h. die Dehnung am Ende des Filaments wird nicht durch Oberflächenwechselwirkung oder dauerhaftes Verkleben behindert). Darüber hinaus sollte die gemessene Dehnungsrate des Filament-Stacheldrahtendes dem erwarteten Wert entsprechend der Aktinkonzentration im Rohr 1,20 entsprechen, was darauf hinweist, dass die Röhrchenlösung korrekt in die mikrofluidische Kammer geflossen ist (Abbildung 4A). Ebenso sollten Filamente, wenn sie einer Pufferlösung ausgesetzt werden, stetig mit einer Geschwindigkeit depolymerisieren, die ihren ADP-Gehaltwiderspiegelt 4 (Abbildung 4A). Wenn bereits gewachsene Aktinfilamente einer Lösung aus fluoreszierend markiertem Cofilin ausgesetzt werden, werden Cofilincluster nukleiert und wachsen sowohl zum spitzen als auch zum Stachelende hin (Abbildung 4B) mit einer Rate, die von der Cofilinkonzentration abhängt. Bei der Beurteilung einer potenziellen Vernetzungsaktivität eines ABP, wie z. B. Faszin, (Abbildung 4C), werden nahegelegene Aktinfilamente, die Bündel bilden, leicht durch ihre höhere Fluoreszenzintensität und eine Änderung ihrer lateralen Schwankungen erkannt.
Der Flüssigkeitsstrom übt eine viskose Reibungskraft auf Aktinfilamente aus, die an der Oberfläche der mikrofluidischen Kammer verankert sind. Der Reibkraftkoeffizient auf F-Aktin beträgt η = 6,10-4 pN·s/μm2, ausgedrückt pro Filament-Mikrometerlänge14. Bei mittleren Durchflussraten, wenn die Filamenthöhe um einen konstanten Durchschnitt von 250 nm über der Oberfläche schwankt, besteht ein Kraftgradient vom frei schwebenden Ende bis zum Filamentverankerungspunkt. Man kann also die angelegte Spannung an jedem Punkt entlang des Filaments berechnen, wobei F = 6ηπLv verwendet wird, wobei v die lokale Strömungsgeschwindigkeit 250 nm über der Oberfläche ist (Abbildung 1B) und L die nachgeschaltete Filamentsegmentlänge ist (d. h. vom betrachteten Punkt bis zum freien Ende). Bei höheren Durchflussraten ist die durchschnittliche Höhe des Filaments nicht konstant, sondern steigt linear vom Verankerungspunkt bis zum freien Ende an, bleibt im Durchschnitt unter250 nm und variiert je nach Durchflussraten, was zu einem komplexeren Zugkraftprofil entlang des Filaments 21 führt.

Abbildung 4: Repräsentative Ergebnisse. Typische Experimente, bei denen Aktinfilamente aus Spektrin-Aktin-Samen polymerisiert und verschiedenen ABPs ausgesetzt werden. Der Übersichtlichkeit halber wird nur ein Bruchteil des Sichtfeldes angezeigt. (A) Ergebnis des grundlegenden Polymerisations-Depolymerisationsexperiments (Abschnitt 8). Filamente werden mit einer Lösung von 0,8 μM 10% Alexa-488 markiertem G-Aktin polymerisiert, für 15 min gealtert, um alle Untereinheiten in ADP-Aktin umzuwandeln (nicht gezeigt), und depolymerisiert, wenn sie nur F-Puffer ausgesetzt werden. Unten: Kymographen, die zur Quantifizierung der Polymerisations- und Depolymerisationsraten verwendet werden. Erfasst bei 1 Bild/5 s, 200 ms Belichtungszeit, 150 mW 488 nm Laser bei 9% Leistung, TIRF (Lasereindringtiefe 250 nm). (B) Fragmentierung von Einzelaktinfilamenten um 500 nM mKirsch-Cofilin-1. Aktin ist mit ATP-ATTO48822 (gelb) markiert und Cofilin-1 ist mit mCherry (blau) verschmolzen. Oben: Bruchteil eines Sichtfeldes. Hinweis: Proteinaggregate auf der Oberfläche. Unten: Kymograph, der die Bindung von Cofilin-1 an ein Filament zeigt (Pfeilspitzen zeigen Cofilin-1-Domänenkeimbildungsereignisse), was zu einem Fragmentierungsereignis führt (Blitzsymbol). Aufgenommen bei 1 Bild/4 s, 200 ms Belichtung, 150 mW 488 nm Laser bei 16% und 100 mW 561 nm Laser bei 12% Leistung, Epifluoreszenz. (C) Bündelung von Aktinfilamenten durch Faszin (Abschnitt 10.5). Filamente wurden zunächst mit 0,8 μM 5% Alexa-488 markiertem G-Aktin polymerisiert und mit 200 nM Fascin gebündelt. Im Vergleich zu einzelnen Filamenten erscheinen Filamentbündel zwei- bis dreimal heller und nicht perfekt auf den Fluss ausgerichtet. Aufgenommen bei 1 Rahmen/10 s, 200 ms Exposition, 20% 200 W Quecksilberlampenintensität, Epifluoreszenz. (A-C) Der Hintergrund wurde mit der Ad-hoc-Funktion von ImageJ subtrahiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
| Proteinname | Spezies | Uniprot ref (Sequenz) | Original-Reinigungsprotokoll Ref. | Kommentare |
| Aktin | Kaninchen | P68135 (volle Länge) | 23 | Zur Fluoreszenzmarkierung siehe Referenz 24 |
| profilin1 | Mensch | P07737 (volle Länge) | 25 | Siehe auch Ref 11 |
| Spectrin-Aktin-Samen | Mensch | N/A | 26, 27 | Siehe auch Ref 11 |
| Cofilin1 | Maus | P18760 (volle Länge) | 28 | |
| Gelsolin | Mensch | P06396 (volle Länge) | 29 | |
| mDia1 formin | Maus | O08808 (AA 552–1255) | 13 | Detaillierteres Protokoll in Ref 24 |
| Fascin1 | Mensch | Q16658 (volle Länge) | 30 |
Tabelle 1: Aktin und Aktin-bindende Proteine 23,24,25,26,27,28,29,30
| Reagenz | Konzentration |
| Tris-HCl pH 7,4 | 5 mM |
| Kcl | 50 mM |
| MgCl2 | 1 mM |
| EGTA | 0,2 mM |
| Atp | 0,2 mM |
| DVB-T | 10 mM |
| DABCO | 1 mM |
Tabelle 2: Zusammensetzung des F-Puffers DABCO und eine relativ hohe Konzentration von DVB-T werden verwendet, um photoinduzierte Schäden an Filamenten aufgrund von Lichteinwirkung während Fluoreszenzmikroskopie-Experimenten zu begrenzen.
| Festlegen von Namen | Druck (mBar) | Durchfluss (nL/min) |
| Max. Druck | 300 | ~ 30 000 (im dominanten Kanal) |
| Hochdruck | 150 | ~ 15 000 (im dominanten Kanal) |
| Mittlerer Druck | 12 | ~ 1500 (im dominanten Kanal) |
| "Veränderungsdruck" | 12 für alle Einlässe, 5 für Steckdose | ~ 500 (in jedem Einlass) |
Tabelle 3: Übereinstimmung zwischen angelegten Drücken und gemessenen Durchflussraten. Die resultierenden Durchflussraten hängen stark vom Versuchsaufbau ab. Die Werte werden für eine mikrofluidische Kammer mit einem 1 cm langen Hauptkanal mit einem Querschnitt von 20 μm x 800 μm (Höhe x Breite) angegeben, der mit jedem Reservoir mit einem 80 cm langen PEEK-Schlauch verbunden ist.
Ergänzungsdatei 1: Klassische Probleme, Ursachen und Lösungen. Sie traten häufig auf Probleme bei der Arbeit mit Mikrofluidik und / oder Einzelaktinfilamenten auf. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Diskussion
Im Vergleich zu Standard-Single-Filament-Verfahren, bei denen Aktinfilamente entlang ihrer Länge durch mehrere Punkte an der Oberfläche verankert oder durch ein Crowding-Mittel wie Methylcellulose nahe an der Oberfläche gehalten werden, bietet die Mikrofluidik eine Reihe von Vorteilen. Da die Wechselwirkungen mit der Oberfläche minimal sind, werden die künstlichen Pausen, die diese Wechselwirkungen sowohl während der Dehnung als auch der Depolymerisation auslösen können, vermieden. Die Filamente werden durch die Strömung parallel zueinander ausgerichtet, was ihre Überwachung und die Messung ihrer Längen erleichtert. Die Lösung um die Filamente wird ständig erneuert und setzt sie konstanten Proteinkonzentrationen aus. Die Möglichkeit, schnell (<1 s, Abbildung 3D,E) zwischen verschiedenen Proteinlösungen zu wechseln, denen die Filamente ausgesetzt sind, ermöglicht die Durchführung zeitgesteuerter sequenzieller Experimente, die oft für kinetische Studien von entscheidender Bedeutung sind. Schließlich kann der viskose Widerstand, der von der fließenden Lösung auf die Filamente ausgeübt wird, ausgenutzt werden, um eine kontrollierte mechanische Spannung auf die Filamente auszuüben (Abschnitt "Repräsentative Ergebnisse"). Man sollte beachten, dass moderate Flüssigkeitsströme (Mid Flow Druckeinstellungen) Filamente nahe genug an die Oberfläche bringen (~ 250 nm), um sie effizient mit TIRF abzubilden und gleichzeitig eine minimale Spannung (<1 pN) zu erzeugen14.
Im Vergleich zu klassischen Single-Filament-Assays erfordert die Mikrofluidik jedoch größere Mengen an Proteinlösungen: typischerweise einige 100 μL, wenn ein Standardexperiment mit weniger als 10 μL durchgeführt werden könnte. Dies kann eine Einschränkung bei der Verwendung wertvoller Proteine sein. Klassische Experimente können verwendet werden, um die relevanten experimentellen Bedingungen (z. B. absolute oder relative Konzentrationen verschiedener Proteine) zu ermitteln, bevor eine Reihe von mikrofluidischen Experimenten gestartet wird. Eine weitere Einschränkung, wie bei allen anderen Einzelfilament-Techniken in vitro, ergibt sich aus der unvollkommenen Passivierung der Deckglasoberfläche. Die Reproduzierbarkeit bei der Deckglasreinigung und Bindung der Passivierungsschicht (BSA, PEGylierung, etc.) ist immer schwer zu kontrollieren. Eine einstufige Passivierungstechnik, die auf der PEG-Silan-Oberflächenbehandlung basiert, ist in vielen Labors zur Technik der Wahlgeworden 7,15. Daher kann die effektive Dichte von Filamentsamen zwischen den Experimenten um etwa das Doppelte variieren, selbst wenn sie so genau wie möglich wiederholt wird. Man sollte einen zufriedenstellenden Bereich der Filamentoberflächendichte anstreben und bereit sein, das Experiment bei Bedarf zu wiederholen. Häufig auftretende Probleme bei der Arbeit mit Mikrofluidik und/oder Einzelaktinfilamenten werden in der Ergänzungsdatei 1 behandelt.
Für das hier vorgestellte Basisprotokoll ist zu beachten, dass Spektrin-Aktin-Samen, die als kurz stabilisierte Filamente angesehen werden können, zufällig orientiert sind, da sie an der Oberfläche haften. Wenn sich die aus diesen Samen gezüchteten Filamente mit dem Fluss ausrichten, wird ihr Teil, der dem Samen am nächsten ist, stark gebogen, jeder mit seinem eigenen Winkel. Die Länge, über die Filamente gebogen werden, ist in der Regel sehr klein, wenn Filamente mittleren oder hohen Strömungen ausgesetzt sind. Tatsächlich ist diese Länge im Allgemeinen kleiner als die Beugungsgrenze (~ 200 nm) und kann daher nicht leicht erkannt werden. Wichtig ist, dass die ABPs, die empfindlich auf Filamentkrümmung reagieren, in dieser stark gebogenen Region unterschiedlich binden und funktionieren. Um verzerrte Ergebnisse zu vermeiden, ist es am einfachsten, diesen Bereich aus der Analyseauszuschließen 21.
Bevor wir mit der Mikrofluidik begannen, um einzelne Aktinfilamente zu manipulieren und zu visualisieren, wurde sie bereits verwendet, um einzelne DNA-Filamente3 zu untersuchen, die weitaus flexibler sind. Dies kann zu bemerkenswerten Unterschieden führen, da der Fluss die DNA dramatisch abwickeln und ihre scheinbare Länge dramatisch verändern kann. Mikrofluidik kann auch, sehr ähnlich wie die hier vorgestellte Methode, verwendet werden, um Mikrotubuli zu untersuchen; Diese sind viel steifer, können aber dennoch an der Strömung ausgerichtet werden, um ihre Dehnung und Depolymerisation zu messen, wobei der schnelle Wechsel der Bedingungen31,32 genutzt wird, oder durch eine senkrechte Strömung gebogen werden, um die Plastizität des Mikrotubuli zu messen7.
Wir haben hier das Protokoll für das Grundlagenexperiment vorgestellt, bei dem Filamente nur an einem Ende verankert sind und bei dem die Strömungsrichtung im Sichtfeld während des gesamten Experiments gleich ist. Diese beiden Eigenschaften können variiert werden. Beispielsweise können Filamente durch mehrere Punkte verankert werden, um ein anderes Kraftprofil entlang des Filaments zu erzeugen. Ebenso kann die Strömungsrichtung (in der Nähe der Verbindung zwischen den Eintrittskanälen, in der Strömungskammer) variiert werden, um Filamente lokal zu biegen, da der nicht verankerte Teil in eine andere Richtung zeigt als das verankerte Filamentsegment21. Filamente, die sich aus zufällig verankerten Spektrin-Aktin-Samen verlängern, können auch vernetzenden Proteinen ausgesetzt werden, um Bündelzu bilden 33 (siehe Abschnitt 10). Durch die Kombination von Mikrofluidik mit anderen Techniken (Mikrostrukturierung, optische Pinzette usw.) oder durch das Entwerfen von mikrofluidischen Kammern mit Kompartimenten zur Änderung von Strömungslinien können mehrere Konfigurationen erstellt werden, um die spezifische ABP-Aktivität auf einzelnen Filamenten zu untersuchen oder kleine Aktinnetzwerkezu bilden 34. Die Anzahl der Kombinationen, zusammen mit dem Vorteil und der Vielseitigkeit der Mikrofluidik, bietet Forschern viele Werkzeuge, um die räumlich-zeitliche Regulation von Aktinnetzwerken auf molekularer Ebene zu entschlüsseln.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Wir danken den B. Ladoux und R.-M. Mège Labor für die Verwendung ihrer UV-Reiniger Ausrüstung, und J. Heuvingh und 0. du Roure für die erste Schulung, die wir über die Vorbereitung von Formen auf Siliziumwafern und die Bereitstellung von Tipps zur Mikrofluidik erhalten haben. Wir danken für die Förderung durch den Europäischen Forschungsratszuschuss StG-679116 (an A.J.) und die Agence Nationale de la Recherche Grants Muscactin und Conformin (an G.R.-L.).
Materialien
| Name | Company | Catalog Number | Comments |
| β-Casein | Merck | C6905 | Used at 8 mg/mL |
| Biopsy punch (with plunger) | Ted Pella | 15115-2 | ID 0.75 mm, OD 1.07 mm |
| Biotin-BSA | Merck | A8549 | Used at 1 mg/mL |
| BSA | Merck | A8022 | Used at 50 mg/mL |
| Coverslip Mini-Rack Teflon holder | Invitrogen | C14784 | for 8 coverslips |
| Coverslips 22x40mm Thickness #1.5 | Menzel Gläser | 631-1370 | |
| DABCO | Merck | D27802 | component in f-buffer |
| DTT | Euromedex | EU0006-D | component in f-buffer |
| Ester NHS Alexa Fluor 488 | Invitrogen | A20000 | Fluorophore for actin labeling on Lys328. |
| EZ-Link Sulfo-NHS-Biotin | Thermo Scientific | 21338 | To biotinylate actin on Lys328 |
| Hellmanex III | Hellma | 9-307-011-4-507 | Glass cleaning detergent |
| ImageJ | NIH | N/A | open source software |
| Laboport | KNF | 811kn.18 | vacuum pump (ultimate vacuum: 240 mbar) |
| Magic invisible tape | Scotch | 7100024666 | standard transparent office tape |
| Micrewtube | Simport | T341-6T | 2 mL microfluidic reservoir tubes |
| Microfluidic device Part 1: Flow Unit S | Fluigent | FLU-S-D-PCKB | Flowmeter |
| Microfluidic device Part 2: Fluiwell-4C-2 mL | Fluigent | 14002001PCK | Reservoir holder |
| Microfluidic device Part 3: MFCS-EZ | Fluigent | EZ-11000001 EZ-00345001 | Pressure controller |
| Model 42 - UVO-Cleaner | Jelight Inc. | 42-220 | Ultraviolet cleaner |
| N6-(6-Aminohexyl)-ATP-ATTO-488 | Jena Bioscience | NU-805-488 | ATP-ATTO used to label actin |
| neutravidin | Thermo Scientific | 31000 | |
| PLL-PEG | SuSoS | PLL(20)-g[3.5]- PEG(2) | Use at 1 mg/mL in PBS. |
| Polydimethylsiloxane (PDMS) Sylgard 184 Silicon Elastomer | Dow Corning | 1673921 | Contains PDMS base and curing agent |
| Polyetheretherketone (PEEK) tubing | Merck | Z226661 | “Blue” : I.D. = 0.25 mm |
| Safety blow gun | Coilhose Pneumatics | 700-S | filtered air |
| Silicon tubing | VWR | 228-0701P | connect PEEK to coupler |
| Stainless steel catheter coupler | Prime Bioscience | SC22/15 | Inserted into PDMS inlets and outlet to connect to PEEK tubing |
| Thermoplastic film | Sigma Aldrich | PM996 | Standard "parafilm" |
| Ultrapure ethanol | VWR | 64-17-5 | |
| Ultrasonic cleaning bath | VWR | USC200TH | To accomodate 1 L beakers |
| Vacuum dessicator | SP Bel-Art | F42022-0000 | to degas the PDMS or solutions |
Referenzen
- Wioland, H., Jégou, A., Romet-Lemonne, G. Celebrating 20 years of live single-actin-filament studies with five golden rules. Proceedings of the National Academy of Sciences of the United States of America. 119 (3), 2109506119 (2022).
- Kuhn, J. R., Pollard, T. D. Real-time measurements of actin filament polymerization by total internal reflection fluorescence microscopy. Biophysical Journal. 88 (2), 1387-1402 (2005).
- Brewer, L. R., Bianco, P. R. Laminar flow cells for single-molecule studies of DNA-protein interactions. Nature Methods. 5 (6), 517-525 (2008).
- Jégou, A., et al. Individual actin filaments in a microfluidic flow reveal the mechanism of ATP hydrolysis and give insight into the properties of profilin. PLoS Biology. 9 (9), 1001161 (2011).
- Gicquel, Y., et al. Microfluidic chips for in situ crystal x-ray diffraction and in situ dynamic light scattering for serial crystallography. Journal of Visualized Experiments: JoVE. (134), e57133 (2018).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. Journal of Visualized Experiments: JoVE. (86), e50549 (2014).
- Schaedel, L., et al. Microtubules self-repair in response to mechanical stress. Nature Materials. 14 (11), 1156-1163 (2015).
- Zimmermann, D., Morganthaler, A. N., Kovar, D. R., Suarez, C. In vitro biochemical characterization of cytokinesis actin-binding proteins. Methods in Molecular Biology. 1369, 151-179 (2016).
- Funk, J., et al. Profilin and formin constitute a pacemaker system for robust actin filament growth. eLife. 8, 50963 (2019).
- Pandit, N. G., et al. Force and phosphate release from Arp2/3 complex promote dissociation of actin filament branches. Proceedings of the National Academy of Sciences of the United States of America. 117 (24), 13519-13528 (2020).
- Wioland, H., et al. ADF/Cofilin accelerates actin dynamics by severing filaments and promoting their depolymerization at both ends. Current Biology: CB. 27 (13), 1956-1967 (2017).
- Pollard, T. D., Mooseker, M. S. Direct measurement of actin polymerization rate constants by electron microscopy of actin filaments nucleated by isolated microvillus cores. The Journal of Cell Biology. 88 (3), 654-659 (1981).
- Kovar, D. R., Harris, E. S., Mahaffy, R., Higgs, H. N., Pollard, T. D. Control of the assembly of ATP- and ADP-actin by formins and profilin. Cell. 124 (2), 423-435 (2006).
- Jégou, A., Carlier, M. -. F., Romet-Lemonne, G. Formin mDia1 senses and generates mechanical forces on actin filaments. Nature Communications. 4, 1883 (2013).
- Breitsprecher, D., et al. Rocket launcher mechanism of collaborative actin assembly defined by single-molecule imaging. Science. 336 (6085), 1164-1168 (2012).
- Courtemanche, N., Lee, J. Y., Pollard, T. D., Greene, E. C. Tension modulates actin filament polymerization mediated by formin and profilin. Proceedings of the National Academy of Sciences of the United States of America. 110 (24), 9752-9757 (2013).
- Niedermayer, T., et al. Intermittent depolymerization of actin filaments is caused by photo-induced dimerization of actin protomers. Proceedings of the National Academy of Sciences. 109 (27), 10769-10774 (2012).
- Gateva, G., et al. Tropomyosin isoforms specify functionally distinct actin filament populations in vitro. Current Biology: CB. 27 (5), 705-713 (2017).
- Aratyn, Y. S., Schaus, T. E., Taylor, E. W., Borisy, G. G. Intrinsic dynamic behavior of fascin in filopodia. Molecular Biology of the Cell. 18 (10), 3928-3940 (2007).
- Pollard, T. D. Rate constants for the reactions of ATP- and ADP-actin with the ends of actin filaments. The Journal of Cell Biology. 103, 2747-2754 (1986).
- Wioland, H., Jegou, A., Romet-Lemonne, G. Torsional stress generated by ADF/cofilin on cross-linked actin filaments boosts their severing. Proceedings of the National Academy of Sciences of the United States of America. 116 (7), 2595-2602 (2019).
- Colombo, J., et al. A functional family of fluorescent nucleotide analogues to investigate actin dynamics and energetics. Nature Communications. 12 (1), 548 (2021).
- Spudich, J. A., Watt, S. The regulation of rabbit skeletal muscle contraction. I. Biochemical studies of the interaction of the tropomyosin-troponin complex with actin and the proteolytic fragments of myosin. The Journal of Biological Chemistry. 246 (15), 4866-4871 (1971).
- Romet-Lemonne, G., Guichard, B., Jégou, A. Using microfluidics single filament assay to study formin control of actin assembly. Methods in Molecular Biology. 1805, 75-92 (2018).
- Gieselmann, R., Kwiatkowski, D. J., Janmey, P. A., Witke, W. Distinct biochemical characteristics of the two human profilin isoforms. European Journal of Biochemistry. 229 (3), 621-628 (1995).
- Lin, D. C., Lin, S. Actin polymerization induced by a motility-related high-affinity cytochalasin binding complex from human erythrocyte membrane. Proceedings of the National Academy of Sciences of the United States of America. 76 (5), 2345-2349 (1979).
- Casella, J. F., Maack, D. J., Lin, S. Purification and initial characterization of a protein from skeletal muscle that caps the barbed ends of actin filaments. The Journal of Biological Chemistry. 261 (23), 10915-10921 (1986).
- Kremneva, E., et al. Cofilin-2 controls actin filament length in muscle sarcomeres. Developmental Cell. 31 (2), 215-226 (2014).
- Le Clainche, C., Carlier, M. -. F. Actin-based motility assay. Current Protocols in Cell Biology. , 1-20 (2004).
- Vignjevic, D., et al. Formation of filopodia-like bundles in vitro from a dendritic network. The Journal of Cell Biology. 160 (6), 951-962 (2003).
- Duellberg, C., Cade, N. I., Holmes, D., Surrey, T. The size of the EB cap determines instantaneous microtubule stability. eLife. 5, 13470 (2016).
- Duellberg, C., Cade, N. I., Surrey, T. Microtubule aging probed by microfluidics-assisted tubulin washout. Molecular Biology of the Cell. 27 (22), 3563-3573 (2016).
- Suzuki, E. L., et al. Geometrical constraints greatly hinder formin mDia1 activity. Nano Letters. 20 (1), 22-32 (2020).
- Wioland, H., Suzuki, E., Cao, L., Romet-Lemonne, G., Jegou, A. The advantages of microfluidics to study actin biochemistry and biomechanics. Journal of Muscle Research and Cell Motility. 41 (1), 175-188 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten